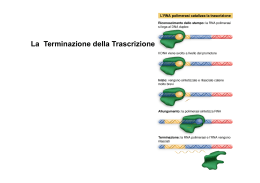
Cap 11 Trascrizione e regolazione negli eucarioti Allison 11 Introduzione e 11.1 1. Cromatina 2. RNA Polimerasi EUCARIOTI in generale • RNA polimerasi I (Pol I) Trascrive gli rRNA ed è localizzata principalmente nel nucleolo • RNA polimerasi II (Pol II) Trascrive i precursori degli mRNA, i miRNA, ed alcuni snRNA • RNA polimerasi III (Pol III) Trascrive i geni che codificano per i tRNA, gli rRNA 5S, lsnRNA U6 (splicing) e l’RNA 7SL (compl SRP – traduzione) PIANTE: • RNA polimerasi IVa (Pol IVa) (controllo silenziamento genico) • RNA polimerasi IVb (Pol IVb) (controllo silenziamento genico) L’isolatore: gli isolatori funzionano come marcatori ai confini della cromatina ed hanno attività di blocco degli enhancer 11.6 - trans attivatori (Amaldi pag 308-310) • TRANS • CIS • Trans-attivatori – Attivatori trascrizionali (Es proteina Gal4 su sito GAL1) Fattori di trascrizione 11.7 – Domini legami di DNA (pagine 311-319) • • • • • Elica-giro-elica (helix-turn-helix) Omeodominio (Homeobox) Dominio a dito di zinco (Zinc finger) Dominio a cerniera di leucina (leucin zipper) Dominio elica-ansa-elica (helix-loop-helix) • controllo combinatoriale Reclutamento complesso trascrizione
Scaricare