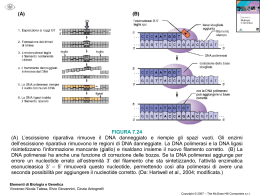
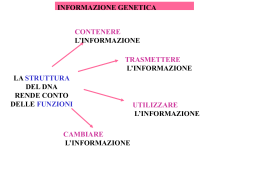
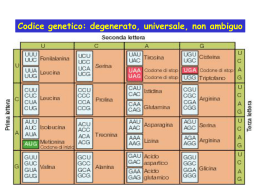
Fisiologia del controllo glicidico In condizioni fisiologiche il livello di glucosio ematico (glicemia) è finemente controllato, oscillando tra 80 e 90 mg/dl a digiuno, per aumentare fino (e non oltre) i 120 140 mg/dl nella prima ora dopo il pasto, e ritornare quindi ai valori basali entro due ore dal termine dell’assorbimento dei carboidrati. Fisiologia del controllo glicidico Lo scopo del mantenimento dell’omeostasi glucidica è quello di fornire al tessuto nervoso, in condizioni di mancato apporto alimentare, la quantità di glucosio sufficiente per la sua vitalità. Il tessuto nervoso è infatti strettamente glucosiodipendente Fisiologia del controllo glicidico Effetti dell’Insulina sul metabolismo Insulina : ‘ormone È dell’abbondanza’ Facilita il trasporto del glucosio nelle cellule dei tessuti ‘insulino-dipendenti (++muscolo scheletrico a riposo e tessuto adiposo) Uptake di glucosio Promuove la glicogenosintesi nel fegato e nel muscolo scheletrico Fisiologia del controllo glicidico Condizioni ‘stressanti Condizioni ‘normali’ (digiuno prolungato, eserciz fisico intenso….) + InsulinaGlucagone CortisoloAdrenalina GH Fisiologia del controllo glicidico (Condizioni ‘basali’) • Dopo una notte di digiuno il 50% del glucosio circolante è utilizzato dal cervello, il resto dalle cellule ematiche,dal rene e dal muscolo • L’origine di tale glucosio è epatica, attraverso la glicogenolisi (75%) e la gluconeogenesi (25%). • Le riserve di glicogeno epatico utilizzabili sono circa 70100 grammi, insufficienti quindi ad assicurare la normoglicemia per un periodo superiore alle 24 ore , da cui la necessità di ripristinare le riserve di glicogeno con l’introduzione di cibo. Fisiologia del controllo glicidico A digiuno 50% Fisiologia del controllo glicidico Dopo il pasto 25% 5560% 1520% Diabete Il Diabete è un disordine cronico del metabolismo caratterizzato da elevati livelli di glucosio plasmatici a digiuno (Iperglicemia) , da diuresi abnorme (poliuria) con presenza di glucosio nelle urine (glicosuria) E’ conseguente alla carenza o al mancato utilizzo dell’insulina Diabete primario • Diabete di tipo I o ‘ Insulinodipendente ’(IDDM), o ‘ diabete giovanile ’ • Diabete di tipo II o ‘ non insulinodipendente ’ (NIDDM), o ‘ diabete dell’età matura ’ Obeso (90%) Non obeso (10%) Diabete Tipo II • Esordio tardivo (generalmente dopo i 50aa) • Esordio subdolo (frequente la diagnosi casuale in paziente asintomatico) casuale in paziente asintomatico) Regolazione del metabolismo glicidico durante esercizio Mentre in condizioni di riposo il muscolo utilizza soprattutto acidi grassi liberi provenienti dal tessuto adiposo (NEFA), nella condizione di esercizio moderato passa ad utilizzare sia NEFA che glucosio intramuscolare (depositi di glicogeno) ed extramuscolare (di origine epatica). All’inizio il glucosio deriva soprattutto dai depositi di glicogeno dei muscoli attivi, che gradualmente si riducono (la concentrazione di glicogeno muscolare è di 120 mmol/Kg nell’adulto, 70mmol/Kg nel bambino). Durante esercizio prolungato, il glucosio di origine epatica non è più sufficiente a soddisfare le richieste periferiche; intervengono allora i NEFA, derivanti dalla lipolisi, che vengono a costituire il maggior substrato energetico. Regolazione del metabolismo glicidico durante esercizio Durante esercizio è necessario il rilascio di una maggiore quantità di glucosio in circolo per soddisfare le aumentate richieste metaboliche. Gli ormoni che interagiscono nell’aumentare il glucosio plasmatico sono Glucagone Adrenalina Cortisolo GH Regolazione del metabolismo glicidico durante esercizio Insulina (effetto inibitorio delle catecolamine sulle beta cellule) Cortisolo Glucagone GH Adrenalina Tess Adiposo Glicogenolisi Lipolisi Glicogenolisi Gluconeogenesi Ossidazione NEFA (lattato, NEFA, aa) NEFA rilascio di Fegato MUSCOLO DUPLICAZIONE La replicazione è semiconservativa Le DNA polimerasi, utilizzando nucleotidi trifosfati (ATP, GTP, CTP, TTP), catalizzano la formazione di un legame esterico tra il gruppo 3’OH del dessosiriboso dell’ultimo nt. al fosfato in 5’ del nt. da aggiungere. L’energia di legame è data dalla liberazione di 2 fosfati dal nucletotide trifosfato utilizzato. DNA Pol n(NUC) + GP˜P˜P n(NUC)GMP + PP REPLICAZIONE Le catene di DNA vengono sintetizzate copiando i filamenti di stampo con la legge della complementarietà. La sintesi richiede una FORCELLA di REPLICAZIONE Necessita dell’elicasi che continui ad aprire l’elica Necessita di un innesco di RNA costruito dalla primasi (RNA polimerasi) 5’ 3’ La sintesi del nuovo filamento ad opera della DNA polimerasi avviene solo in direzione 5’→3’ poiché i 2 filamenti sono antiparalleli la direzione di un filamento (guida) è continua e la direzione dell’altro filamento (tardivo) è discontinua (frammenti di OKAZAKI) La Replicazione necessita di un apparato enzimatico complesso: DNA Polimerasi Proteine iniziatrici Primasi e Elicasi; Proteine che stabilizzano il DNASS e lo proteggono dalla degradazione; Proteine che stimolano le DNA polimerasi. Topoisomerasi levano i superavvolgimenti Ligasi PROCARIOTI E.C.: oriC 245 pb (dnaA) + ATP si lega al 9 mer e separa i filamenti a livello dei13 mer elicasi:(dnaB)+ATP e dnaC 1020 subunità A/T Primasi + elicasi: primosoma terminazione nella zona ter: incontro delle 2 forcelle di replicazione le 2 eliche si separano ad opera della topoisomerasi II sul filamento con progressione discontinua 3’ 5’ T G G T 5’ 3’ DNA polimerasi III: forma un legame esterico 34 cal./mole (2Pi) STRUTTURA core: α : sintesi; θ : ?; ε : attività 3’→5’ esonucleasica e correzzione di bozze; τ : tiene insieme i 2 core. β : (dimero) pinza si lega al primer e al DNA stampo, fa da morsa legandosi al core scorrendo lungo il filamento rendendo al massimo la processività della polimerasi γ : (5 subunità: δ , δ ’, γ , χ , ϕ ) caricatore della pinza utilizza ATP Replisoma: insieme delle 2 molecole di DNA polimerasi III che proseguono insieme SSB stabilizzano i singoli filamenti e li proteggono dalla degradazione Elicasi apre la doppia elica scorrendo lungo il DNA a singolo filamento e richiede la presenza di ATPi Primasi sintetizza i primer DNA polimerasi I: azione esonucleasica 5’→3’ sull’RNA, rimuove RNA e sintetizza il DNA tra un frammento e l’altro in direzione sempre 5’→3’. I frammenti di Okazaki contengono 10002000 nucleotidi Ligasi: saldatura in presenza di E e AMP, unisce l’estremità 3’ dell’ultimo nucleotide aggiunto da DNA polimerasi I con il 1° messo precedentemente dalla DNA polimerasi III. toposisomerasi II + ATP taglia i 2 filamenti topoisomerasi I taglia 1 filamento, Correzione di bozze delle DNA polimerasi III DNA totale 4x106 pb; Replicazione 40 min. (5001000 nt. al sec.) Fedeltà 1 errore ogni 105106 nt durante la replicazione Corr. Di Bozze: 1 errore ogni 107108 nt. 9 Riparazione postreplicativa:1 errore ogni 10 nt. EUCARIOTI (ssb) (β ) Elicasi si muove sul filamento guida RPA: proteine con la stessa funzione di SSB in procarioti Primasi sintetiza i primers RFC caricatore della pinza; PCNA conferisce un’alta processività alla Pol δ ; stessa funzione di β di DNA Pol III (pinza) RNAsiH e Fen1 tolgono i primer Polimerasi α , β , γ .δ , ε . (≅ 12) α (4 subunità), insieme a primasi e a RFC polimerizza il 1° tratto del filamento a sintesi discontinua; RFC spiazza α e quindi associa PCNA a δ δ (+ ε ?) (412 subunità) sintetizza il filamento giuda e allunga i frammenti di Okazaki; sintesi dei frammenti di DNA al posto dei primers; ha attività esonucleasica 3’→5 e quindi correzione di bozze. β e ε : attività di riparo γ : DNA mitocondriale Telomerasi contiene uno stampo di RNA che sintetizza molte copie di sequenze telomeriche: è una trascriptasi inversa che scivola lungo la sequenza aggiunta aggiungendone altre. Molto attive durante la vita embrionale, le ellule somatiche non hnno attività telomerasica e quindi perdono i telomeri ad ogni replicazione (50100 nt) e hanno quindi un numero di divisioni limitato. E’ stato postulata una relazione inversa tra telomeri e età Telomeri: sequenze ripetute in tandem nei ciliati 5’TTGGG3’, nell’uomo: 5’TTAGGG3’ 10000 ripetizioni Origini: in lievito sino a 400; Replicone 1 origine (50000300000 pb); repliconi vicini tendono ad aprirsi contemporaneamente. Replicazione asincrona, ma programmabile e prevedibile; dipende dall’organizzazione della cromatina (etero e eucromatina) CICLO CELLULARE FASE S = REPLICAZIONE 200300 pb libere di istoni durante la replicazione frammenti di Okazaki 100200 nt (DNA avvolto sul nucleosoma) Velocità: 50 nt/s (ingombro degli istoni) DNA totale 5x109 pb DNA Polimerazazzione Esonuclesi Esonucleasi Polimerasi 5’ 3’ 5’ 3’ 3’ PROCARIOTI Attività 5’ I + + + rimuove primer/primer DNA II + + III + + polimerizza f. guida e tardivo/corr. di bozze riparo EUCARIOTI α + inizia la catena di DNA sul f. ritardo/riparo + β γ riparo + + polimerizza DNA mitocondriale δ + imerizza il DNA/primer DNA/corr.di bozze + pol + ε associata a δ /riparo + FEN1 rimuove primer Telomerasi (RNA) + telomerico sintesi di DNA Trascrizione CAPACITA’ DI ESPRIMERSI DNA RNA PROTEINE (nt) trascrizione (nt) traduzione (aa) Trascrizione Traduzione Procarioti citoplasma citoplasma Eucarioti nucleo citoplasma TRASCRIZIONE *1) Gli RNA vengono sintetizzati copiando un solo filamento di DNA detto: stampo o codificante 5’TTAGCGAT3’ f. di senso 3’AATCGCTA5’f. codificante ↓ 5’UUAGCGAU3’ RNA 2) l’allungamento avviene in direzione è 5’→3’ 3) vengono utilizzati nucleotidi trifosfati (UTP al posto di TTP) *4) le RNA polimerasi sono capaci di iniziare; nell’RNA il 1° nucleotide è trifosfato * differenze tra replicazione e trascrizione la trascrizione in eucarioti è seguita dalla maturazione del trascritto primario che porta al RNA RNA: 1 solo filamento; riboso; adenina, guanina, citosina, uracile. Tipi Funzione % Stabilità P E mRNA informazione specifica: proteina 2.5 36m mh tRNA trasferimento specifica: aminoacido 1020 + + rRNA officina (rRNA+proteine ribosomiali) 8090 ++ ++ snRNA maturazione mRNA in eucarioti GENI Struttura Pro Eu mRNA 1 (per ogni proteina) secondaria tRNA (aa: 20) 4070 100300 secondaria complessa (basi rare) rRNA 515 (rRNA) 1001000 (rRNA) secondaria complessa (rRNA+proteine ribosomiali) ansa PROCARIOTI 1 RNA polimerasi: mRNA, rRNA, tRNA basi appaiate EUCARIOTI RNA polimerasi I: rRNA (28S, 18S, 5.8S) RNA polimerasi II: mRNA, snRNA RNA polimerasi III: tRNA, rRNA (5S), snRNA (U6, 7S) la trascrizione in eucarioti è seguita dalla maturazione del trascritto primario (es.premRNA mRNA) TRASCRIZIONE in PROCARIOTI 1 polimerasi composta da 4 subunità α α β β ’(core)+σ =oloenzima INIZIO il core: lega debolmente al DNA e può esplorare il DNA, ma non riconoscere l’inizio di un gene σ : si lega efficientemente a regioni specifiche al promotore, induce il DNA ad aprirsi (bolla di trascrizione ≅ 17 pb);il core viene strettamente legato al promotore e cosi posizionato per iniziare la trascrizione dal 1° nucleotide del gene. σ σ ALLUNGAMENTO σ si allontana dopo la trascrizione di 89 bp: inizia la polimerizzazione in direzione 5’→3’ad opera della subunità β ; RNA pol scorre lungo il DNAss, aprendo a valle una bolla di trascrizione (β ’), dove si forma un ibrido RNA/DNA,quando la RNA pol avanza,l’RNA si stacca e il DNA si riavvolge ad α elica. Molte RNA polimerasi trascrivono lo stesso gene in successione (Ibrido DNA/RNA) TERMINAZIONE presenza di sequenze di terminazione (palindromiche), trascritte lentamente e formano strutture a stelo: CCCAGCCCGGCAUAACAUCCGGGCUUUUUU. DNA Pol fa una pausa sulla sequenza UUUUU 3’e quindi si stacca. A A U C A A C U GC GC CG CG GC 5’ CCC AU UUUUU 3’ In presenza della proteina ρ le sequenze di terminazione sono meno complesse TRASCRIZIONE in EUCARIOTI 120 3 Polimerasi (L e β :7080%identità) GC Promotore basale per mRNA 100 GC 80 30 CAAT Box TATA Box +1 site Initiation of transcription TATA: posiziona la RNAPII al punto di inizio CAAT: regola l’andamento della trascrizione CAAT: GC: aiuta RNAPII a legarsi al punto di inizio, lega SP1 GC: TyrSerProTyrSerProSe (2652) Promotore nudo no TATA box: 1C/+1A INIZIO RNAPII trascrive mRNA e 4 snRNA Necessita di fattori di trascrizione generali Fosforila CTD 11 fattori Lega DNA e TFIID Provoca una distorsione di 80°C Elicasi + ATP TERMINAZIONE ALLUNGAMENTO Aggiunta del cappuccio Poliadenilazione MODIFICAZIONI POSTTRASCRIZIONALI Il trascritto primario (PremRNA) viene maturato in mRNA Capuccio Poliadenilazione Rimozione introni Capuccio Metil transferasi aggunge CH3 al 7’ di G; anche i primi 2 nt sono metilati Funzioni: protezione al 5’; esportazione; traduzione Aggiunta di guanina al 1° nt trascritto dopo la trascrizione di 2030 nt.; legame 5’5’ trifosfato Struttura del cappuccio Poliadenilazione 4050 nt 50 nt PremRNA+RNA nucleari= hnRNA; hnRNA+proteine=hnRNP Proteine (34 a 120 kD) con motivi di legame per RNA (RNP). Prevengono la formazione di strutture secondarie, interessate in tagli: PoliA, Splicing e trasporto dell’mRNA 12 A 12A Funzioni di PoliA (200250 A): Protezione dalla degradazione Efficienza della traduzione PoliA non presente negli mRNA per istoni Rimozione degli introni (splicing) esoni: DNA, trascritti e tradotti (N: 10) SPLICEOSOMA: Introni: trascritti e rimossi (N1: 9) 100 proteine snRNP (snRNA:197210 nt, 6/10 proteine:RNP) hnRNP (hnRNA, proteine) RNA elicasi Proteine SR A U A C Laccio viene degradato I 2 esoni adiacenti vengono legati Promotori geni rRNA e tRNA Trascrizione di rRNA +10 +60 +47 +96 hUBF TFIIIA TFIIIC SL1 (TBP) TFIIIB (TFIIB,TBP) Trascrizione di 1 unità di rRNA nel nucleolo Modificazioni posttrascrizionali e assemblaggio ribosomi RNAPI Modificazioni posttrascrizionali tRNA 3’CCA trascritto in E.C. Trasporto dell’mRNA dal nucleo al citoplasma Complesso di carico: Proteina cargo, Ran+GTP, Esportina1, Procarioti trascrizione e traduzione avvengono contemporaneamente nel citoplasma NLS Eucarioti NES Ran+GDP Ran+GTP TRADUZIONE CODICE GENETICO Dobbiamo effettuare un passaggio da un codice a 4 lettere (nucleotidi) ad un codice a 20 lettere (amminoacidi) DNA CAGT 4 lettere RNA CAGU 4 lettere Proteine ARNDC etc 20 lettere Come si passa dai nucleotidi agli amminoacidi? • Qual è la codificazione? lunghezza dell’unità di • Qual è la sequenza di basi delle parole di codice relative a ciascuno degli aa codificati? • Quali sono le modalità di riconoscimento tra le parole dei due linguaggi? No. nucleotidi/a.a. Possibilità 1 41=4 2 42=16 3 43=64 Minimo codice proponibile I tre ruoli dell’RNA nella sintesi proteica • L’RNA messaggero (mRNA) porta l’informazione copiata dal DNA sotto forma di una serie di “parole” di tre basi dette CODON • L’RNA transfer (tRNA) decifra il codice, mediante uno specifico ANTICODON, al quale è associato un particolare amminoacido • L’RNA ribosomale (rRNA) si associa con una serie di proteine per formare i ribosomi, le fabbriche che sintetizzano le proteine La struttura dei ribosomi nei procarioti e negli eucarioti La struttura tridimensionale dell’RNA evidenzia la sua funzione decodificante Il ruolo dell’RNA nella sintesi proteica Poiché il codice genetico utilizza 61 codon diversi per specificare gli amminoacidi, ci si dovrebbe aspettare che nella sintesi proteica siano coinvolti 61 diversi tRNA, alcuni con lo stesso amminoacido. Molti tRNA riconoscono più di un codon. Quindi il numero dei tRNA è molto inferiore a 61. Ipotesi del Vacillamento: flessibilità di appaiamento tra la terza base del codon e la corrispondente base dell’anticodon Appaiamento non standard (tentennamento) L’inizio della sintesi proteica nei batteri avviene vicino alla sequenza di Shine-Delgarno nell’mRNA Negli eucarioti l’inizio della sintesi proteica generalmente avviene, generalmente, all’estremità 5’ dell’mRNA Visione d’insieme della traduzione Fasi Della Traduzione: inizio • Attacco delle subunità ribosomali e del primo a.a. portato dal tRNA iniziatore all’mRNA • Posizionamento sul primo codon da tradurre PROCARIOTI Subunità 40S/60S Fattori d’inizio tRNA iniziatore Fonte di Energia EUCARIOTI 30S/ 50S Sì formilmetionina GTP Sì metionina ATP/GTP Il processo di inizio negli eucarioti coinvolge un gruppo diverso di fattori (chiamati eIF) e un particolare tRNA iniziatore tRNAMet, che porta una metionina non formilata Fasi Della Traduzione: allungamento Allungamento della catena polipeptidica mediante attacco di aminoacil-tRNA e sintesi del legame peptidico. PROCARIOTI Subunità Fattori di Allungamento tRNA Fonte di energia 30S/50S EUCARIOTI 40S/60S Sì Sì tutti tutti GTP GTP Fasi Della Traduzione: terminazione • Avviene quando il ribosoma arriva ad un codone di stop (UAA, UAG, UGA) che si colloca nel sito A • Rilascio della catena polipeptidica e rilascio delle due subunità ribosomali PROCARIOTI EUCARIOTI Subunità 30S/50S 40S/60S Fattori di terminazione 3 1 GTP GTP Fonte di energia La traduzione simultanea da parte di ribosomi multipli e il loro rapido ricambio aumenta l’efficienza della sintesi proteica Eventi post-traduzionali Specifici segnali, contenuti nella sequenza amminoacidica delle proteine, le dirigono alle loro destinazioni cellulari finali sintesi proteica inizia sui ribosomi liberi nel citoplasma. proteine destinate a nucleo e mitocondri sono completate e hanno segnali che permettono il legame e l’entrata gli organelli a cui sono destinate. proteine destinate a: RE, Golgi, lisosomi e all’esterno lla cellula completano la loro sintesi su ribosomi attaccat a superficie del RE. rano, quindi nel RE grazie all’interazione di una sequenza gnale idrofobica con un canale della membrana. Le modificazioni covalenti delle proteine dopo la traduzione includono: proteolisi, glicosilazione e fosforilazione.
Scarica