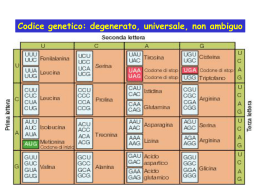
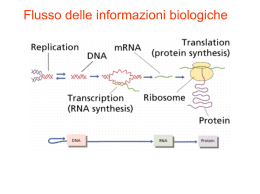
La stabilità del complesso di trascrizione, e quindi del numero di copie di RNA sintetizzate dalla RNA polimerasi II, dipende dai fattori di trascrizione (proteine) che compongono il complesso mRNA di Eucarioti e Procarioti mRNA maturo (dopo splicing ed eventuale editing) che può essere esportato nel citoplasma per essere tradotto in proteina Il genoma dei batteri è organizzato in operon Un operon è una unità trascrizionale indipendente, formata da (2-15) geni regolati da un solo promotore I geni di un operon sono diversi, ma concorrono allo sviluppo di una medesima funzione Negli Eucarioti sono presenti molti diversi RNA codificati da geni di dimensioni molto differenti: - codificanti: mRNA - non-codificanti: rRNA tRNA snRNA snoRNA tiRNA RNA circolari miRNA gRNA piRNA SRP-RNA RNA della telomerasi Xist RNA lncRNA (long non coding RNA) NUCLEOLO La trascrizione dei geni in vivo può chiamare in causa contemporaneamente più RNA polimerasi gene 4 gene 1 DNA gene 2 RNA RNA polimerasi gene 3 L’assemblaggio del ribosoma eucariotico è uno spettacolare esempio di processo altamente dinamico e regolato. Le subunità ribosomali minore (40S) e maggiore (60S) sono il risultato dell’assemblaggio di circa 80 proteine ribosomali e quattro diversi rRNA. La costruzione e la localizzazione intracellulare delle particelle pre-ribosomali coinvolgono una moltitudine di fattori transitoriamente associati ed enzimi che consumano energia in oltre 50 step, fattori di trasporto (come Bud20) che viaggiano con le particelle ribosomali pre-60S per raggiungere il citoplasma dove vengono rilasciati prima dell’ inizio della traduzione. Targeted proteomics reveals compositional dynamics of 60S pre-ribosomes after nuclear export- Mol Syst Biol. 2012;8:628. doi: 10.1038/msb.2012.63 - Altvater et al. Oltre 200 fattori non-ribosomali coinvolti in assemblaggio, maturazione e trasporto intracellulare delle particelle pre-40S e pre-60S mentre viaggiano dal nucleolo al citoplasma I pre-rRNA prodotti dalla Pol I subiscono modificazioni co-trascrizionali nel nucleolo dove avviene l’assemblaggio con le proteine ribosomali. Le particelle pre-60S incontrano ~ 100 fattori mentre viaggiano verso il complesso del poro nucleare (NPC) seguitando a subire variazioni della composizione; al contrario, le particelle pre-40S subiscono un minor numero di modificazioni mentre viaggiano verso gli NPC Targeted proteomics reveals compositional dynamics of 60S pre-ribosomes after nuclear export- Mol Syst Biol. 2012;8:628. doi: 10.1038/msb.2012.63 - Altvater et al. Il numero di geni codificanti proteine nei vertebrati non è radicalmente diverso dal numero di geni negli invertebrati (es. circa 20.000 geni nell’uomo contro 19.000 geni in Caenorhabditis elegans). La prevalenza dello splicing alternativo nei vertebrati è importante per loro superiore complessità Lo splicing alternativo è prevalente negli eucarioti multicellulari (nei mammiferi riguarda il 95% dei geni); quelli unicellulari hanno per lo più un solo introne o introni corti e presenti solo in alcuni geni Alternative splicing: a pivotal step between eukaryotic transcription and translation - Nat Rev Mol Cell Biol. 2013 Mar;14(3):153-65. doi: 10.1038/nrm3525 - Kornblihtt et al. - Erratum in Nat Rev Mol Cell Biol. 2013 Mar;14(3). doi:10.1038/nrm3560 Lo splicing occupa una posizione centrale nel flusso delle informazioni genetiche degli eucarioti, dipende da vari fattori di regolazione e dal tipo di cromatina Lo splicing alternativo è almeno altrettanto importante per il differenziamento come la regolazione della trascrizione Nella comunicazione tra cellule, varie molecole segnale influenzano lo splicing alternativo (i) modificando l’attività di chinasi e fosfatasi che controllano trascrizione e traduzione, (ii) la localizzazione subcellulare dei regolatori dello splicing e (iii) l’acetilazione degli istoni che influenzano la struttura della cromatina Alternative splicing: a pivotal step between eukaryotic transcription and translation - Nat Rev Mol Cell Biol. 2013 Mar;14(3):153-65. doi: 10.1038/nrm3525 - Kornblihtt et al. - Erratum in Nat Rev Mol Cell Biol. 2013 Mar;14(3). doi:10.1038/nrm3560 L'organizzazione dei geni in esoni ed introni e l'evoluzione dello splicing alternativo hanno portato due vantaggi, (i) la produzione di proteine che uniscono, in una singola molecola, domini funzionali già testati dalla selezione naturale e (ii) la possibilità per un singolo gene di produrre due o più varianti di mRNA maturo, simili ma non identiche, ampliando notevolmente la capacità codificante dei genomi eucariotici Lo splicing è realizzato dallo spliceosoma assemblato nei siti di splicing di ogni introne. Sulla base della sua sequenza, un sito di splicing può essere forte o debole. La competizione tra siti forti e deboli vicini nel pre-mRNA è un fattore che porta a splicing alternativo. Alternative splicing: a pivotal step between eukaryotic transcription and translation - Nat Rev Mol Cell Biol. 2013 Mar;14(3):153-65. doi: 10.1038/nrm3525 - Kornblihtt et al. - Erratum in Nat Rev Mol Cell Biol. 2013 Mar;14(3) - doi:10.1038/nrm3560 Lo splicing alternativo è governato da sequenze cis regolatorie nel pre-mRNA, ESE (exonic splicing enhancers) ESS (exonic splicing silencers) ISE (intronic splicing enhancers) ISS (intronic splicing silencers) e da due principali famiglie di proteine regolatrici dello splicing alternativo, •SRSF (Ser/Arg-rich splicing factors) •hnRNP (heterogeneous nuclear ribonucleoproteins) Alternative splicing: a pivotal step between eukaryotic transcription and translation - Nat Rev Mol Cell Biol. 2013 Mar;14(3):153-65. doi: 10.1038/nrm3525 - Kornblihtt et al. - Erratum in Nat Rev Mol Cell Biol. 2013 Mar;14(3). doi:10.1038/nrm3560 attggaaaccgaaacccgttggtcacctctgcaatagccctccctccctcacttctacaattttgtgaca gtggtcttgttttctgcattctctgcttcacgtgcttgttttgttggagcgcgtttgcatgctgctttaa attctgaaatattaaaaaaatttcgaagtttttcagcacatgggatgggagttttgaatttcaatttttt aaaaacatttttctgtgattagtgccgtcgtggcacggctgttagccgcctatccggtttattcgatact ttGTGAGTTTTTTGTAACTTTATGGTCGTCGAAATGGGAAAACTTGGCCACCAATATAAGTTTGGAAAAC AATTTCCTAAAAATAAAATAATTGAACTTTTCCGATGAATAAAAAAATCGATCAGATATTCTGGAAAAAA AATCGATAAATTAATCGATTTTCTTGGAAAATACATCGAAAAATTGAGAAAAATAGAAAAATGAATGTTT TTCGATTACCGATTTATTGATTTTTCGTGAAAACTGAGTTCAGATAATTTTAAAAGCAATGTTTTTCATT TTTCAAATCAGAATCACTATAGTTTTGAAAAATCAATAATTAATTTATTGATTTTTCAATATAATTTTTT GGAAAAAATAGAAAAATCCCTTTCTAAAAGTTTTAAATTTCCAAGAAAAATTCATTTTCAAAATCACCAA CGCGCTCTATAGAGTAGTCGATGAAAATCTCCGTTAAGGGTGCATGGGCAAAACGCGCTCGAACGACAAT TGTTATTGTATGTTTGGTCTTGCAACGAAAAGTTTGAAAAATTGAAAAAAAGTTGTGTCTGATACATTTT TTTTTGGCATTTTCTGCTATTTTACACCAGAAAAAATTTAATAAACATAAAAAATCGAAATTTTTCAAGT TGGACAATTTTCAGtgagcatcttatccatcctagttctcagttcaggacttgtgcacattcgtttagag ccagatattcgcaaagccttttcaccggatgattcagatgctggata GTAAGTGACTACTGACCTTGAAG CCTCCTTCCTCCACCAGTCAGAAATAACACGTTTTTTCGCAATGTTTTTCTTTTTCTAATTCGATTTCCC TTTCTCCCTTTCTTATTGTGATTTGGTCAATGTTTGGTTGACTGGGAAGAAAATTGAATTTTTTTGGAAT TCCACTTGAAGTTAAAAAACCCAAAATAAATATTTGATCAAAAATAAATAAGAAAAAAAAGAAAACTTTA AAGCAAATGAAAATTTCGTTCGTAACTATTTTGTTAATTTTTTTAAAACTCCTATTTTAAATATATGCTT TTTGCGGAAATTTCTATAAATTTTTTTACATTTTTC AGtgaaacccgtgtctggctggaatactacggac tcgacatctatccggaacgagcattctgtatttttaccgccaagcgcgaaaattccagtattctccagga aggcgcactggcagacGTAAGTTGATTCTCCGTCACGCCCACTTTTCTGGCGGGAATTTAAAAAATTTC A Gatttatactgtggacaatcgactatcggcggcagttggctaccaagatggggatggacgaaaaaattgc gatccactctgcgacttgaacagcccctttcacttgttagcg GTAGGTGGTGGTCTAGGGTGTCATTTTT CGATTTTTTCAATTATTCGATGTTTTTAGTGAAAATCGAAAAATCTAAAAATTGAAAATCGAAAAATGAA AGAAACATTGTTTTTTGGGGACCAAACATCTTAATGAATTTAACAACAGGGAAAACTGAACAGAAACCTG GACGGTCTTATCCCATTTATCTATATTCTTAAAATGAATGATGGAGAAAAAAGTTAAAATAAAAACATTA TCAGCTTTTTGTAAGTTTTTCTCAAAAATTGTTCGATTTTTCGATTTTCTAAAAAGTCGAAAAACCGAAA CCCTTGGTGGTGGTGGTGGTGGACTAGAAAACTCTTCAACGACCACATGGCAATTTTC AGaatttgacgc ggagaaacaatggtaccacaagtgtattcacctatccggatatgccatatagcggactggatattttcct gggacttcacttgagtaatgcggattttggtaagattttttttgaaatgttaaatgaaaagttgaaaaat agtttttatgatttagccactttccagttaaaatttcatttttttaactataaaaagttctggaaaaatg aatttctAGgccgccgatcctaaaAGTgcaccatttcgcAGaAGTacGTacAGTttcccatctatccctA GTgGTcttGTtttctgcattctctgcttcacGTgcttGTtttGTtggAGcgcGTttgcatgctgctttaa attctgaaatattaaaaaaatttcgaAGTttttcAGcacatgggatgggAGTtttgaatttcaatttttt aaaaacatttttctGTgattAGTgccGTcGTggcacggctGTtAGccgcctatccgGTttattcgatact ttGTGAGTTTTTTGTAACTTTATGGTCGTCGAAATGGGAAAACTTGGCCACCAATATAAGTTTGGAAAAC AATTTCCTAAAAATAAAATAATTGAACTTTTCCGATGAATAAAAAAATCGATCAGATATTCTGGAAAAAA AATCGATAAATTAATCGATTTTCTTGGAAAATACATCGAAAAATTGAGAAAAATAGAAAAATGAATGTTT TTCGATTACCGATTTATTGATTTTTCGTGAAAACTGAGTTCAGATAATTTTAAAAGCAATGTTTTTCATT TTTCAAATCAGAATCACTATAGTTTTGAAAAATCAATAATTAATTTATTGATTTTTCAATATAATTTTTT GGAAAAAATAGAAAAATCCCTTTCTAAAAGTTTTAAATTTCCAAGAAAAATTCATTTTCAAAATCACCAA CGCGCTCTATAGAGTAGTCGATGAAAATCTCCGTTAAGGGTGCATGGGCAAAACGCGCTCGAACGACAAT TGTTATTGTATGTTTGGTCTTGCAACGAAAAGTTTGAAAAATTGAAAAAAAGTTGTGTCTGATACATTTT TTTTTGGCATTTTCTGCTATTTTACACCAGAAAAAATTTAATAAACATAAAAAATCGAAATTTTTCAAGT TGGACAATTTTCAGtgAGcatcttatccatcctAGTtctcAGTtcAGgacttGTgcacattcGTttAGAG ccAGatattcgcaaAGccttttcaccggatgattcAGatgctggatAGTAAGTGACTACTGACCTTGAAG CCTCCTTCCTCCACCAGTCAGAAATAACACGTTTTTTCGCAATGTTTTTCTTTTTCTAATTCGATTTCCC TTTCTCCCTTTCTTATTGTGATTTGGTCAATGTTTGGTTGACTGGGAAGAAAATTGAATTTTTTTGGAAT TCCACTTGAAGTTAAAAAACCCAAAATAAATATTTGATCAAAAATAAATAAGAAAAAAAAGAAAACTTTA AAGCAAATGAAAATTTCGTTCGTAACTATTTTGTTAATTTTTTTAAAACTCCTATTTTAAATATATGCTT TTTGCGGAAATTTCTATAAATTTTTTTACATTTTTC AGTgaaacccGTGTctggctggaatactacggac tcgacatctatccggaacgAGcattctGTatttttaccgccaAGcgcgaaaattccAGTattctccAGga AGgcgcactggcAGacGTAAGTTGATTCTCCGTCACGCCCACTTTTCTGGCGGGAATTTAAAAAATTTC A GatttatactGTggacaatcgactatcggcggcAGTtggctaccaAGatggggatggacgaaaaaattgc gatccactctgcgacttgaacAGcccctttcacttGTtAGcgGTAGGTGGTGGTCTAGGGTGTCATTTTT CGATTTTTTCAATTATTCGATGTTTTTAGTGAAAATCGAAAAATCTAAAAATTGAAAATCGAAAAATGAA AGAAACATTGTTTTTTGGGGACCAAACATCTTAATGAATTTAACAACAGGGAAAACTGAACAGAAACCTG GACGGTCTTATCCCATTTATCTATATTCTTAAAATGAATGATGGAGAAAAAAGTTAAAATAAAAACATTA TCAGCTTTTTGTAAGTTTTTCTCAAAAATTGTTCGATTTTTCGATTTTCTAAAAAGTCGAAAAACCGAAA CCCTTGGTGGTGGTGGTGGTGGACTAGAAAACTCTTCAACGACCACATGGCAATTTTCAGaatttgacgc ggAGaaacaatgGTaccacaAGTGTattcacctatccggatatgccatatAGcggactggatattttcct gggacttcacttgAGTaatgcggattttgGTaAGattttttttgaaatGTtaaatgaaaAGTtgaaaaat AGTttttatgatttAGccactttccAGTtaaaatttcatttttttaactataaaaAGTtctggaaaaatG Tipi diversi di alternative splicing Duplice effetto dell’allungamento trascrizionale sullo splicing alternativo: (a) Durante una fase di allungamento veloce, la RNA pol II favorisce il reclutamento dello spliceosoma al sito forte di splicing al 3’ di un introne a valle di un sito debole di splicing al 3’ dell'introne a monte, il risultato è lo skipping dell’esone. Invece, durante un allungamento lento (destra) viene favorito il reclutamento dello spliceosoma sull’introne a monte, il risultato è lo splicing con inclusione dell'esone; Alternative splicing: a pivotal step between eukaryotic transcription and translation - Nat Rev Mol Cell Biol. 2013 Mar;14(3):153-65. Duplice effetto dell’allungamento trascrizionale sullo splicing alternativo: (b) quando entrambi i siti di splicing al 3’ sono ugualmente forti e l'introne a monte ha un sito di legame (verde) per un fattore di splicing che inibisce l’inclusione dell'esone (fattore negativo NF), l’allungamento veloce della Pol II (a sinistra) favorisce il reclutamento dello spliceosoma su ambedue gli introni, garantendo l’inclusione dell'esone. Al contrario, l’allungamento lento (destra) dà il tempo al fattore di splicing negativo di legarsi prima dello spliceosoma, che quindi si legherà al sito di splicing al 5‘ dell'introne a valle con risultante skipping dell’esone. Alternative splicing: a pivotal step between eukaryotic transcription and translation - Nat Rev Mol Cell Biol. 2013 Mar;14(3):153-65. doi: 10.1038/nrm3525 - Kornblihtt et al. - Erratum in Nat Rev Mol Cell Biol. 2013 Mar;14(3) - doi:10.1038/nrm3560 Nel nucleo, il processo di editing altera la sequenza degli mRNA serve per il trasporto di colesterolo nel sangue serve per l’assorbimento di lipidi nell’intestino LA SINTESI PROTEICA INIZIA NEL CITOSOL Se si completa nel Citosol, le Proteine potranno: - restare nel citosol - essere trasportate nel nucleo - essere traslocate nei mitocondri e nei perossisomi Se si completa nel Reticolo Endoplasmatico Rugoso, le Proteine potranno: - raggiungere la membrana - essere secrete in modo costitutivo o regolato - essere trasportate nei lisosomi Sono noti molti lncRNA, codificati da una parte significativa del genoma, con funzione poco sconosciuta. Quelli con funzione nota regolano la trascrizione attraverso la modulazione della cromatina, altri sono utilizzati nella regolazione post-trascrizionale, organizzazione di complessi proteici, signalling cellula-cellula, regolazione allosterica delle proteine. (a) L’RNA è adatto per interagire in modo specifico; per riconoscere un acido nucleico target basta l’appaiamento di piccole sequenze (6 nucleotidi). Per riconoscere in modo specifico una singola base di un acido nucleico, una proteina ha bisogno di un motivo di almeno 35 aminoacidi (105 coppie di basi della sequenza genomica); (b) un RNA si può piegare in complesse strutture tridimensionali che possono specificatamente legarsi a vari ligandi, comprese piccole molecole e peptidi; RNA in unexpected places: long non-coding RNA functions in diverse cellular contexts - Nat Rev Mol Cell Biol. 2013 Oct 9. doi: 10.1038/nrm3679 - Geisler S, Coller J. (c) L’RNA è adatto per una espressione transiente in quanto la trascrizione (e maturazione) genera RNA completamente funzionali che possono essere degradati rapidamente. Questo fatto questo permette di usare RNA per rapidi impulsi. La formazione di una proteina funzionale è molto più complicata come pure la sua demolizione. RNA in unexpected places: long non-coding RNA functions in diverse cellular contexts - Nat Rev Mol Cell Biol. 2013 Oct 9. doi: 10.1038/nrm3679 - Geisler S, Coller J. (d) l’RNA è malleabile ed è più tollerante alle mutazioni. Anche se alcune mutazioni nei geni codificanti proteine sono silenti, molte sono deleterie, quali le mutazioni nonsenso che generano polipeptidi troncati. L’RNA può invece tollerare mutazioni anche all'interno delle regioni responsabili del riconoscimento del target; (e) gli eventi RNA-dipendenti possono essere ereditari. Ad esempio, molti pseudogeni processati erano una volta RNA trascritti che sono stati integrati nel genoma. Inoltre, la telomerasi utilizza uno stampo di RNA per aggiungere ripetizioni nei telomeri alle estremità dei cromosomi. RNA in unexpected places: long non-coding RNA functions in diverse cellular contexts - Nat Rev Mol Cell Biol. 2013 Oct 9. doi: 10.1038/nrm3679 - Geisler S, Coller J. lncRNAs regolano la trascrizione attraverso vari meccanismi: (a-c) lunghi RNA non codificanti (lncRNA) possono modulare la cromatina sia in modo trascrizione-indipendente (parte a) che in modo trascrizione -dipendente (parti b e c). I lncRNA possono legare uno o più complessi che modificano la cromatina ed indirizzare la loro attività su loci specifici di DNA (parte a). A seconda della natura degli enzimi legati, le modificazioni della cromatina mediate da un lncRNA possono attivare o reprimere l’espressione genica. I complessi che modificano la cromatina legati al dominio C-terminale (CTD) della Pol II possono modificare la cromatina durante la trascrizione del lncRNA (parte b). La trascrizione di lncRNA può anche provocare un rimodellamento della cromatina che può sia favorire che inibire il legame di fattori di regolazione (parte c). A seconda della natura dei fattori che legano durante il rimodellamento, l'espressione genica può risultare attivata o no. RNA in unexpected places: long non-coding RNA functions in diverse cellular contexts - Nat Rev Mol Cell Biol. 2013 Oct 9. doi: 10.1038/nrm3679 - Geisler S, Coller J. (d-g) i lncRNA possono modulare sia il macchinario generale della trascrizione (parti d ed e) che fattori regolatori specifici (parti f e g). I lncRNA possono legare direttamente la Pol II per inibire la trascrizione (parte d). La formazione di strutture triplex lncRNA -DNA può anche inibire l'assemblaggio del complesso di pre-inizio trascrizione (parte e). I lncRNA si possono ripiegare in strutture che mimano siti che legano il DNA (a sinistra) o che inibiscono o aumentano l'attività di specifici fattori di trascrizione (a destra, parte f). I lncRNAs possono anche regolare l'espressione genica legando specifici fattori di trasporto per inibire la localizzazione nucleare di specifici fattori di trascrizione (parte g). RNA in unexpected places: long non-coding RNA functions in diverse cellular contexts - Nat Rev Mol Cell Biol. 2013 Oct 9. doi: 10.1038/nrm3679 - Geisler S, Coller J. Figura: influenza dei lncRNA nel processamento e nella regolazione posttrascrizionale degli mRNA: (a,b) i long non coding RNA (lncRNA) possono modulare il processamento degli mRNA. I pattern di splicing possono essere influenzati da lncRNA che si legano con il pre-mRNA (parte a). Per esempio lo splicing del primo introne del pre-mRNA del gene MYC del neuroblastoma è impedito da un trascritto naturale antisenso. I lncRNA antisenso che legano un mRNA possono dirigerne l’editing, forse attraverso l'associazione del duplex con gli ADAR ( adenosina deaminasi che agisce sull’RNA), enzimi che catalizzano la conversione dell'adenosina in inosina negli RNA a doppio filamento (parte b); (c) possono up-regolare la traduzione attraverso l'associazione con la regione 5' di un mRNA; RNA in unexpected places: long non-coding RNA functions in diverse cellular contexts - Nat Rev Mol Cell Biol. 2013 Oct 9. doi: 10.1038/nrm3679 - Geisler S, Coller J. (d-f) i lncRNA contenenti sequenze Alu si possono legare con gli elementi Alu presenti nella regione 3' non tradotta (UTR) di un mRNA, ed il tratto di RNA a doppia elica che si forma può attivare un percorso di degradazione dell’mRNA molecolarmente simile al decadimento mediato da nonsenso (parte d) . LncRNA possono mascherare i siti di legame per miRNA su un mRNA target per bloccare il silenziamento indotto dal miRNA attraverso la formazione di un RISC (RNA-induced silencing complex) (parte e). LncRNA lineari o circolari possono funzionare come esche per sequestrare miRNA dai loro mRNA target (parte f). RNA in unexpected places: long non-coding RNA functions in diverse cellular contexts - Nat Rev Mol Cell Biol. 2013 Oct 9. doi: 10.1038/nrm3679 - Geisler S, Coller J. (e) come cargo di esosomi che mediano il trasferimento di materiale tra cellule, RNA shuttle esosomali (exRNA) possono agire come molecole di signalling durante la comunicazione cellula-cellula; il cargo degli esosomi include mRNA, miRNA e lncRNA; (f) I lncRNA espressi dalle regioni promotore di geni che codificano per gli anticorpi formano un loop R (ripiegamento) per portare i promotori alla ricombinazione mediante reclutamento di adenosina deaminasi ( AID). RNA in unexpected places: long non-coding RNA functions in diverse cellular contexts - Nat Rev Mol Cell Biol. 2013 Oct 9. doi: 10.1038/nrm3679 - Geisler S, Coller J. Espressione differenziale di long non-coding RNA in tre differenti neoplasie umane Associazione di hotair, lncRNA trascritto nel dominio Hox C, induce invasione/metastasi nel carcinoma mammario, cambia il fenotipo cellulare, lega enzimi che modificano gli istoni Dati di espressione Dati di prognosi Gupta et al. Nature, 464, 1071-1076, 2010 La distruzione di alcuni lncRNAs potrebbe essere importante per produrre nuovi farmaci anticancro HOTAIR PRNCR1 (prostate cancer non-coding RNA 1) MALAT1, HULC Tumori mammari primari e metastasi. Tumori del colon retto e metastasi epatiche. Cancro della prostata Carcinoma epatocellulare Tumore della vescica, del colon, della cervice uterina e del polmone UCA1 regola la progressione del ciclo cellulare nel cancro della vescica Yang C, Li X, Wang Y, Zhao L, Chen W. Long non-coding RNA UCA1 regulated cell cycle distribution via CREB through PI3-K dependent pathway in bladder carcinoma cells. Gene. 2012 Coinvolto nel melanoma umano ANRIL è in qualche modo legato a malattie cardiovascolari, aneurismi intracranici, tumori in genere, diabete di tipo 2, periodontiti, malattia di Alzheimer, endometriosi, fragilità nell’anziano, glaucoma. Promuove la progressione tumorale nel carcinoma epatocellulare Congrains A, Kamide K, Katsuya T, Yasuda O, Oguro R, Yamamoto K, Ohishi M, Rakugi H. CVD-associated non-coding RNA, ANRIL, modulates expression of atherogenic pathways in VSMC. Biochem Biophys Res Commun. 2012 SPRY4-IT1 lncRNA-HEIH (High Expression In HCC) UCA1 urothelial carcinoma associated 1 Formazione dei microRNA Formazione dei microRNA RISC è l’acronimo di : RNA Induced Silencing Complex
Scaricare