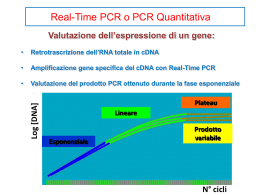
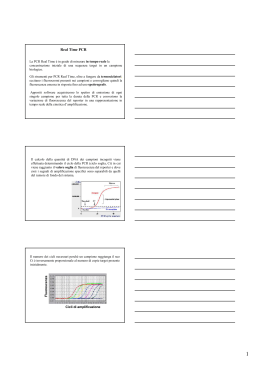
Analisi PCR quantitativa Real time PCR Perché Real-Time? Misura l'amplificazione in tempo reale durante la fase esponenziale della PCR, quando cioè l'efficienza di amplificazione è influenzata minimamente dalle variabili di reazione, permettendo di ottenere risultati molto più accurati e quantitativi Real time PCR Analisi PCR Analisi PCR quantitativa quantitativa impiego di marcatori fluorescenti il cui accumulo segue la stessa cinetica della reazione PCR. La misurazione della fluorescenza dà in tempo reale la quantità dello specifico prodotto di PCR Termociclatore con detector per fluorocromi e software per analisi dati Linea di base (baseline): valore al di sopra del quale inizia l’accumulo di un amplificato Linea soglia (Threshold): scelta dall’operatore in modo da intersecare le curve di tutti i campioni nella fase esponenziale Ciclo soglia: E’ il ciclo della reazione di amplificazione in cui il segnale di fluorescenza del campione è maggiore rispetto a quello della Threshold Linea soglia scelta d a l l ’ o p e r a t o r e In maniera da intersecare le curve di tutti i campioni nella fase esponenziale Indica il valore al di sopra del quale inizia l’accumulo di un amplificato E ’ i l c i c l o d e l l a re a z io n e d i amplificazione in cui il segnale di fluorescenza del campione è maggiore rispetto a q u e l l o d e l l a T h r e s h o l d Real time PCR Sistemi di rilevamento 1. Coloranti che si intercalano nella doppia elica SYBR Green 1 La molecola fluorescente si lega random a tutte le doppie eliche, includendo i dimeri di primers È necessario ottimizzare la metodica per evitare la formazione di prodotti aspecifici All’inizio del processo di amplificazione,Tm la miscela di reazione contiene DNA denaturato, primers e la molecola fluorescente Durante l’allungamento si verifica un aumento di fluorescenza che corrisponde all’ aumento del numero di copie dell’amplicone SYBR Green 1 metodica semplice, non costosa MA aspecifica Analisi della curva di melting 82.6 88.1 melting peak Tm 2. Sonde specifiche marcate con molecole fluorescenti Sonde TaqMan La sonda di tipo TaqMan è un oligonucleotide che, come i primers della PCR, viene disegnato per essere complementare alla sequenza bersaglio da amplificare: è disegnata in modo da ibridarsi all’interno del frammento amplificato nella reazione di PCR Primer 3’ 5’ 3’ R Q 5’ Q 3’ 3’ 5’ 3’Primer 5’ 5’ R 3’ 3’ REPORTER: fluorocromo ad alta energia che emette fluorescenza fluorocromo a bassa energia che spegne la fluorescenza del reporter 5’ 5’ 3’ Si libera 1 molecola di reporter per ogni copia di DNA duplicata 5’ 5’ Se R e Q si trovano vicini, Q spegne l'effetto di R perchè i fotoni di R vengono assorbiti da Q 3’ 3’ 5’ 3’ R 5’ Q 5’ 5’ Probe Forward primer Reverse primer Quantificazione ASSOLUTA i campioni sono quantificati in modo assoluto Necessita di standard di cui si conosce la concentrazione assoluta (utilizzo di una standard curve) Per tutti gli unknowns devono essere saggiate identiche quantità di campioni RELATIVA la quantificazione viene effettuata paragonando i CT Necessita di controlli endogeni (non si utilizza una standard curve) Gli unknowns vengono “quantificati” paragonando il loro con quello del controllo endogeno CT VOGLIO QUANTIFICARE IN TEMPO REALE LA PRESENZA DI UN MICRORGANISMO ALL’INTERNO DI UNA MATRICE 1. Devo avere a disposizione una sonda specie-specifica in grado di amplificare una regione specifica del genoma del microrganismo in esame, di dimensioni comprese tra 100-700 bp verifica in PCR (specificità e curva di melting) 2. Devo preparare concentrazioni cellulari a titolo noto da cui estrarre e dosare il DNA totale 108 cellule 125 ng 106 cellule 10 ng 104 cellule 0.9 pg 3. Devo sottoporre ciascun campione ottenuto a q-PCR per ottenere delle curve di calibrazione, per trovare il range di linearità e la corrispondenza tra: CT : quantità di DNA : quantità di cellule A QUESTO PUNTO POSSO ANALIZZARE LA MATRICE IN ESAME Estraggo il DNA totale dalla matrice ed eseguo una real-time PCR utilizzando tutti i dati ottenuti in fase di messa a punto del metodo: primers specifici curva di calibrazione Quantitativa relativa Plot di amplificazione Control Sample Numero di cicli CT Studio dell’espressione genica 1. Primers specifici per amplificare il gene di interesse 2. Estrazione dell’RNA e Real-time PCR per valutare se il gene viene trascritto L’ RNA estratto deve venire retrotrascritto in DNA =REVERSE TRANSCRIPTASE PCR La RT-PCR amplifica un RNA convertendolo prima in DNA (o DNA omplementare all’RNA) tramite l’enzima trascrittasi inversa, e poi amplificandolo tramite PCR con primers specifici per la sequenza di interesse. Estrarre l’RNA totale dal campione biologico che si vuole esaminare Trasformare il pool di RNA (che conterrà anche l’mRNA trascritto dal gene di interesse) in cDNA tramite la reazione di trascrizione inversa Evidenziare il cDNA di interesse tramite la reazione di PCR Quantitativa relativa: analisi dei dati Normalizzare il gene target con un controllo endogeno (ref) espresso costitutivamente (CT) in una condizione di controllo Variare le condizioni di crescita del ceppo in esame ed eseguire l’analisi. Il controllo endogeno non dovrebbe variare. La variazione di espressione del gene in studio va valutata in riferimento all’espressione dello stesso gene nelle condizioni di controllo Comparare ciascun CT così ottenuto con il CT del controllo ratio = ( Etarget)ΔCt target (control-treated) (Eref)ΔCt ref (control-treated) E = efficienza della reazione: in teoria ad ogni ciclo di amplificazione si ha raddoppio delle copie di DNA Il valore così ottenuto permette di determinare la concentrazione relativa del target
Scaricare