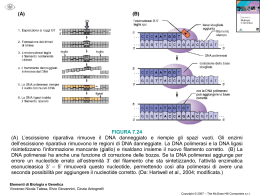
Retrotrascrizione l’mRNA viene convertito in cDNA per mezzo dell’enzima trascrittasi inversa (DNA polimerasi RNAdipendenti ricavate dai virus della mieloblastosi aviaria AMV o della leucemia murina di moloney MuLV) in presenza di nucleotidi e di un singolo primer. Il primer utilizzato può essere specifico per il gene che si intende amplificare oppure aspecifico (oligo (dT) oppure random esameri). 1 Il risultato della reazione di retrotrascrizione produce degli ibridi RNA-DNA. Per clonare le molecole di cDNA, le catene di RNA devono essere distrutte e sostituite da DNA. Un metodo per far ciò consiste nell’uso di RNasi H, che taglia l’RNA negli ibridi; oppure L’RNA viene allontanato dal DNA per semplice denaturazione ottenuta con riscaldamento. Il cDNA fa poi da stampo nella successiva reazione di riamplificazione (PCR classica) in cui si utilizzano primers gene specifici (oppure un primer specifico ed un primer aspecifico). 2 PCR DNA polimerasi: enzima capace di produrre una catena di DNA complementare ad una catena a singolo filamento. Necessita di uno stampo e di un innesco con un 3’-OH libero. La PCR (reazione a catena della polimerasi) consente l’amplificazione di un frammento di DNA specifico a partire da una minima quantità di DNA stampo. La reazione richiede: uno stampo di DNA, una miscela di nucleotidi e due inneschi (primers) che definiscono i punti di inizio della sintesi del DNA. Se si parte da DNA a doppio filamento è necessario separare le catene perché i primers possano poi legarsi alla zona a loro complementare. La PCR, quindi, consiste nel ripetersi di cicli in cui si fa variare la temperatura: a) denaturazione (94-95°C) rottura dei legami idrogeno e separazione dei filamenti b) appaiamento (T specifica) i primers legano la catena nella zona a loro complementare c) estensione (T ottimale per la polimerasi in uso) produzione dei frammenti Alla fine di x cicli di PCR, il prodotto viene controllato: tramite gel di agarosio oppure tramite sequenziamento ed utilizzato. 3 Scelta dei primers: la scelta dei primers viene effettuata seguendo alcuni criteri: a) devono avere una lunghezza compresa tra 17 e 30 nucleotidi b) la percentuale di GC ideale è del 50%; se il contenuto di GC è più basso sii devono usare primers lunghi per evitare Tm troppo basse c) evitare sequenze che contengano lunghe ripetizioni di singoli nucleotidi (più di 3-4) d) i primers non dovrebbero presentare strutture secondarie significative e) i primers non devono presentare complementarietà tra loro Scelta dei Primers 1) Cercare la sequenza del gene di interesse in banca dati 2) Identificare inizio e fine dell’ORF (se si vuole clonare la porzione codificante) 3) Scrivere sequenza dei primers cercando di rispettare le regole dette Ad esempio >AK057661 1 61 121 181 241 301 361 421 481 541 601 661 721 781 841 901 961 1021 1081 1141 1201 1261 1321 agtcgcggct aggttgggca gtgccctggc tgatagtaga ttggatggag gatccatttg gaaaaagccc cttccagagc aggagttgca ttgatcaggc agtacattgc aacacctttg tgatgcaaca agagattgct catttcagcc aagaacaagc ttcttagtcc gaagttctgg atcatgccct ttagttttag ttctaggggt tttactgaaa agatcagata gcgagatttg ggcttctctg ctctggtctg tattgcaaga cttgggaaga aaccacagcg ttgtagagca agctgctttc ggctttagat cagcttagac agcaaagcta tacgatcata actagatgta acacaaacaa tgcggaggag tgtttcccca aaactgccca tattcttttg gacctccaat taaattaata atgtttcacc gtaagtgact aatatagaaa ggcacttttg aggaggaact actccagtag acttgcttca tttcacaaag acgtctcccc aagccaaaaa agcaccaaaa gacatggtgt tattcatacg gatatacagc cagcaaaatg gaagccgatg gaagtagaat agtgtgacat aaggtagatg aatcaagaag agctaatatg aaaaacctct ggtttggcca ttgattttgg taaaaggatg agtaaaatgg ggggtgccgg gaggagacta actcatgagg gagacaagag tttgtgtgtt gcgaagaaaa cttgggcttc cagagaccat cagctgacat ctcggaagcg gcaagactga agctccgaaa aagagacttt caaggagacc tagaatttgc accagtgtgg gtaatgacat gtattgagta ttaaaacaat ttctaaaatc cccggttctt gaagaaaaac aaaatgatac tggcccgggc aagcagacta agacaaacca gaacctcact agcagaggcg gtcgacaaca aagatggatc gaaactaaag tggaattctc gtttgacagg gataaaagag ggccaagaag ggagcttgag agtggttcgt taaagagaac aaattccagt ttcagctgct aagtatactt gctgattttt caaaattatg tcagtgttcc tacatggttg tcaataccta cgatggcgca aagggtacaa caaagttatg ctgccgggga cattttccag acttcagcga aacctcatta cggatccaga aggaaccgga gctgaagcag caactcactg ttggaggagt gtggaggtcg ttagagaggc agaaagtgtc agcatcccct ttggccacat tttgcagtag gaattcatga ataactgtat gtttaccctt gagtgttttt gttaaactca 4 Se possibile la miscela di reazione va preparata in ghiaccio o si devono utilizzare Taq (es. AmpliTaq Gold) modificate che sono in grado di partire solo dopo essere state portate a 95°C. In tal modo si ce rca di minimizzare il rischio che i primer annilino a regioni non desiderate. Nested PCR: per ridurre il rischio di aspecifici si ricorre alla nested PCR. In questo caso il prodotto di una prima reazione di PCR viene usato come stampo per una seconda razione in cui si usano oligonucleotidi posti più internamente rispetto alla coppia iniziale. Polymerase chain reaction (PCR) Consente l’amplificazione del DNA stampo. A. applicazioni B. Componenti della reazione C. procedura 5 A. PCR applicazioni Amplificazione del DNA per clonaggio di geni mutagenesi amplificazione di sequenze B. PCR componenti della reazione stampo (~104 molecole) DNA polimerasi termostabile (Taq or Pfu polimerasi) 2 DNA primers (1017 molecole) 4 deossinucleotidi tampone I 2 primers legano il DNA su filamenti opposti primers stampo Riscaldamento: separa I filamenti Raffreddamento: appaiamento dei primers 5' 5' Primer #1 Primer #2 6 C. procedura 1. denaturare il DNA stampo stampo primers DNA polimerasi Denaturaz. a 94°C 2 appaiamento dei primers appaiamento a ~ 50ºC primers legano la regione complementare 3. estensione utilizzando la DNA polimerasi estensione a 72ºC 4. ripetizione steps 1-3, ~ 35 volte (35 cicli) 7 secondo ciclo denaturazione secondo ciclo appaiamento secondo ciclo estensione 8 35 cicli stampo Prodotto finale I primers sono incorporati nel prodotto Prodotto ottenuto da I molecola di stampo = (quantità di stampo) x 2 (numero di cicli) = (1) x 235 = 3.4 x 1010 molecole 34,000,000,000 Real Time PCR La PCR Real Time è in grado di misurare in tempo reale la concentrazione iniziale di una sequenza target in un campione biologico. Gli strumenti per PCR Real Time, oltre a fungere da termociclatori, eccitano i fluorocromi presenti nei campioni e convogliano quindi la fluorescenza emessa in risposta fino ad uno spettrografo. Appositi software acquisiscono lo spettro di emissione di ogni singolo campione per tutta la durata della PCR e convertono la variazione di fluorescenza del reporter in una rappresentazione in tempo reale della cinetica d’amplificazione. 9 Il calcolo della quantità di DNA dei campioni incogniti viene effettuata determinando il ciclo della PCR (ciclo soglia, Ct) in cui viene raggiunto il valore soglia di fluorescenza del reporter e dove cioè i segnali di amplificazione specifici sono separabili da quelli del rumore di fondo del sistema. Fluorescenza Il numero dei cicli necessari perché un campione raggiunga il suo Ct è inversamente proporzionale al numero di copie target presente inizialmente. Cicli di amplificazione Il vantaggio in termini di precisione e di intervallo di quantificazione rispetto alle PCR tradizionali, è dovuto alla possibilità di quantificare il DNA al ciclo soglia, che è sempre calcolato nella fase esponenziale della reazione PCR, fase in cui i reagenti sono ancora lontani dall’esaurimento e gli elementi di variabilità sono così ridotti al minimo. 10 SYBR Green: principio SYBR Green I Utilizza una molecola fluorescente non specifica che si lega al solco minore del DNA SYBR Green All’inizio del processo di amplificazione, la miscela di reazione contiene DNA denaturato, primers e la molecola fluorescente 11 SYBR Green Dopo l’annealing dei primers, si legano poche molecole fluorescenti alla doppia elica. SYBR Green Durante l’elongazione si verifica un aumento di fluorescenza che corrisponde all’ aumento del numero di copie dell’amplicone SYBR green • Metodica semplice Possono essere utilizzati primers in uso in qualitativa • Non costosa • Non-specifica – La molecola fluorescente si lega random a tutte le doppie eliche, includendo i dimeri di primers – È necessario ottimizzare la metodica per evitare la formazione di prodotti aspecifici 12 In seguito sono state utilizzate delle sonde caratterizzate dal fatto di essere un oligonucleotide dotato sia di un fluorocromo “reporter” che di un “quencher”. Quando la sonda è intatta, la fluorescenza del reporter è spenta dalla vicinanza del quencher; ma quando la sonda è rotta dalla Taq polimerasi, l’attività dei fluorocromi viene rilevata. Uno dei saggi più diffusi per PCR real-time è il sistema “TaqMan”, il quale utilizza una sonda contenente due fluorocromi: FAM (6-carboxy-fluorescein) come reporter fluorescente legato covalentemente all’estremità in 5’ e TAMRA (6-carboxy-tetramethyl-rhodamine) come quencher fluorescente legato covalentemente all’estremità in 3’. Se è avvenuta l’ibridazione, l’attività 5’ esonucleasica della Taq polimerasi rompe la sonda interna durante la fase di estensione, emettendo un segnale di fluorescenza. La possibilità di utilizzare vari tipi di fluorocromi per marcare sonde diverse consente di rilevare contemporaneamente più di una sequenza target in una sola reazione, realizzando così una reazione in multiplex. Sonda TaqMan Presenta all’estremità 5’ un fluoroforo “Reporter” ed all’estremità 3’ una molecola“Quencher” 5’ 3’ 13 Reporter-Quencher 5’ REPORTER (R): fluorocromo ad alta energia che emette fluorescenza 3’ QUENCHER (Q): fluorocromo a bassa energia che spegne la fluorescenza del reporter R Q 5’ 3’ Se R e Q si trovano vicini, Q spegne l'effetto di R perchè i fotoni di R vengono assorbiti da Q Real-Time PCR: attività 5’>3’ esonucleasica R 5’ Q 3’ 5’ 3’ R Q 5’ 5’ 3’ R Q 5’ 5’ 3’ L’aumento di fluorescenza del Reporter è direttamente proporzionale al numero di ampliconi generati Forward primer Probe Reverse primer 14 Real-Time PCR: la reazione COMPONENTI DELLA REAZIONE: 1) DNA target 2) DNA polimerasi 3) Due oligonucleotidi 4) dNTPs 5) Probe fluorescente Run RealReal-Time thermal cycle 50 cycles 95°C 3 min 15 s Activates Amplitaq Gold 65°C 55°C 50°C 1 min 30 s 2 min Activates UNG (Uracile N-Glicosilasi) Sonde FRET (Fluorescence Resonance Energy Transfer) Simili alle sonde TaqMan perché si legano al DNA bersaglio e vengono idrolizzate, ci sono però due sonde ognuna marcata con un solo fluorocromo (accettore e donatore) Quando le sonde non sono legate alle sequenze target il segnale fluorescente proveniente dall'accettore non è rilevato donatore accettore Durante lo step di annealing PCR, entrambe le sonde FRET ibridizzano alle sequenze target: ciò avvicina il fluoroforo donatore all'accettore permettendo il trasferimento di energia tra i due fluorofori e la produzione di un segnale fluorescente da parte dell'accettore che viene rilevato. 15
Scaricare