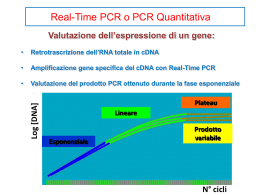
PCR quantitativa “Real-Time” e sue applicazioni in ambito biomedico Laboratorio di Oncoematologia Pediatrica Dott.ssa Virginia Libri RT-PCR convenzionale Retrotrascrizione dell’RNA in cDNA Oligo d(T) Random esameri Primer specifico per il gene Amplificazione del cDNA tramite PCR Tradizionale reazione multiciclica suddivisa in 3 fasi: Denaturazione,Annealing,Estensione Separazione elettroforetica dei prodotti di PCR (ampliconi) Gli ampliconi sono ottenuti dopo uno specifico numero di cicli di amplificazione Vengono fatti migrare su un gel di agarosio Polymerase Chain Reaction: principio Amplificazione selettiva in vitro di una sequenza di DNA target COMPONENTI DELLA REAZIONE DI PCR: 1) Sequenza di DNA target 2) DNA polimerasi termostabile (Termophilus aquaticus, Taq polimerasi) 3) Due oligonucleotidi (15-25 bp) specifici per la sequenza target 4) dNTPs Polymerase Chain Reaction: principio CICLI TERMICI: DENATURAZIONE (94-96°C) ANNEALING (50-65°C) EXTENSION (72°C) Polymerase Chain Reaction: Multiple PCR Cycles 2 copies DNA fragment to be amplified 4 copies 8 copies Polymerase Chain Reaction: vantaggi 1) Velocità e facilità d’uso In poche ore si possono ottenere milioni di copie della sequenza di DNA target 2) Sensibilità Virtualmente è possibile utilizzare il DNA di una singola cellula come template (contaminazione) 3) Robustezza Amplificazione possibile anche utilizzando DNA di bassa qualità Polymerase Chain Reaction: svantaggi 1) Solo sequenze note Necessario conoscere la sequenza del DNA da amplificare per sintetizzare i primers 2) Dimensioni e quantità limitate Dime nsioni me d ie dell’amplic o n e 0 .1 - 5 k b Quantità limitata rispetto alle tecniche di clonaggio 3) Proofreading Ta q p o l i m e r a s i m a n c a d e l l ’ a t t i v i t à 3 ’ 5 ’ esonucleasica, 20 cicli di reazione su un frammento di 1 kb produrranno 40% di frammenti con una mutazione puntiforme (1/10000) Polymerase Chain Reaction: resa Resa teorica: 2n P=(2)n T Il prodotto (P) incrementa esponenzialmente con il numero di cicli di PCR (n) Il prodotto di PCR dipende da T,numero di copie di template di partenza Log[DNA] Plateau Lineare Esponenziale Prodotto variabile N° cicli termici Polymerase Chain Reaction: plateau Resa effettiva: effetto plateau Il processo di duplicazione non procede “all’infinito”, esso è limitato da: Quantità dei primers Attività della Taq polimerasi Reannealing dei filamenti Raggiunto il plateau non si osserva più un incremento nei prodotti Il plateau non dipende da T Anche se la quantità iniziale di template è la medesima, il plateau è raggiunto in tempi diversi ed in cicli diversi 25 PCR product 20 15 10 5 0 0 10 20 Cycle 30 40 Soluzioni Utilizzare i dati ottenuti durante la fase esponenziale Il prodotto di PCR è proporzionale al template iniziale Questo è reso possibile mediante il rilevamento, di una fluorescenza, che è proporzionale al prodotto di PCR La fluorescenza, durante ogni ciclo di amplificazione, può essere rilevata utilizzando uno strumento quantitativo ma anche dei marcatori fluorescenti il cui accumulo segue la stessa cinetica della reazione di PCR RT-PCR convenzionale Reverse transcription Real-time RT-PCR Reverse transcription PCR reaction Nested PCR reaction Gel electrophoresis DNA sequencing Southern blot Manual or automated analysis PCR reaction Quantitative result Perché Real-Time? Misura l'amplificazione in tempo reale durante la fase esponenziale della PCR, quando cioè l'efficienza di amplificazione è influenzata minimamente dalle variabili di reazione, permettendo di ottenere risultati molto più accurati rispetto alla PCR tradizionale "end point" RT-PCR quantitativa •Rilevamento della fluorescenza associata all’amplificazione •Il prodotto di PCR non viene analizzato su gel di agarosio •Analisi del prodotto di fluorescenza tramite computer Plot lineare Incremento di fluorescenza Cicli di PCR Analisi tramite software Chimiche fluorescenti per PCR Real-Time •La fluorescenza si genera durante la PCR per effetto di diverse possibili reazioni chimiche •Le chimiche principali sono basate sia sul legame di coloranti fluorescenti che si intercalano nella doppia elica di DNA, come il SYBR Green, sia sull'ibridazione di sonde specifiche. SYBR Green SYBR Green:SYBR principio Green I Utilizza una molecola fluorescente non specifica che si lega al solco minore del DNA SYBR Green All’inizio del processo di amplificazione, la miscela di reazione contiene DNA denaturato, primers e la molecola fluorescente SYBR Green Dopo l’annealing dei primers, si legano poche molecole fluorescenti alla doppia elica. SYBR Green Durante l’elongazione si verifica un aumento di fluorescenza che corrisponde all’ aumento del numero di copie dell’amplicone SYBR green Metodica semplice Possono essere utilizzati primers in uso in qualitativa Non costosa Non-specifica La molecola fluorescente si lega random a tutte le doppie eliche, includendo i dimeri di primers È necessario ottimizzare la metodica per evitare la formazione di prodotti aspecifici Curva di dissociazione Dopo l'amplificazione, i campioni vengono riscaldati e la variazione dell'energia di fluorescenza viene monitorata per generare una curva di dissociazione Curva di dissociazione Tm melting peak 82.6 88.1 TaqMan TaqMan La Real-Time PCR si può realizzare mediante l’impiego: coloranti intercalanti ( es. SYBR green), che si legano in maniera aspecifica a tutto il DNA sonde ad ibridazione, specifiche per il frammento di interesse, marcate con molecole fluorescenti Esistono diversi tipi di sonde: Dual-labeled (come le sonde TaqMan) Molecular beacons Scorpion Sonde FRET (Fluorescence Resonance Energy Transfer) Sonde TaqMan La sonda di tipo TaqMan è un oligonucleotide che, come i primers della PCR, viene disegnato per essere complementare alla sequenza bersaglio da amplificare R 3’ 5’ 5’ Primer 3’ 5’ Q 3’ 5’ 3’ Primer 5’ La sonda è disegnata in modo da ibridarsi all’interno del frammento amplificato nella reazione di PCR 3’ Dimensione dell’amplicone Forward Probe Reverse Sonda TaqMan Presenta all’estremità 5’ un fluoroforo “Reporter” ed all’estremità 3’ una molecola “Quencher” 5’ 3’ Reporter-Quencher Dye Quencher 5,6 FAM BHQ-1/TAMRA HEX/JOE BHQ-2 Texas Red/ROXBHQ-2 Cy5/Quasar670 BHQ-2 ( or-3) 6-carbossifluoresceina 6-carbossitetrametilrodamina Reporter-Quencher 5’ REPORTER (R): fluorocromo ad alta energia che emette fluorescenza 3’ QUENCHER (Q): fluorocromo a bassa energia che spegne la fluorescenza del reporter R Q 5’ 3’ Se R e Q si trovano vicini, Q spegne l'effetto di R perchè i fotoni di R vengono assorbiti da Q Real-Time PCR: attività 5’>3’ esonucleasica 3’ 5’ 3’ R Q 5’ R 3’ Q 5’ 5’ R 3’ 5’ Q 5’ L’aumento di fluorescenza del Reporter è direttamente proporzionale al numero di ampliconi generati Forward primer Probe Reverse primer Real-Time PCR: la reazione COMPONENTI DELLA REAZIONE: 1) DNA target 2) DNA polimerasi 3) Due oligonucleotidi 4) dNTPs 5) Probe fluorescente Run Real-Time thermal cycle 50 cycles 95°C 3 min Activates Amplitaq Gold 15 s 65°C 55°C 50°C 30 s 2 min Activates UNG (Uracile N-Glicosilasi) 1 min Curve di amplificazione Fluorescenza Plot lineare Cicli di amplificazione Per ogni campione si ottiene una curva di amplificazione il cui CT(=Threshold Cycle) è inversamente proporzionale alla quantità di template iniziale Normalised reporter fluorescence (Rn) Plot di amplificazione Linea soglia scelta d a l l ’ o p e r a t o r e In maniera da intersecare le curve di tutti i campioni nella fase esponenziale Indica il valore al di sopra del quale inizia l’accumulo di un amplificato E’ il ciclo della reazione di amplificazione in cui il segnale di fluorescenza del campione è maggiore rispetto a quello d e l l a T h r e s h o l d PCR cycle number Quantificazione ASSOLUTA i campioni sono quantificati in modo assoluto Necessita di standard di cui si conosce la concentrazione assoluta (utilizzo di una standard curve) Per tutti gli unknowns devono essere saggiate identiche quantità di campioni RELATIVA la quantificazione viene effettuata paragonando i CT Necessita di controlli endogeni (non si utilizza una standard curve) Gli unknowns vengono “quantificati” paragonando il loro CT con quello del controllo endogeno Quantitativa assoluta Ct 10 1 35 10 2 10 3 unknown sample 10 4 25 10 5 10 6 3500 copies 10 7 15 1 2 3 4 5 6 7 log10 quantity Il valore così ottenuto viene normalizzato rispetto a quello di un gene espresso costitutivamente (-actina, GAPDH, ATPs, 2 etc) Quantitativa relativa Plot di amplificazione Control Sample Numero di cicli CT Quantitativa relativa: analisi dei dati Normalizzare il target con un controllo endogeno (r) espresso costitutivamente (CT) Comparare ciascun CT così ottenuto con il CT di un trattamento di controllo anche detto “calibratore” (cb) (CT) 2- (CT,r- CT,cb)= 2- CT Il valore così ottenuto permette di determinare la concentrazione relativa del target Sonde FRET (Fluorescence Resonance Energy Transfer) Simili alle sonde TaqMan perché si legano al DNA bersaglio e vengono idrolizzate, ci sono però due sonde ognuna marcata con un solo fluorocromo (accettore e donatore) Quando le sonde non sono legate alle sequenze target il segnale fluorescente proveniente dall'accettore non è rilevato Durante lo step di annealing PCR, entrambe le sonde FRET ibridizzano alle sequenze target: ciò avvicina il fluoroforo donatore all'accettore permettendo il trasferimento di energia tra i due fluorofori e la produzione di un segnale fluorescente da parte dell'accettore che viene rilevato donatore accettore Molecular Beacons I "molecular beacons" contengono un fluoroforo e un quencher non fluorescente alle estremità opposte di un oligonucleotide, che sono disegnate in modo da essere complementari tra loro formando una struttura stem-loop La vicinanza del quencher al reporter fluorescente impedisce l’emissione di fluorescenza fluoroforo quencher Il loop è complementare ad una sequenza all'interno del prodotto amplificato Molecular Beacons Durante lo step di annealing PCR, la sonda ibridizza alla sua sequenza target: ciò separa il colorante fluorescente dal r e p o r t e r, p r o d u c e n d o u n segnale fluorescente La quantità di fluorescenza prodotta ad ogni ciclo, o dopo la PCR, dipende dalla quantità di prodotto specifico in quel d a t o m o m e n t o EXCITATIONE FRET ANNEALING Amplicon A d i f f e r e n z a d e l l e s o n d e Ta q M a n , l e m o l e c u l a r b e a c o n s n o n vengono distrutte durante durante la reazione di amplificazione per cui possono reibridizzarsi durante il successivo ciclo Probes : Scorpions La progettazione delle scorpion probes è simile a quella delle molecular probes, con la differenza che al 3’ terminale del probe vi è una sequenza, PCR primer, che è specifica per l’ estensione del target Questa sequenza è legata al 5’ terminale di un primer per mezzo di un “blocker” 5’ 3’ Probes : Scorpions Step 1 Lo Scorpions primer è esteso su un target di DNA Scorpions primer Probe Target di DNA Probes : Scorpions Step 2 Durante il processo di denaturazione si verifica l’allontanamento del quencher dal reporter e del primer di innesco dal DNA target Q Primer di innesco R DNA target Probes : Scorpions Step 3 Raffreddandosi lo Scorpion esteso subisce un riarrangiamento interno ed emette fluorescenza in maniera target specifica. Un primer non esteso viene quenciato Riassumendo: Metodi di rilevamento della fluorescenza SYBR Green TaqMan Molecular Beacons Scorpion probe Real-Time PCR: applicazioni Quantificazione virale Quantificazione dell’espressione genica Efficacia della terapia farmacologica Misura dei danni al DNA Controllo di qualità e validazione dei saggi Detenzione dei patogeni Controllo degli OGM Genotyping MRD MRD (Minimal Residual Disease): Quota residua di cellule neoplastiche non eradicate dalla terapia di induzione della remissione o dalle successive misure terapeutiche Tali elementi neoplastici, presenti ad un livello inferiore alla capacità di rilevazione delle metodiche convenzionali, sono in grado di espandersi e dare origine alla recidiva TH (Tirosina Idrossilasi) Enzima che catalizza la prima reazione di biosintesi delle catecolamine HVA-VMA TH (Tirosina Idrossilasi) Perché la scelta di studiare l’espressione della TH? Le cellule di Neuroblastoma (NB) hanno la caratteristica di sintetizzare catecolamine, che risultano alterate nel 95% dei pazienti. La TH è uno dei marker tumorali tessuto-specifici espresso dalle cellule di NB,ma non dagli altri tumori a piccole cellule rotonde Pertanto lo studio di questo enzima può essere utilizzato per identificare le metastasi midollari al momento della diagnosi e per monitorare lo stato della MRD in modo da individuare eventuali segnali precoci di ricaduta in pazienti i n r e m i s s i o n e c l i n i c a TH (Tirosina Idrossilasi) Possibile ruolo della TH come fattore prognostico negativo nel Neuroblastoma (Pession et al. Oncology Reports 10:357-362,2003) La concordanza con le analisi diagnostiche standard evidenziata nello stadio IV consente di considerare la TH quale utile parametro predittivo alla diagnosi Negli stadi localizzati, dove in 3/24 (12.5%) è stata osservata la positività isolata dell’espressione della TH suggestiva di infiltrazione midollare minima, questa indagine potrebbe rappresentare un valido strumento di approfondimento diagnostico La valutazione della TH inoltre potrebbe entrare tra le analisi diagnostiche indici di contaminazione neoplastica di cellule staminali emopoietiche periferiche o midollari raccolte in pazienti positivi alla diagnosi (studio in corso)
Scaricare