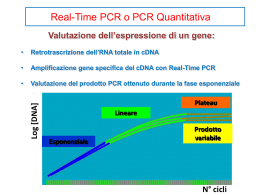
End point PCR vs quantitative RealReal-Time PCR PCR - Polymerase Chain Reaction ideata nel 1983 da Kary B. Mullis il quale ottenne, per questo, il premio Nobel per la chimica (1993). Questa tecnica, utilizzando i principi della duplicazione del DNA, permette di ottenere in poco tempo notevoli quantità di materiale specifico Polymerase Chain Reaction: Multiple PCR Cycles 2 copies DNA fragment to be amplified Resa: 2n 4 copies 8 copies Polymerase Chain Reaction: principio Amplificazione selettiva in vitro di una sequenza di DNA target COMPONENTI DELLA REAZIONE DI PCR PCR: 1) Sequenza di DNA target 2) DNA polimerasi termostabile (Termophilus aquaticus,, Taq polimerasi) aquaticus polimerasi) 3) Due Primers Primers,, oligonucleotidi sintetici (20(20-25 bp bp)) complementari a sequenze fiancheggianti il tratto di DNA da studiare 4) dNTPs 5) Ioni Mg++ Polymerase Chain Reaction: principio CICLI TERMICI: DENATURAZIONE (94 (94--96 96° °C) ANNEALING (50 (50--65 65° °C) EXTENSION (72 (72° °C) END POINT PCR Il campione si sottopone al numero di cicli stabiliti (30(30-40), alla fine della reazione un’aliquota si sottopone ad elettroforesi su gel di agorosio per verificare: 1. la lunghezza dell’amplificato (controllo del peso molecolare in bp mediante ladder ladder)) 2. L’assenza di frammenti aspecifici 3. La quantità di amplificato ottenuto (proporzionale all’intensità della banda) PCR PHASES L’amplificazione di ogni target è definita da 3 fasi: 1 – esponenziale; 2 – lineare; 3 – plateau. All reagents are present Some of the reagents start wearing out Cycle All the reagents finished PCR: effetto plateau Il processo di duplicazione non procede “all’infinito”, esso è limitato da: Quantità dei primers primers,, dNTP, dNTP, MgCl2, Buffer Attività della Taq polimerasi Reannealing dei filamenti Raggiunto il plateau non si osserva più un incremento nei prodotti La PCR end-point non quantitativo ma qualitativo. è un metodo Alla fine della reazione non sempre la quantità di DNA del campione d’origine è proporzionale all’intensità della banda ottenuta. Utilizzando particolari accorgimenti può diventare un metodo semi-quantitativo. Polymerase Chain Reaction: vantaggi 1) Velocità e facilità d’uso In poche ore si possono ottenere milioni di copie della sequenza di DNA target 2) Sensibilità Virtualmente è possibile utilizzare il DNA di una singola cellula come template (contaminazione) 3) Robustezza Amplificazione possibile anche utilizzando DNA di bassa qualità Polymerase Chain Reaction Reaction:: svantaggi 1) Solo sequenze note Necessario conoscere la sequenza del DNA da amplificare per sintetizzare i primers. 2) Dimensioni e quantità limitate Dimensioni medie dell’amplicone 0.1-5kb Quantità limitata rispetto alle tecniche di clonaggio. 3) Proofreading Taq polimerasi manca dell’attività 3’ 3’→ →5’ esonucleasica, esonucleasica, 20 cicli di reazione su un frammento di 1 kb produrranno ∼40% di frammenti con una mutazione puntiforme (∼ (∼1/10000). 4) Analisi in End Point ed effetto Plateau Per analizzare il campione bisogna aspettare la fine della reazione; raggiunta la fase di plateau la quantità di amplificato non è più proporzionale alla concentrazione iniziale di campione. campione. Soluzioni Utilizzare i dati ottenuti durante la fase esponenziale in cui il prodotto di PCR è proporzionale al template iniziale Questo è reso possibile mediante il rilevamento di una fluorescenza il cui accumulo segue la stessa cinetica della reazione di PCR e che che,, quindi quindi,, è proporzionale al prodotto di PCR Si utilizzano particolari termociclatori in grado misurare la fluorescenza emessa da particolari fluorofori ad ogni ciclo. ciclo. Quantitative RealReal-time PCR Quantitative RealReal-time PCR Tecnica che consente la simultanea amplificazione e quantificazione del DNA target. AMPLIFICAZIONE: prevede essenzialmente gli step di una classica PCR QUANTIFICAZIONE: effettuata aggiungendo composti la cui fluorescenza emessa ad ogni ciclo di reazione è proporzionale alla quantità di amplificato. Real-Time PCR: la reazione COMPONENTI DELLA REAZIONE: 1) DNA target 2) DNA polimerasi 3) Due oligonucleotidi 4) dNTPs 5) Fluorocromo Perché RealReal-Time? Misura l'amplificazione in tempo reale durante la fase esponenziale della PCR, quando cioè l'efficienza di amplificazione è influenzata m i n i m a m e n t e d a l l e v a r i a b il i d i r e a z i o n e , permettendo di ottenere risultati molto più accurati rispetto alla PCR tradizionale "end point" Chimiche fluorescenti per PCR RealReal-Time •La fluorescenza si genera durante la PCR per effetto di diverse possibili reazioni chimiche •Le chimiche principali sono basate sia sul legame di coloranti fluorescenti che si intercalano al dsDNA, come il SYBR Green, sia sull'ibridazione di sonde specifiche. SYBR Green SYBR Green:SYBR principio Green I Utilizza una molecola fluorescente non specifica che si lega al solco minore di tutte le molecole di dsDNA. dsDNA. SYBR Green All’inizio del processo di amplificazione, la miscela di reazione contiene DNA denaturato, primers e la molecola fluorescente SYBR Green Dopo l’annealing dei primers, si legano poche molecole fluorescenti alla doppia elica. SYBR Green Durante l’elongazione si verifica un aumento di fluorescenza che corrisponde all’aumento del numero di copie dell’amplicone SYBR green Metodica semplice Possono essere utilizzati primers in uso in qualitativa Non costosa Non-specifica Non La molecola fluorescente si lega random a tutte le doppie eliche eliche,, includendo i dimeri di primers È necessario ottimizzare la metodica per evitare la formazione di prodotti aspecifici Curva di dissociazione Dopo l'amplificazione, i campioni vengono riscaldati e la variazione dell'energia di fluorescenza viene monitorata per generare una curva di dissociazione Curva di dissociazione •T dissociazione proporzionale alla grandezza dell’amplimero dell’amplimero.. •La presenza di una o più curve di dissociazione dipende dal fatto che la popolazione di amplimeri sia omogenea o eterogenea. •L’ampiezza del picco è proporzionale alla quantità di amplificato. TaqMan TaqMan La Real Real--Time PCR si può realizzare mediante l’impiego: l’impiego: coloranti intercalanti ( es es.. SYBR green), che si legano in maniera aspecifica a tutto il DNA sonde ad ibridazione,, specifiche per il frammento di interesse, marcate con molecole fluorescenti Esistono diversi tipi di sonde: Dual--labeled (come le sonde TaqMan) Dual Molecular beacons Scorpion Sonde FRET (Fluorescence Resonance Energy Transfer) Sonde TaqMan La sonda di tipo TaqMan è un oligonucleotide che, come i primers della PCR, viene disegnato per essere complementare alla sequenza bersaglio da amplificare R 5’ 3’ 5’ Primer 3’ 5’ Q 3’ 5’ Primer 3’ 5’ La sonda è disegnata in modo da ibridarsi all’interno del frammento amplificato nella reazione di PCR 3’ Forward Probe Reverse Sonda TaqMan Presenta all’estremità 5’ un fluoroforo “Reporter” ed all’estremità 3’ una molecola “Quencher” 5’ 3’ Reporter--Quencher Reporter 5’ REPORTER (R): fluorocromo ad alta energia che emette fluorescenza 3’ QUENCHER (Q): fluorocromo a bassa energia che spegne la fluorescenza del reporter R Q 5’ 3’ Se R e Q si trovano vicini, Q spegne l'effetto di R perchè i fotoni di R vengono assorbiti da Q Reporter--Quencher Reporter Dye Quencher 5,6 FAM BHQ-1/TAMRA HEX/JOE BHQ-2 Texas Red/ROXBHQ-2 Cy5/Quasar670 BHQ-2 ( or-3) 6-carbossifluoresceina 6-carbossitetrametilrodamina Real-Time PCR: attività 5’>3’ esonucleasica R 5’ Q 3’ 5’ 3’ R Q 5’ 5’ 3’ R Q 5’ 3’ 5’ L’aumento di fluorescenza del Reporter è direttamente proporzionale al numero di ampliconi generati Forward primer Probe Reverse primer RT RT--PCR quantitativa •La fluorescenza emessa ad ogni ciclo di amplificazione viene rilevata e analizzata tramite computer. Plot lineare Incremento di fluorescenza Cicli di PCR Normalised reporter fluorescence (Rn) Plot di amplificazione Linea soglia scelta d a l l ’ o p e r a t o r e In maniera da intersecare le curve di tutti i campioni nella fase esponenziale Indica il valore al di sopra del quale inizia l’accumulo di un amplificato E’ il ciclo della reazione di amplificazione in cui il segnale di fluorescenza del campione è maggiore rispetto a quello d e l l a T h r e s h o l d PCR cycle number Curve di amplificazione Fluorescenza Plot lineare Cicli di amplificazione Per ogni campione si ottiene una curva di amplificazione il cui CT(=Threshold Cycle) è inversamente proporzionale alla quantità di template iniziale Quantificazione ASSOLUTA i campioni sono quantificati in modo assoluto Necessita di standard di cui si conosce la concentrazione assoluta (utilizzo di una standard curve) Per tutti gli unknowns devono essere saggiate identiche quantità di campioni RELATIVA la quantificazione viene effettuata paragonando i CT Necessita di controlli endogeni (non si utilizza una standard curve) Gli unknowns vengono “quantificati” paragonando il loro ∆CT con quello del controllo endogeno Quantitativa assoluta Ct 10 1 35 10 2 10 3 unknown sample 10 4 25 10 5 10 6 3500 copies 10 7 15 1 2 3 4 5 6 7 log10 template quantity Il valore così ottenuto viene normalizzato rispetto a quello di un gene espresso costitutivamente (β-actina, GAPDH, ATPs, β2 etc etc)) Quantitativa relativa Effettuata comparando i valori di Ct per determinare il cambiamento di espressione di un gene target in un campione rispetto ad un altro campione scelto come calibratore. Plot di amplificazione Control Sample Numero di cicli ∆CT Quantitativa relativa Per ogni esperimento in quantificazione relativa è necessario: • TARGET – La sequenza di DNA da analizzare. • CALIBRATORE – Il campione da usare come riferimento per l’analisi comparativa (Trattato/non Trattato, campione al T0 in uno studio time course) • CONTROLLO ENDOGENO – Un gene espresso costitutivamente in tutti i campioni analizzati necessario per normalizzare i dati rispetto alla quantità di DNA caricato e a variazioni di efficienza della reazione. Quantitativa relativa: analisi dei dati Normalizzare il target con un controllo endogeno (r) espresso costitutivamente (∆CT) Comparare ciascun ∆CT così ottenuto con il ∆CT di un calibratore (cb) (∆∆ ∆∆C ∆∆ T) CT,r-- ∆CT, CT,cb cb)= 2- ∆∆CT ∆∆CT 2- (∆CT,r Il valore così ottenuto permette di determinare la concentrazione relativa del target Step per il calcolo del 2- ∆∆CT ∆∆ CT 1. Normalizzazione con un controllo endogeno (HK) Ct gene interesse – Ct HK = ∆Ct 2. Normalizzazione con il calibratore ∆Ct Campione – ∆Ct Calibratore = ∆∆Ct 3. Applicare la formula 2- ∆∆CT ∆∆ CT ESEMPIO Campione Ct HK Ct gene terget (IL-10) Brain (calibratore) 14 32 Liver 16 28 Di quanto varia l’espressione dell’ IL-10 tra Liver e Brain? 1. ∆Ct Brain = 18 ∆Ct Liver = 12 2. ∆Ct campione – ∆Ct calibratore = 12-18 = -6 3. 2- ∆∆CT = 26 = 64 L’IL-10 è 64 volte più espressa nel Liver rispetto al Brain!
Scaricare