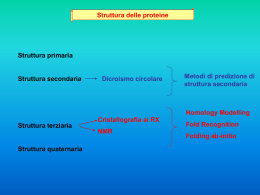
Struttura delle proteine Struttura primaria Struttura secondaria Dicroismo circolare Metodi di predizione di struttura secondaria Homology Modelling Struttura terziaria Struttura quaternaria Cristallografia ai RX NMR Fold Recognition Folding ab-initio Programmi per analizzare la struttura primaria delle proteine dal sito Expasy: Colorseq colora gli amminoacidi (idrofili, idrofobici, carichi positivamente o negativamente,aromatici) ThreetoOne converte gli amminoacidi dalla nomenclatura tre lettere a quella ad una lettera ProtScale calcola l’idrofobicità degli amminoacidi in una sequenza SYFPEITHI – predice i siti di binding di peptidi con MHC type I and II Coils predice regioni random coil in proteine Compute pI/MW valuta il punto isoelettrico (pH a cui la carica netta è 0) ed il peso molecolare Struttura secondaria Metodi di predizione della struttura secondaria delle proteine: Metodi di Chou-Fasman si basa sull’analisi statistica della composizione in residui delle strutture secondarie presenti nella PDB. (http://fasta.bioch.virginia.edu/fasta_www2/fasta_www.cgi?rm=misc1) GOR si basa sull’analisi statistica della composizione in residui delle strutture secondarie presenti nella PDB. (http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_gor4.html) AGADIR per predire la percentuale di residui in elica (http://www.embl-heidelberg.de/Services/serrano/agadir/agadir-start.html) PHD prende in input o una sequenza o un allineamento multiplo ed usa le reti neurali. (http://www.embl-heidelberg.de/predictprotein/predictprotein.html) Vuole una registrazione PSIPRED utilizza un sistema di due reti neurali. (http://bioinf.cs.ucl.ac.uk/psipred/) PREDATOR si basa sull’applicazione del metodo del k-esimo vicino che usa le reti neurali (http://bioweb.pasteur.fr/seqanal/interfaces/predator-simple.html) JPRED3 (http://www.compbio.dundee.ac.uk/Software/JPred/jpred.html) fa un consensus di vari metodi Chou and Fasman Prediction PHD Vuole una registrazione Output of JPred prediction ALTRI PROGRAMMI Struttura terziaria Banche dati che riguardano la struttura delle proteine: PDB (Protein Data Bank) DSSP (Dictionary of Protein Secondary Structure) raccolta delle strutture secondarie. HSSP (Homology derived Secondary Structure of Proteins) contiene informazioni utili per costruire modelli di proteine a struttra non nota e che abbiano una percentuale di identità di sequenza maggiore del 30%. FSSP (Fold classification based on Secondary Structure alignment of Proteins) include l’allineamento con le proteine di struttura simile e riporta i residui che sono equivalenti nelle strutture. PDBsum (http://www.ebi.ac.uk/thornton-srv/databases/pdbsum/) riassume per ogni proteina tutte le informazioni derivanti dalle varie banche dati correlati. SCOP [Structural Classification of Proteins] organizza le strutture proteiche gerarchicamente seguendo criteri evolutivi e di similarità strutturale. CATH presenta una classificazione strutturale simile a quellla offerta da SCOP, basata su confronti di strutture. Programmi di visualizzazione di proteine (ad esempio RASMOL) Le possibili opzioni di visualizzazione: Wireframe consente di visualizzare tutti gli atomi descritti nel file di coordinate come intersezioni di segmenti che rappresentano i legami chimici tra gli atomi stessi. Trace indica la linea spezzata che unisce gli atomi dei soli carboni alfa. Space filling che utilizza sfere di raggio pari ai raggi atomi di Van der Waals. Questom odo è molto efficace per definire la superifcie ed il volume occupato dalla proteina. Altri programmi sono: SwissPDBViewer che è molto utilizzato anche per costruire modelli di proteine Per confrontare strutture proteiche possono essere utilizzati vari programmi: DALI (http://www2.embl-ebi.ac.uk/dali/) carboni alfa di coppie di proteine. paragona mappe di contatti tra SSAP (http://www.biochem.ucl.ac.uk/~orengo/ssap.html) confronta strutture proteiche trasformando ciascuna delle due proteine in una matrice di distanze intramolecolari in cui ogni elemento della matrice (i,j) corrisponde ad un vettore che uniscel’ i-esimo al j-esimo carbonio alfa. CE (http://cl.sdsc.edu/ce.html) confronta coppie di strutture proteiche in base all’identificazione di frammenti allineati. Qualche esempio?
Scaricare