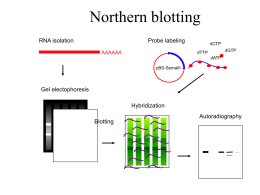
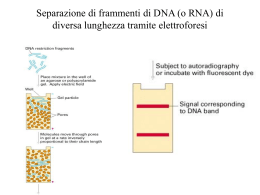
Arrays di acidi nucleici Sia i macroarrays che i microarrays sono stati sviluppati per soddisfare l’esigenza di misurare contemporaneamente l’espressione di più geni. Entambe le tecnologie si basano sullo stesso principio: 1. Come sonda si usano olgonucleotidi o molecole di cDNA non marcati, immmobilizzati in posizioni precise di un supporto solido A B C D 1 a b g d 2 e z h q 3 i k l m 4 n x o Array p 2. L’array viene ibridizzato con una miscela complessa di molecole marcate rappresentative dell’mRNA espresso dalle cellule in esame mRNA RT Nucleotidi marcati cDNA I saggi di microarray sono basati sul principio dell’ibridazione inversa Macrorray • Le molecole sonda vengono legate a membrane di nylon • Come tracciante viene utilizzata la radioattività • Analisi di qualche decina o poche centinaia di geni Microrray: tecnologia Affymetrix • Le molecole sonda sono oligonucleotidi sintetizzati direttemente su microchip di silicio con un metodo fotolitografico. • Su ogni microchip vengono sintetizzati fino a 400000 oligonucleotidi diversi. •La metodica è stata sviluppata in modo da permettere misurazioni assolute dell’abbondanza dei singoli mRNA Schema metodica mRNA TGAACTGATAGATGACGTAG Poly A Design complementary oligo ACTTGACTATC TACTGCATC Chemically synthesize oligo on chip 107- 108 identical oligos per feature Label – Label – Hybridize labeled total mRNA Detect label Microrray: tecnologia Affymetrix 50µm ~ 50 - 400 chips/wafer 50µm Thousands of identical probes/feature 1.28cm 1.28cm up to ~ 400,000 features/chip Sintesi fotolitografica Lamp Mask Chip Ibridazione e rivelazione Biotin Biotin 7 8 10 -10 identical probes/feature Phycoerythrin Phycoerythrin Streptavidin Streptavidin Biotin Biotin Rappresentazione di ogni trascritto sul chip Perfect Match mRNA * Mismatch * * * * * Perfect Match Mismatch • Per ogni gene vengono sintetizzati 20 oligonucleotidi uguali all’mRNA (perfect match). • Per ognuno di questi, ne viene sintetizzato un altro con una base sbagliata (mismatch). • L’espressione del gene si ottiene sommando le letture dei perfect match e sottraendo quelle dei mismatch. • Questi accorgimenti sono indispensabili per eliminare i problemi dovuti alla eventale ibridizzazione disugule o non specifica degli oligonucleotidi. Letture possibili Detectable Perfect Match Mismatch Undetectable Undetectable PIN PRINTING INK JET PRINTING cDNA Microarray Modalità di marcatura Modalità di marcatura Microrray di cDNA (o di oligonucleotidi lunghi) • Le molecole sonda sono cDNA o oligonucleotidi lunghi 70-80 paia di basi, sintetizzati tradizionalmente e egati ad un vetrino da microscopio per mezzo di un processo di stampa a getto. • Su ogni vetrino trovano posto fino a 20000 geni. • L’intensità del segnale per ogni gene dipende dai livelli di messaggero e dall’efficienza di ibridazione. Pertanto questa metodica non consente misurazioni assolute ma solo relative. • Metodica comparativa. • Per confrontare tra di loro più campioni è necessario confrontarli tutti con un campione standardizzato. mRNA Reverse transcription Labeled 1st strand cDNA Sample 1 (test) Sample 2 (reference) Hybridization on the cDNA microarray Bead Arrays Analisi statistica a tutti i livelli Identificazione degli spot Analisi statistica a tutti i livelli Eliminazione degli artefatti Analisi statistica a tutti i livelli Normalizzazione Analisi statistica a tutti i livelli Identificazione dei geni modulati Analisi statistica a tutti i livelli Clustering dei dati Analisi statistica a tutti i livelli Clustering dei dati Attenzione !!! L’enorme numero di geni analizzati dai microarray è il punto più forte, ma anche più debole della metodica. Infatti sono possibili moltissimi errori (importanza di avere campioni replicati), e il trattamento dell’informazione non è banale! L’acquisizione dei dati è solo la parte iniziale della procedura. La parte più complicata è l’elaborazione della enorme quantità di dati generati da questi esperimenti, necessaria per rispondere ai quesiti biologici di partenza. I dati più significativi devono essere poi verificati con altri sistemi (northern, real time RT-PCR) Importanza della bioinformatica per la determinazione del significato biologico degli esperimenti Applicazioni 1. Definizione delle basi molecolari e identificazione di nuovi markers prognostici per neoplasie e altre patologie Applicazioni 1. Definizione delle basi molecolari e identificazione di nuovi markers prognostici per neoplasie e altre patologie Applicazioni 2. Studio del controllo trascrizionale. In particolare, identificazione dei siti di legame per i fattori trascrizionali coinvolti nella coregolazione Applicazioni Applicazioni 3. Annotazione del genoma, ossia identificazione delle regioni effettivamente trascritte Applicazioni 4. Farmacogenomica e tossicogenomica
Scaricare