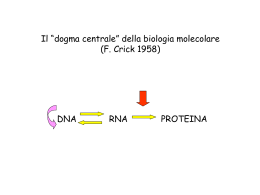
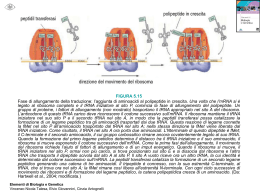
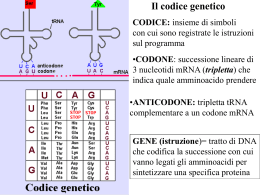
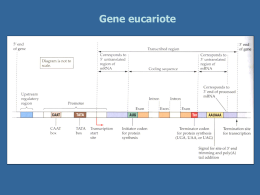
Sintesi proteica Definizione La sintesi proteica è costituita da una sequenza di eventi che portano alla formazione di un polimero di amminoacidi (proteina o polipeptide) ad opera dei ribosomi a partire da una sequenza di codoni di una molecola di mRNA TRADUZIONE Consiste nella trasformazione dall’alfabeto a 4 lettere degli acidi nucleici (basi azotate) all’alfabeto completamente diverso delle PROTEINE (amminoacidi,AA) Nel processo di traduzione sono coinvolte più di 100 macro-molecole tRNA mRNA Proteine Ribosomi rRNA Proteine ribosomiali (Basiche) …Pre-traduzione Flusso Genetico DNA Replicazione 1 DNA RNA 2 DNA Proteine Basi Azotate:Adenina-Timina; Guanina-Citosina DNA-polimerasi Trascrizione DNA RNA Basi Azotate:Adenina-Uracile; Guanina-Citosina RNA-polimerasi-DNA dipendenti Il meccanismo di trascrizione non possiede sistemi di controllo aposteriori Tasso di errore: 10-4 Individua il sito specifico per la trascrizione sul filamento stampo di DNA mRNA-tRNA-rRNA Trascrizione Da una porzione specifica del DNA (CISTRONE) in direzione 5’3’ Corrisponde uno specifico mRNA complementare 3’5’ L’mRNA prodotto all’interno del nucleo viene traslocato nel citoplasma Trasformato in «matrice» per il corretto allineamento degli aa di un nuovo polipeptide Traduzione o Sintesi Proteica Molecole necessarie per la traduzione mRNA-ribosomi Aminoacidi tRNA Differenti classi di enzimi Aminoacil-tRNA sintetasi Peptidil-trasferasi Ioni Mg2+ Nucleosidi trifosforici ATP GTP Fattori proteici che intervengono durante le diverse fasi della sintesi proteica Caratteristiche mRNA eucariotico • Ad entrambe le estremità dell’mRNA (5’ e 3’) troviamo sequenze che non vengono tradotte, ma hanno un ruolo come regolatore • Estremità 5’: la sequenza non tradotta TRATTO LEADER presenta sempre un nucleoside «guanosina modificato» (Cap 5’) e seguito da codone di inizio AUG • Il TRATTO LEADER ha la funzione di: -impedire al tratto 5’ mRNA di non essere digerito dall’enzima esonucleasi -favorire il trasporto dell’mRNA fuori dal nucleo -favorire il posizionamento dell’mRNA sul ribosoma per l’inizio della traduzione • Estremità 3’: la sequenza non tradotta TRAILER segue uno dei codoni di terminazione UAG,UAA,UGA • È costituito da una serie di ribonucleotidi Adenina (50-250)coda di poli A Aiuta i ribosomi a riconoscere l’mRNA come una molecola da dover essere tradotta Traduzione o Sintesi Proteica 3 tappe Fase Di Inizio Fase Di Allungamento Fase Di Terminazione 1. Fase di inizio @ La traduzione dell’mRNA avviene in direzione 5’3’ ed inizia dall’estremità N-terminale della proteina futura @ Nelle cellule eucariote il primo aminoacido è rappresentato dalla metionina (met) ovvero l’aa codificato dal codone di inizio AUG che troviamo all’estremità 5’ dell’mRNA @ Fattori di inizio (eIF-1,eIF-1A,eIF-3) si associano alla subunità 40S ribosomiale quando inizia la sintesi proteica tRNA+Met+GTP+fattore di inizio eIF-2 Complesso di pre-inizio Subunità minore 40S eIF-4A eIF-4E eIF-4G Si associa al Cap 5 Complesso di inizio Complesso di inizio Met-tRNA Il complesso di inizio scorre lungo tutto il filamento di mRNA direzione 5’ 3’ fino ad arrivare al codone AUG di inizio Il tRNA iniziatore o anche metionil-tRNA lega al codone AUG il suo anticodone complementare UAC Rilascio dei fattori di inizio sia per l’idrolisi del GTP sia per eIF-5 e si associa la subunità maggiore 60S L’attacco della subunità 60S richiede dispendi di energia generato dll’idrolisi del GTP-legato in GDP+Pi (fosfato inorganico) 2. Fase di Allungamento @ Ha inizio quando le due subunità ribosomiali, 40S e 60S, si sono unite in ribosomi @ Formazione di due siti: sito A e sito P Sito A (aminoacilico) Sede dell’attacco dell’aminoaciltRNA Sito P (peptidilico) Sede in cui si trova il peptidiltRNA, ovvero legato alla catena polipeptidica in crescita In questi siti avviene il riconoscimento dei codoni del mRNA ed il legame dell’anticodone del tRNA 2. Fase di Allungamento Caratterizzata da una serie di cicli di formazione del legame peptidico ed il numero dipende dalla quantità di aminoacidi che costituiranno la catena peptidica Ogni ciclo di allungamento prevede l’intervento di: - 1 aminoacil-tRNA - Fattori di allungamento - Idrolisi di 1 molecola di GTP come forma di energia 1° ciclo di allungamento tRNA si posizione nel sito A grazie ad un fattore di allungamento che lascia il ribosoma grazie all’idrolisi di una molecola di GTP Formazione del primo legame (formazione di un dipeptide) peptidico tRNA-Met Nel sito P tRNA-aa Nel sito A 2. Fase di Allungamento Nel sito A Avviene il legame tra le basi presenti sul codone dell’mRNA e le basi dell’anticodone del tRNA grazie al fattore di allungamento eIF-1 Consente il legame al ribosoma solo del tRNA che lega l’aa appropriato Sulla subunità minore ribosomiale Sito A e sito P interazione codone-anticodone Sulla subunità maggiore ribosomiale È associata all’estremità aminoacilica 2. Fase di Allungamento I siti A e P sono estremamente vicini per consentire la formazione del legame aminopeptidico tra due aa Peptidil-trasferasi Ribozima, Si forma un legame peptidico tra il gruppo –NH+ dell’aminoacido del sito A ed il gruppo –CO- dell’aa del sito P ovvero un RNA presente nella subunità maggiore con azione catalitica Il sito P avrà un tRNA-dipeptidico ed il sito A avrà legato solo il tRNA privo di aa 2. Fase di Allungamento TRASLOCAZIONE Fattore di allungamento eEF-2 GTP GDP+ Pi @ Il ribosoma unito si sposta di una tripletta (tre basi nucleotidiche) sull’mRNA in direzione 5’3’ @ Dal sito A si ha il distacco del tRNA privo di aminoacido e rientra nel citoplasma @ Dal sito A al sito P si ha la traslocazione del tRNA-dipeptidico 2. Fase di Allungamento NOTE I cicli della fase di allungamento si ripetono n volte fino a quando nel sito A del ribosoma non si posiziona uno dei tre codoni di stop UAG,UAA,UGA Durante la fase di allungamento o meglio durante la traslocazione se si utilizza un microscopio elettronico si osserva che le due subunità ribosomiali ruotano leggermente in direzioni opposte aumentando cosi lo spazio intrasubunità e consentendo al ribosoma stesso di scivolare lungo l’mRNA 3. Fase di Terminazione Sul sito A si lega un codone di stop presente lungo l’mRNA Nessun tRNA-aminoacilico complemetare nel sito A si legherà con il proprio anticodone In tal caso si legherà un fattore proteico di rilascio o anche fattore di terminazione: eRF-1 Attraverso la cristallografia a raggi X il fattore di terminazione proteico ha una struttura molto simile a quella di un tRNA 3. Fase di Terminazione Sito A + Alterazione dell’attività catalitica dell’enzima peptidil-trasferasi fattore di terminazione tRNA-H20 tRNA-aa Si ha cosi l’idrolisi del legame tra il polipeptide ed il tRNA agganciato al sito P La neo-proteina viene rilasciata nel citoplasma Il ribosoma si distacca dall’mRNA Le subunità ribosomiali si dissociano GTP GDP+ Pi Traduzione o Sintesi Proteica La velocità di sintesi proteica è regolata da un fattore di inizio iEF-2 nelle cellule eucarite Dove tale fattore è stato inattivato le cellule presentano una marcata riduzione della velocità di attivazione della sintesi proteica Durante la sintesi proteica le proteine si piegano su se stesse e prendono la struttura tridimensionale che le contraddistingue per la loro attività biologica specifica Traduzione o Sintesi Proteica La proteina di nuova sintesi è inattiva nel momento in cui si stacca dal ribosoma e prima di assumere un ruolo funzionale nella cellula deve subire uno o più modifiche post-traduzionali Le modifiche post-traduzionali più frequenti sono: @ Modifica o rimozione dell’aminoacido N-terminale @ Rimozione di corte sequenze aminoacidiche @ Formazione di legami o ponti disolfuro (S-S, cisteina-cisteina) @ Aggiunta di alcuni aminoacidi, di gruppi carbossilici, di gruppi fosforici, di gruppi metilici o di catene oligosaccaridiche laterali Delocalizzazione proteica post-traduzionale Smistamento delle proteine di nuova sintesi o anche detto sorting La cellula eucariota per delocalizzare le proteine di nuova sintesi utilizza sistemi molto sofisticati Costituiti da: segnali, recettori ed altri fattori come le proteine di accompagnamento (chaperoni) e segnali specifici presenti sulle neoproteine che ne determinano la destinazione 1. Le proteine destinate al citosol vengono semplicemente rilasciate dai ribosomi citoplasmatici grazie alla presenza di sequenze aminoacidiche specifiche che fungono da segnali di smistamento Delocalizzazione proteica post-traduzionale 2. Le proteine destinate ad essere inserite nella membrana plasmatica, all’interno dei lisosomi o quelle destinate ad essere escrete dalla cellula hanno i primi 15-30 aminoacidi (in prevalenza apolari) noti come peptide segnale Grazie alla presenza del peptide di segnale la sintesi proteica si arresta alla fine della traduzione dello stesso e può prosegurie soltanto dopo che il ribosoma si lega alla membrana del Reticolo Endoplasmatico (ER) Dal reticolo endoplasmatico le proteine glicosilate vengono trasportate verso l’apparato del Golgi etc. Distruzione delle proteine L’emivita di una proteina dipende dalla natura del residuo N-terminale Le proteine che devono essere degradate vengono fatte legare ad un’altra proteina presente nelle cellule eucariote che prende il nome di UBIQUITINA Attention Informazioni contenute nel DNA CODICE GENETICO PROTEINE 20 aa PROTEINOGENI 3 basi nucleotidiche dell’mRNA 1 aminoacido Es. UUU fenilalanina (Phe) Es. AUG codone di inizio codifica per metionina (Met) Attention Es. 6 triplette differenti codificano per Arginina (Arg) e per Leucina (Leu) Codici degenerato Questo fenomeno si verifica perché il numero di combinazioni per le triplette è numericamente superiore e per non avere triplette non senso a seguito di mutazioni puntiforme, ovvero modifiche di un solo nucleotide Mutazione silente = si ha la modifica di un nucleotide della tripletta ma si ha la sintesi di una proteina con modifica di un solo aminoacido Attention Modifiche di laboratorio iCAG= isocitosina nucleotide modificato sul filamento di mRNA (codone) iGUC= isoguanina (anticodone) nucleotide modificato sul filamento IODOTIROSINA= aa non naturale Possibile sintesi di nuove proteine Problemi etici di tRNA
Scaricare