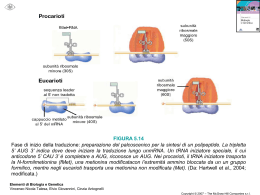
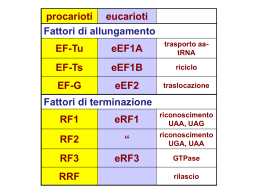
16 Il meccanismo della sintesi proteica BO ZZ A L’evento fondamentale della sintesi proteica è la formazione di un legame peptidico tra il gruppo amminico di un nuovo amminoacido e il gruppo carbossilico della catena peptidica nascente. Al momento della formazione del nuovo legame peptidico la catena peptidica nascente e il nuovo amminoacido si trovano ambedue legati a tRNA posizionati rispettivamente nel sito P e nel sito A del ribosoma (fig. 16.1). Il legame peptidico si forma senza previa rottura del legame tra l’amminoacido e il tRNA che la porta. Infatti nel centro peptil-transferasico del ribosoma le estremità dei due tRNA si trovano ravvicinate tra loro in modo che il gruppo amminico del nuovo amminoacido possa attaccare il gruppo carbossilico dell’ultimo amminoacido del peptide nascente che è legato al 3' dell’altro tRNA. Quindi il peptide nascente che si trovava legato a un tRNA viene trasferito su un altro tRNA con l’interposizione del nuovo amminoacido. Per questa ragione la reazione fondamentale della sintesi proteica viene chiamata reazione della peptidil-transferasi o peptidil-transferasica. Dopo il trasferimento, il peptide nascente viene a trovarsi legato al tRNA nel sito A, ma il successivo processo di traslocazione ripristina la situazione iniziale con il peptidil-tRNA nel sito P e il sito A libero per accogliere un nuovo aa-tRNA. Questa reazione si ripete ciclicamente per ogni amminoacido da aggiungere alla catena nascente durante la fase di allungamento della traduzione, mentre i processi che avvengono durante le fasi di inizio e di terminazione sono chiaramente diversi. Come illustrato in figura 16.2, la subunità minore è la prima che interagisce con l’mRNA per formare il compesso di inizio; solo con l’aggiunta della subunità maggiore la sintesi della proteina può effettivamente iniziare. Il ribosoma intero quindi scorre lungo la regione codificante dell’mRNA decodificando il messaggio e sintetizzando la catena peptidica, fino a quando incontra un codone di terminazione. Qui la proteina neosintetizzata viene rilasciata e il ribosoma si stacca dall’mRNA ritornando così nel pool cellulare di ribosomi liberi. Questi devono essere dissociati nelle due subunità, che possono così essere riutilizzate per successivi cicli di traduzione. Descriveremo ora in dettaglio gli eventi che caratterizzano l’inizio, l’allungamento e la terminazione della traduzione, nei procariotici e negli eucariotici. La maggior parte delle informazioni disponibili si riferiscono soprattutto a pochi organismi modello: E. coli per i procarioti, lievito e mammiferi per gli eucarioti. Come vedremo, la maggior differenza tra procarioti ed eucarioti la si riscontra nella fase di inizio della traduzione, forse perché questa è la fase dove maggiormente agiscono i meccanismi di regolazione della sintesi proteica, meccanismi di cui ovviamente gli eucarioti fanno massimo uso. Va precisato anche che per sintesi proteica eucariotica intendiamo quella soste- 000-000_C16_CS3.indd 1 2-03-2010 15:25:46 F 2 ISBN 978-88-08-18138-1 Traduzione e sue regolazioni nuta dal principale apparato di traduzione citoplasmatico. Ma non va trascurato che le cellule eucariotiche contengono anche apparati di traduzione minori, ma altrettanto importanti, localizzati negli organelli, quali i mitocondi di tutti gli eucarioti e i cloroplasti delle cellule vegetali. Non parleremo qui di questi apparati di traduzione particolari; sottolineiamo solo che per molti aspetti essi si avvicinano di più alla traduzione procariotica che a quella eucariotica principale. 16.1 - Inizio nei procarioti tRNA di inizio BO ZZ A Sia nei procarioti che negli eucarioti la sintesi di tutte le proteine inizia all’estremità ammino-terminale con l’amminoacido metionina. Questa è codificata quasi sempre dal codone AUG, ma come vedremo più avanti, in alcuni rari casi vengono usati anche i codoni GUG e UUG. Nei procarioti questa metionina di inizio è modificata chimicamente per aggiunta di un gruppo formilico che ne blocca il gruppo amminico, come mostrato in figura 16.3. La formilazione della metionina avviene a opera dell’enzima metionil-tRNA transformilasi, solo dopo essere stata caricata sul tRNA. Poiché durante il processo di sintesi proteica il gruppo amminico degli amminoacidi è essenziale per la formazione del legame peptidico con l’amminoacido precedente, la presenza del gruppo H N C R H C O H N C R H C O H Centro peptidil-transferasico N C R H C O H N C R H C O H2N H C R N C R C O H C O O O Sito P Sito A Figura 16.1 La reazione della peptidil-transferasi. Nel centro peptidil-transferasico del ribosoma, il gruppo amminico del nuovo amminoacido (rosso), legato al tRNA posizionato nel sito A, attacca 000-000_C16_CS3.indd 2 H OH Sito P O Sito A il gruppo carbossilico dell’ultimo amminoacido della catena peptidica nascente (nero) che si trova legato al tRNA posizionato nel sito P. 2-03-2010 15:25:46 ISBN 978-88-08-18138-1 CAPITOLO 16 • Il meccanismo della sintesi proteica 3 F formilico impedisce alla metionina modificata di essere utilizzata nella fase di allungamento, ma non impedisce che venga utilizzata come primo amminoacido della catena. D’altra parte residui di metionina sono presenti anche in posizioni interne alle sequenze proteiche. Infatti nella cellula ci sono due tipi di tRNA per la metionina: il tRNA di inizio tRNAiRMetf per la metionina che poi verrà formilata e il tRNAiMet per le metionine che saranno inserite internamente alle catene peptidiche. Quanto detto farebbe supporre che tutte le proteine cellulari abbiano all’estremità amminica una formil-metionina, ma sappiamo che ciò non è sempre vero. O meglio, ciò è vero solo al momento dell’inizio della sintesi della proteina ma successivamente, durante la fase di allungamento ZZ A Figura 16.2 Le subunità ribosomali vengono riciclate e riutilizzate per numerosi cicli di traduzione. Nella fase di inizio l’mRNA lega la subunità minore, a cui poi si unisce anche la subunità maggiore. Il ribosoma scorre sull’mRNA decodificando la sequenza nucleotidica e sintetizzando la proteina. Giunto al codone di terminazione il ribosoma rilascia la proteina completata, si dissocia dall’mRNA e rientra nel pool di ribosomi liberi. Questi devono essere dissociati nelle due subunità per potere essere riutilizzati per altri cicli di traduzione. Allungamento BO Inizio Met-tRNAifMet H2N CH3 CH3 S S CH2 CH2 Metionil-tRNA transformilasi CH2 CH C H O O O Figura 15.3 fMet-tRNAifMet (N-formil-metionil- tRNAifMet) si genera per formilazione di Met-tRNAifMet. La reazione è catalizzata 000-000_C16_CS3.indd 3 Terminazione C fMet-tRNAifMet CH2 NH CH C O O dalla metionil-tRNA transformilasi con formil-tetraidrofolato come cofattore. 2-03-2010 15:25:46 F 4 ISBN 978-88-08-18138-1 Traduzione e sue regolazioni o dopo il completamento della proteina, specifici eventi di maturazione posttraduzionale possono risultare, per alcune proteine, nella rimozione del gruppo formilico o anche di uno o più amminoacidi. Un’altra particolarità del tRNA di inizio è che mentre tutti gli altri amminoacil-tRNA entrano nel sito A del ribosoma per passare poi, una volta convertiti in peptidil-tRNA, nel sito P, il tRNA di inizio fMet-tRNAifMet entra direttamente nel sito P, anzi nell’emisito P, cioè nella regione della subunità 30S che andrà poi a formare il sito P nel ribosoma intero. Il riconoscimento del sito di inizio sull’mRNA BO ZZ A L’inizio della traduzione coinvolge sempre l’interazione preliminare tra la subunità minore del ribosoma, fMet-tRNAifMet, e mRNA. Questi componenti vanno a costituire il complesso d’inizio 30S; solo successivamente si ha il reclutamento della subunità maggiore del ribosoma e la formazione del complesso di inizio 70S che poi procede nell’allungamento. È necessario che il complesso di inizio si assembli sull’mRNA in corrispondenza del codone di inizio, che è in genere un codone AUG. Di triplette AUG però, in un qualsiasi mRNA ne esistono molte altre che non rappresentano codoni di inizio. Infatti, ci possono essere codoni AUG nella fase di lettura aperta (ORF) che porta l’informazione dell’mRNA e che codificano per metionine interne alla proteina sintetizzata; e ce ne possono essere anche nelle altre due fasi di lettura, quelle che non vengono lette come codoni durante la sintesi proteica. Come viene selezionato un vero codone AUG di inizio in corrispondenza del quale si deve assemblare il complesso di inizio? È stato possibile isolare e analizzare la sequenza della regione di mRNA su cui si assembla il complesso di inizio e che corrisponde al sito di legame al ribosoma. Infatti, come mostrato in figura 16.4A, facendo interagire in vitro ribosomi, mRNA, tRNA di inizio e GTP è stato possibile ottenere complessi di inizio che però restavano bloccati non potendo procedere nell’allungamento. Questi complessi venivano digeriti con endonucleasi che degradavano tutto l’mRNA tranne le regioni protette dai ribosomi; queste venivano poi riestratte e analizzate per la sequenza nucleotidica. Il confronto delle sequenze dei siti di legami al ribosoma di diversi mRNA così preparati ha messo in evidenza che in pochi nucleotidi a monte dell’AUG di inizio è presente una sequenza molto conservata. In particolare, come mostrato in figura 16.4B, la sequenza AGGAGG precede, 7 nt a monte, il codone di inizio AUG. Questa sequenza rappresenta il sito di legame dei ribosomi all’mRNA e viene comunemente chiamata “sequenza di Shine-Dalgarno”, dai nomi dei due ricercatori australiani John Shine e Lynn Dalgarno che l’hanno identificata. Questi ricercatori hanno anche dimostrato che essa è complementare all’estremità 3' dell’rRNA 16S che è un costituente della subunita 30S del ribosoma (fig. 16.4B). In realtà la sequenza di Shine-Dalgarno (AGGAGG) rappresenta un “consenso”, cioè è una sequenza media rispetto alla quale ciascun mRNA può in parte deviare. Infatti l’appaiamento anche di solo 4 o 5 basi con l’rRNA è sufficiente per l’interazione specifica tra mRNA e subunità ribosomale 30S e quindi per l’assemblaggio del complesso di inizio nella posizione corretta. Naturalmente solo i veri codoni AUG di inizio sono preceduti dalla sequenza Shine-Dalgarno. Gli altri AUG, per metionine interne o che si trovano nelle due fasi di lettura chiuse, non presentano Shine-Dalgarno e non sono utilizzate per iniziare la sintesi proteica. È da notare che un sito di inizio di questo tipo può trovarsi anche in posizioni interne lungo l’mRNA, cosicché una molecola di mRNA può avere anche più sequenze codificanti, ciascuna fornita di un suo sito di legame al ribosoma (Shine-Dalgarno+AUG) e di un suo codone di terminazione, come avviene negli mRNA policistronici dei procarioti. 000-000_C16_CS3.indd 4 2-03-2010 15:25:46 ISBN 978-88-08-18138-1 CAPITOLO 16 • Il meccanismo della sintesi proteica 5 F A AUG AUG AUG AUG A AUG mRNA B 5′ NNAGGAGGNNNNNNNAUGNN 3′ ZZ 3′ AUUCCUCCACUAG BO rRNA 16S Figura 16.4 Identificazione del “sito di legame per il ribosoma” (sequenza di Shine-Dalgarno) dell’mRNA procariotico. (A) Una preparazione di mRNA veniva fatta interagire con ribosomi in condizioni in cui questi restavano bloccati sul sito di inizio. Successivamente il trattamento con un’endonucleasi risultava nella degradazione di tutto l’mRNA tranne le brevi regioni protette dai ribosomi. Queste venivano recuperate e ne veniva analizzata la sequenza nucleotidica. 5′ (B) Il confronto delle sequenze nucleotidiche di vari di questi siti di legame per il ribosoma ha messo in evidenza la presenza 7 nucleotidi a monte del codone di inizio AUG, di una sequenza conservata il cui consenso è AGGAGG. Tale sequenza, chiamata sequenza di Shine-Dalgarno, è complementare all’estremità 3’ dell’rRNA 16S che fa parte della subunità piccola del ribosoma. I fattori di inizio nei procarioti Per il processo di inizio sono necessari, oltre all’mRNA, ai ribosomi e al tRNA di inizio caricato con la metionina, anche vari fattori proteici, che nei procarioti sono solo tre: IF1, IF2 e IF3 (IF, Initiation Factor). Tutti e tre i fattori d’inizio interagiscono con la subunità minore del ribosoma e sono necessari per la formazione del complesso di inizio 30S che include, oltre a una subunità ribosomale 30S, anche un fMet-tRNAifMet e un mRNA. • IF3 è coinvolto nella dissociazione dei ribosomi interi nelle due subunità e successivamente, legato alla subunità 30S, è essenziale perché questa si leghi in modo specifico al sito di inizio sull’mRNA. • IF1 contribuisce con IF3 alla dissociazione del ribosoma 70S e si lega alla subunità 30S nella regione che andrà a formare il sito A del ribosoma. • IF2 è una GTPasi (cioè lega e idrolizza GTP per svolgere il suo ruolo) che interagisce con la subunità 30S, promuove l’associazione con fMet-tRNAifMet all’emisito P e impedisce l’interazione con altri tRNA carichi. 000-000_C16_CS3.indd 5 2-03-2010 15:25:46 F 6 ISBN 978-88-08-18138-1 Traduzione e sue regolazioni Il processo di inizio della traduzione nei procarioti ZZ A IF1 IF3 BO Figura 16.5 Inizio della traduzione nei procarioti. (A) I fattori IF1 e IF3 legano la subunità 30S libera e impediscono che si riassoci alla 50S, mantenendola così disponibile per il legame all’mRNA; (B) La subunità 30S con IF3 si lega all’mRNA sul sito di inizio (riconosciuto mediante appaiamento di basi tra l’estremità 3’ dell’rRNA 16S e la sequenza di Shine-Dalgarno). In corrispondenza dell’emisito P si lega anche IF2-GTP, a cui poi si unisce il tRNA iniziatore fMet-tRNAifMet, completando così la formazione del cosiddetto complesso di inizio 30S; (C) Il rilascio dei fattori IF1 e IF3 permette il legame della subunità 50S; il successivo rilascio anche di IF2 con idrolisi del GTP, porta al complesso di inizio 70S. A La sequenza degli eventi descritta in figura 16.5 è uno dei possibili modelli. Infatti c’è ancora qualche incertezza sull’ordine temporale delle interazioni tra i vari componenti. Poiché dopo ogni ciclo di sintesi proteica i ribosomi vengono rilasciati in forma di ribosomi interi 70S, è necessario che questi vengano dissociati nelle due subunità affinché queste possano essere ri-utilizzate in un altro ciclo di sintesi. I ribosomi 70S tendono a dissociarsi fino a che viene raggiunto un equilibrio dinamico tra ribosomi interi e subunità e sicuramente i fattori IF1 e IF3 sono coinvolti in questa dissociazione, anche se non c’è ancora un’opinione concorde su alcuni dettagli della reazione. Il modello mostrato in figura 16.5A implica un primo intervento di IF1, che legandosi alla 30S nel ribosoma intero contribuisce alla dissociazione delle due GTP IF2 B fMet-tRNAiMet mRNA Complesso di inizio 30S C GDP Complesso di inizio 70S 000-000_C16_CS3.indd 6 2-03-2010 15:25:46 ISBN 978-88-08-18138-1 CAPITOLO 16 • Il meccanismo della sintesi proteica 7 F BO ZZ A subunità. IF3, invece, interagisce solo con la subunità già dissociata e, impedendone la riassociazione con la subunità 50S, la sottrae all’equilibrio e la mantiene disponibile per un nuovo inizio. Dopo che IF1 e IF3 hanno portato a termine la dissociazione dei ribosomi nelle due subunità, la 30S può essere utilizzata per la formazione di un nuovo complesso d’inizio 30S che comprenderà, oltre alla subunità stessa, i tre fattori e un tRNA di inizio (alcuni chiamano complesso di inizio il passo successivo che comprende anche l’associazione all’mRNA). Come mostrato in figura 16.13B, IF2·GTP va a legarsi alla subunità 30S in prossimità dell’emisito P, dove promuove l’ingresso del tRNA di inizio fMet-tRNAifMet. Questo è uno dei passaggi che presenta qualche incertezza; infatti, mentre in genere si pensa che IF2 interagisca prima con la subunità ribosomale per poi reclutare fMet-tRNAifMet, altri suggeriscono che IF2 si leghi prima a fMet-tRNAifMet e poi lo trasporti nel sito P, analogamente a quello che succede durante l’allungamento dove il fattore EF-Tu lega un amminoacil-tRNA e lo porta nel sito A (vedi oltre). È interessante notare che il tRNA di inizio, fMet-tRNAifMet, è l’unico tRNA che entra nel ribosoma senza passare attraverso il sito A, come invece fanno tutti gli altri tRNA portati dal fattore di allungamento EF-Tu. Comunque, contemporaneamente o subito dopo il posizionamento di fMet-tRNAifMet nell’emisito P si ha il legame, mediato da IF3, dell’mRNA alla subunità 30S. Come detto precedentemente, questa interazione è facilitata e resa specifica dall’appaiamento di basi complementari tra la sequenza di Shine-Dalgarno e l’estremità 3' dell’rRNA 16S nella subunità piccola del ribosoma e tra il codone AUG e l’anticodone del tRNA di inizio fMet-tRNA già posizionato nell’emisito Pi. È da notare che nel complesso di inizio 30S la presenza di IF1 e IF2, rispettivamente nelle adiacenze degli emisiti A e P, impedisce l’interazione della subunità ribosomale con qualsiasi altro tRNA carico tranne l’fMet-tRNAifMet, specificamente posizionato da IF2. Un’altra interessante osservazione è che se si va a saggiare l’interazione dei tre fattori uno alla volta, individualmente, con la subunità 30S, si trova che si formano legami poco stabili, mentre l’interazione si fa molto più stabile se si fa interagire la 30S contemporaneamente con i tre fattori. Il passo successivo è l’associazione del complesso di inizio 30S con la subunità ribosomale 60S, che risulta nella formazione del complesso di inizio 70S. Come mostrato in figura 4C, perché ciò avvenga è necessario dapprima il rilascio dal complesso 30S dei fattori IF1 e IF3 che permette l’assemblaggio della subunità grande e successivamente il rilascio di IF2, che dipende dall’idrolisi del GTP a GDP. Il complesso d’inizio 70S, costituito da un ribosoma intero 70S, un mRNA e un fMet-tRNAifMet, è pronto per entrare nella fase di allungamento della traduzione. 16.2 - Inizio negli eucarioti Come nei procarioti, anche negli eucarioti la traduzione inizia con la formazione di un complesso tra la subunità ribosomale minore, l’mRNA e il tRNA di inizio, che vanno a costituire il cosiddetto complesso di pre-inizio 43S. Tuttavia il meccanismo che porta all’assemblaggio del complesso è sostanzialmente diverso da quello che abbiamo visto per i procarioti. Di fatto, di tutto il processo di sintesi proteica l’inizio è quello che differisce di più tra procarioti ed eucarioti. Innanzitutto anche se, come nei procarioti, negli eucarioti abbiamo una metionina di inizio, questa non è formilata nel suo gruppo amminico. Esistono comunque due tRNA per la metionina, uno utilizzato per l’inizio, chiamato tRNAiMet, e uno per la fase di allungamento, tRNAMet. Un’altra differenza sostanziale tra procarioti ed eucarioti, schematizzata in Figura 15.6A e B, è il meccanismo di interazione tra mRNA e subunità piccola del ribosoma che porta alla formazione del complesso di inizio. Nei procarioti, 000-000_C16_CS3.indd 7 2-03-2010 15:25:46 F ISBN 978-88-08-18138-1 Traduzione e sue regolazioni 8 come abbiamo visto sopra e come schematizzato in figura 16.6A, la presenza della sequenza di Shine-Dalgarno permette un inizio della traduzione “interno” all’mRNA, e quindi anche la possibilità di mRNA policistronici. Negli eucarioti invece la subunità minore del ribosoma interagisce inizialmente con l’estremità 5' dell’mRNA e da lì migra, mediante un processo di “scansione” (scanning), lungo il 5'UTR fino a trovare il primo AUG, che verrà utilizzato come codone di inizio della traduzione (fig. 16.6B). È chiaro che questo meccanismo di inizio è incompatibile con la presenza di più sequenze codificanti in una singola molecola di mRNA; infatti la grande maggioranza degli mRNA eucariotici è monocistronica, cioè ogni mRNA codifica una sola proteina. Fattori di inizio negli eucarioti A BO cap ZZ A Mentre, come abbiamo visto sopra, per i procarioti sono necessari tre fattori, ciascuno costituito da una singola catena peptidica, negli eucarioti i fattori sono più del doppio e, soprattutto, alcuni di essi sono molto grandi e costituiti da numerose subunità, per esempio 12 subunità per il fattore eIF3, per cui in totale sono necessari oltre 30 diversi polipeptidi per l’inizio della traduzione. I fattori d’inizio della traduzione eucariotica vengono indicati con la sigla eIF (eukaryotic Initiation Factor) cui seguono numeri e lettere di specificazione. Per dare un’idea della complessità del sistema, la figura 16.7 riporta l’elenco dei fattori di inizio della traduzione nei mammiferi (H, sapiens), anche se qui ci limiteremo a considerare solo alcuni dei fattori principali. SD AUG B cap AUG Scansione C cap IRES AUG Figura 16.6 Confronto schematico dei meccanismi di inizio della traduzione. (A) Nei procarioti si ha un inizio interno dipendente dalla presenza della sequenza di Shine-Dalgarno (SD) poco a monte dell’AUG di inizio. (B) Negli eucarioti in genere la subunità piccola lega il cap all’estre- 000-000_C16_CS3.indd 8 mità 5’ dell’mRNA e poi procede nella scansione del 3’UTR fino a trovare l’AUG di inizio. (C) Per alcuni virus e mRNA eucariotici si ha un meccanismo di inizio cap-indipendente in cui la subunità piccola lega una sequenza IRES interna e da lì procede per scansione fino all’AUG di inizio. 2-03-2010 15:25:46 ISBN 978-88-08-18138-1 CAPITOLO 16 • Il meccanismo della sintesi proteica 9 F Figura 16.7 Fattori di inizio per la traduzione cap-dipendente degli eucarioti. Fattore Ruolo eIF1 Lega la subunità 40S, controlla l'accuratezza del riconoscimento del codone AUG di inizio e contribuisce con eIF3 a impedire l'associazione prematura con la 60S eiF1A Promuove il legame di Met-tRNAiMet alla subunità 40S e contribuisce con eIF3 alla interazione forte di questa con l'mRNA eIF2 (3 subunità) Interagisce con Met-tRNAiMet formando il complesso ternario “Met-tRNAiMet/eIF2/GTP” eIF2B (5 subunità) Catalizza lo scambio GTP/GDP per il riciclo di eIF-2 (è chiamato anche GEF = Guanine-nucleotide Exchange Factor) eIF3 (12 subunità) Lega la subunità 40S e ne promuove il legame con Met-tRNAiMet e mRNA e impedisce l’associazione della 60S prima che si sia formato il complesso di inizio 43S eIF4E Riconosce e lega il 5’cap eIF4F Costituisce un’impalcatura per l'assemblaggio di eIF4F, Interagendo con eIF4E ed eIF4A eIF4G Ha attività RNA elicasica ATPasi-dipendente eIF4 Stimola l’elicasi e lega simultaneamente eIF4F eIF4A eIF4B Interagisce con il complesso di pre-inizio 43S (40S/Met-tRNAiMet/eIF3/eIF2-GTP) e promuove l'idrolisi del GTP associato a eIF2 eIF5B Legato a GTP recluta il complesso ternario “Met-tRNAiMet/eIF2/GTP” sulla subunità 40S eIF6 Lega la subunità 60S libera e impedisce la sua associazione con la 40S ZZ A eIF5 BO Processo di inizio della traduzione negli eucarioti La figura 16.8 mostra schematicamente la sequenza degli eventi che porta all’assemblaggio del complesso di inizio negli eucarioti, dove la situazione è notevolmente più complicata che nei procarioti e, anche qui, con notevoli incertezze e discordanze sull’ordine temporale di alcune interazioni. La figura 16.8A mostra la formazione del cosiddetto complesso di pre-inizio 43S. I fattori eIF3 e eIF1A svolgono le stesse funzioni, ancorché con alcune differenze, di IF3 e IF1 procariotici, cioè contribuiscono alla dissociazione del ribosoma intero 80S nelle due subunità legando la subunità piccola 40S. Il reclutamento del tRNA di inizio sulla 40S è mediato da due proteine, ambedue GTPasi: eIF2·GTP e eIF5B·GTP. Il primo interagisce con il tRNA di inzio Met-tRNAiMet formando un complesso ternario (Met-tRNAiMet/eIF2/GTP) il cui reclutamento sulla 40S necessita di eIF5B·GTP, che è il vero analogo del IF2-GTP procariotico. Si ha così la formazione del complesso di pre-inizio 43S che comprende la subunità 40S, il Met-tRNAiMet e i fattori fino a qui riportati. Nel frattempo e indipendentemente si forma un complesso tra l’mRNA e il fattore eIF4 (fig. 16.8B). Questo è composto dalle subunità eIF4A, eIF4B, eIF4E, eIF4G. Quest’ultima è una grande proteina (220 kDa) che costituisce un’impalcatura su cui si monta il complesso. Come mostrato in figura, essa è legata a eIF4E e a eIF4A insieme alle quali costituisce l’intermedio che prende il nome di eIF4F. Questo interagisce con l’mRNA mediante un’interazione specifica tra la subunità eIF4E e il cap (cappuccio) che si trova legato al primo nucleotide di tutti gli mRNA eucariotici. Infatti eIF4E viene anche chiamata “proteina che lega il cap” (cap binding protein). Al complesso si aggiunge poi la subunità eIF4B. Successivamente, l’interazione tra il complesso di pre-inizio 43S e il complesso eIF4F/mRNA risulta nella formazione del complesso di inizio 48S in cui l’estre- 000-000_C16_CS3.indd 9 2-03-2010 15:25:47 F ISBN 978-88-08-18138-1 Traduzione e sue regolazioni 10 B A elF6 cap AUG elF1A elF3 mRNA elF4F elF4A elF4E elF4G GTP elF5B GTP elF2 elF4B A Met-tRNAiMet BO Complesso di inizio 48S ZZ Complesso di pre-inizio 43S C Complesso di inizio 80S Figura 16.8 Inizio della traduzione cap-dipendente negli eucarioti. (A) Formazione del complesso di pre-inizio 43S costituito dalla subunità piccola del ribosoma complessata con i fattori eIF1, eIF3, eIF5-GTP e il complesso ternario Met-tRNAiMet /eIF2/GTP). (B) Formazione del complesso tra mRNA e il fattore eIF4. (C) L’interazione tra il complesso di pre-inizio 43S e il comples- 000-000_C16_CS3.indd 10 so eIF4/mRNA risulta nella formazione del complesso di inizio 48S in cui l’estremità 5’ dell’mRNA è legata alla subunità ribosomale 40S. Successivamente, il processo di scansione porta la subunità ribosomale 40S con i fattori associati fi no al codone AUG di inizio. L’unione della subunità ribosomale 60S e il rilascio di tutti i fattori portano alla formazione del complesso di inizio 80S. 2-03-2010 15:25:47
Scaricare