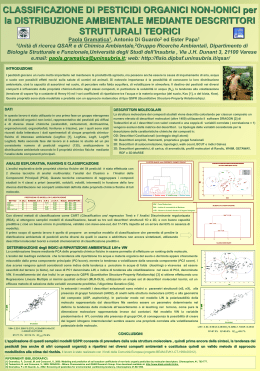
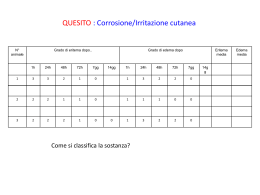
MODELLAMENTO QSAR E PREDIZIONE DELLA TOSSICITA’ DI FENOLI Francesca Battaini, Ester Papa e Paola Gramatica Unità di ricerca QSAR e di Chimica Ambientale, Dipartimento di Biologia Strutturale e Funzionale, Università degli Studi dell’Insubria (Varese) Web: http://fisio.dipbsf.uninsubria.it/qsar/ e-mail: [email protected] INTRODUZIONE I fenoli sono composti chimici in grado di persistere nei diversi comparti ambientali e di penetrare facilmente negli organismi espletando la loro azione tossica. Ampiamente utilizzati anche come precursori di molti prodotti, fanno parte degli HPV (High Production Volume Chemicals), sostanze prodotte in quantità superiore alle 1000 tonnellate/anno, per le quali, come richiesto dal recente White Paper dell’Unione Europea [1], è necessario conoscere proprietà chimico-fisiche e tossicità entro il 2005. La loro tossicità è stata oggetto di ampi studi su diversi end-point, ma evidentemente non sono disponibili dati sperimentali per tutti i fenoli e per tutti gli organismi. Il lavoro svolto ha lo scopo di produrre modelli che predicono in modo quantitativo, basandosi sulla struttura delle molecole (QSAR-Quantitative Structure-Activity Relationship), la tossicità di questi composti allo scopo diminuire il lavoro sperimentale necessario per l’ottenimento di tali dati. DESCRITTORI MOLECOLARI DATI La struttura molecolare dei composti utilizzati è stata rappresentata usando diversi descrittori molecolari calcolati con il software DRAGON di R.Todeschini [2]. L’ampio set di descrittori comprende: In questo lavoro sono stati utilizzati i dati di tossicità misurati in Tetraymena pyriformis, protozoo ciliato, per 98 fenoli, variamente sostituiti, ottenuti dalla letteratura [3]. I valori sono espressi in mM/l e in scala logaritmica come il log dell’inverso della IGC50(concentrazione che inibisce la crescita nel 50% della popolazione). descrittori 0D – descrittori costituzionali descrittori 1D – ricavati dalla formula bruta della molecola (descrittori empirici, gruppi funzionali, proprietà e frammenti) descrittori 2D – determinati dal grafo molecolare (descrittori topologici, di autocorrelazione, BCUTs ed indici di Galvez) descrittori 3D – derivati dalle coordinate spaziali (descrittori geometrici, di carica, di aromaticità, WHIMs, GETAWAY, RDF e 3D-MoRSE). Istogramma relativo alla disrtibuzione delle risposte Mappa di Kohonen 10x10 15 14 A questi descrittori è stato aggiunto il coefficiente di ripartizione ottanolo/acqua (Log Kow) che fornisce una misura della lipofilicità delle molecole. Training 13 Test Numero di osservazioni 12 METODI CHEMIOMETRICI Sono state applicati diversi metodi chemiometrici per selezionare un training set ottimale per i 11 SELEZIONE DEL TRAINING TEST 10 9 8 7 6 5 4 3 2 1 modelli QSAR. Queste tecniche sono: 0 <= -1 (-1;-.5] (-.5;0] (0;.5] (.5;1] (1;1.5] (1.5;2] >2 l’Analisi delle Componenti Principali (PCA): questa analisi permette di concentrare l’informazione di un grande numero di K o h o n e nm a po fm o le c u la rs tru c tu re variabili in un numero esiguo di componenti tra loro ortogonali e di identificare gli oggetti che fanno parte di classi e gli 10x10 1 outliers. Le componenti più significative sono state utilizzate nella Cluster Analysis e nelle Mappe di Kohonen per eliminare 1 l’informazione ridondante. 3 la Cluster Analysis: in questo lavoro è stata usata un’analisi di tipo agglomerativo dove le variabili sono rappresentate 5 dalle componenti significative dei descrittori molecolari. Per trovare il miglior modo di raggruppare i composti sono state 7 usate due formule di distanza (Euclidea e di Manhattan) e diversi metodi per determinare la similarità tra i nuovi cluster 9 2 81 83 94 6 48 44 45 87 100 101 50 73 71 72 5 63 726 49 91 97 84 95 47 7 15 35 65 12 32 64 59 52 107 93 99 88 86 90 11 31 6 33 8 885 (Complete, average, ecc.). 105 55 1 0 89 92 6103 57 58 60 104 153 54 9 14 34 62 61 25 46 102 8 1 0 4 45 43 24 23 2 4 3 28 29 80 96 98 21 106 20 75 77 70 56 66 30 36 39 910 17 37 67 38 68 69 13 16 19 74 342 40 41 79 78 222 82 2 3 2 2 2 2 te stse t tra in in gse t le Mappe di Kohonen: le reti neurali artificiali sono un altro modo di raggruppare gli oggetti simili in cluster usando “self organised topological feature maps”, con questa analisi l’informazione multidimensionale viene visualizzata in un grafico bidimensionale. La posizione dei composti nelle celle della mappa mostra il livello di similarità tra le strutture dei fenoli presi in esame. La selezione del training set è stata effettuata mediante la selezione per ogni cella dell’oggetto più caratteristico cioè quello più vicino al centroide. Leave-one-out- viene escluso dal training un oggetto alla volta e predetto Leave-more-out- viene escluso casualmente il 50% delle molecole del training L o g 1 /IG C 5 0 = -1 .2 5 + 0 .5 L o g k o w + 0 .3 C -X Y-scrambling- ottenuta mediante una permutazione casuale delle risposte L o g 1 /IG C 5 0 = -2 .3 3 + 0 .5 L o g K o w + 0 .3 7 U i+ 0 .3 3 C -X 2 .1 2 .1 1 .5 1 .5 0 .9 0 .9 rispostacalcolata La selezione delle variabili più correlate alla risposta e più predittive è stata realizzata con il metodo dei minimi quadrati ordinari (OLS) e con la tecnica dell’Algoritmo Genetico (GA-VSS). I modelli sono stati validati con diversi metodi: MODELLI DI REGRESSIONE LINEARE OTTENUTI CON UN TRAINING SET DI 55 OGGETTI (test set di 43 oggetti) rispostacalcolata MODELLI DI REGRESSIONE - MLR 0 .3 -0 .3 -0 .9 0 .3 -0 .3 -0 .9 tra in in gs e t te s ts e t -1 .5 -1 .5 -1 .0 -0 .5 0 .0 0 .5 1 .0 r is p o s tas p e r im e n ta le 1 .5 2 .0 2 .5 tra in in gs e t te s ts e t -1 .5 -1 .5 -1 .0 -0 .5 0 .0 0 .5 1 .0 1 .5 2 .0 2 .5 r is p o s tas p e r im e n ta le Validazione esterna- verificata su un test esterno di validazione N. ogg. N. Var. Descrittori 98 3 C-X Ui 98 2 C-X Log Kow 98 1 Log Kow 55 3 C-X 55 2 55 1 2 Q 2 2 LMO (50%) Q ext R 2 SDEP SDEC 87.0 86.5 88.0 0.27 0.26 84.7 84.5 85.6 0.29 0.28 76.0 75.9 77.2 0.36 0.35 86.9 85.0 86.5 88.4 0.28 0.27 C-X Log Kow 85.2 84.5 83.2 86.8 0.30 0.28 Log Kow 74.6 73.9 77.7 76.7 0.40 0.38 Ui Log Kow Q Log Kow I descrittori più importanti risultano essere il Log Kow che è legato alla lipofilia della molecola e, quindi, alla capacità di quest’ultima di attraversare le membrane biologiche determinando una tossicità di base; Ui che indica il livello di insaturazione e C-X che indica il numero di frammenti contenenti alogeni. Fattori di polarità che risultano quindi essere importanti nel rappresentare la tossicità potenziale dei fenoli. CONCLUSIONI I modelli ottenuti, riassunti in tabella, hanno buone capacità predittive; il coefficiente di ripartizione ottanolo/acqua, ampiamente utilizzato per modellare la tossicità di composti chimici, si dimostra un buon descrittore, ma può essere validamente affiancato da altri descrittori molecolari teorici, calcolabili quindi anche per molecole non ancora sintetizzate, dando luogo a modelli altamente predittivi. Il lavoro è stato realizzato con fondi della Comunità Europea (progetto BEAM). Si ringrazia Federchimica per la borsa di studio a Francesca Battaini (progetto H.E.L.E.N). RIFERIMENTI BIBLIOGRAFICI [1] White Paper on the strategy for a future Chemicals Policy. www.europa.eu.int/comm/environmental/chemicals/whitepaper.htm [2] Todeschini, R., Consonni, V. e Pavan, E. 2001. DRAGON – Software for the calculation of molecular descriptors, rel. 1.12 for Windows [3] Schultz,T.W. et all. Quantitative structure-activity relationships for the Tetrahymena piryformis population growth end-point: a mechanism of action approach. Practical Application of QSAR in Environmental chemistry and toxicology, 241-262 (1990)
Scaricare