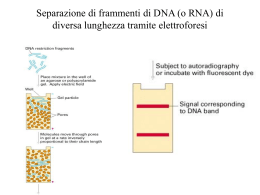
Ibridazione degli acidi nucleici e misurazione dell’espressione di un gene Condizioni che possono destabilizzare la doppia elica provocando la separazione delle due catene (denaturazione) 1. 2. 3. 4. Alte temperature pH alcalino estremo (>13) Bassa forza ionica [Na+] Presenza in soluzione di sostanze che rompono i ponti a idrogeno (urea, formamide). La denaturazione della doppia elica si accompagna a grosse variazioni delle proprietà fisiche delle soluzioni di DNA 1. Diminuzione della viscosità 2. Aumento di assorbanza a 260nm 3. Variazione dell’attività ottica Curve di denaturazione La denaturazione non è un processo irreversibile. Infatti se dopo la separazione delle eliche si fa scendere gradualmente la temperatura, le singole eliche complementari si possono riappaiarea doppia elica può riformarsi. Questo processo si chiama ibridazione. La discesa graduale della temperatura e la permanenza delle molecole per un certo tempo pochi gradi al di sotto della temperatura di denaturazione sono fondamentali affinchè ci possa essere ibridazione, processo dipendente dai moti di agitazione termica. Se dopo la denaturazione la soluzione viene portata a bassa temperatura non si ha ibridazione, ma stabilizzazione della struttura secondaria delle singole catene. Per mezzo dell’ibridazione si possono formare delle doppie eliche di DNA-DNA, DNA-RNA o RNA-RNA. La condizione fondamentale perché ciò avvenga è che in soluzione si mettano molecole con sequenza complementare (antiparallele). L’ibridazione è una forma di riconoscimento molecolare estremamente specifica. In condizioni opportune di temperatura e forza ionica (stringenza) si possono ottenere anche delle eliche in cui sono tollerati degli appaiamenti non perfetti Effetti della stringenza di ibridazione Cinetiche di rinaturazione Sintesi chimica oligonucleotidi: Con questo sistema si possono sintetizzare in vitro singoli filamenti di DNA di grandezza compresa tra 6 e 100 nucleotidi Metodi di marcatura degli acidi nucleici Le DNA polimerasi batteriche possono essere purificate e utilizzate per sintetizzare DNA in vitro dATP dCTP dGTP dTTP Primer sintetico 3’ 5’ 5’ 3’ DNA stampo (singolo filamento) DNA polimerasi Le DNA polimerasi batteriche possono essere purificate e utilizzate per sintetizzare DNA in vitro DNA polimerasi 5’ 5’ 3’ DNA stampo (singolo filamento) Le DNA polimerasi batteriche possono essere purificate e utilizzate per sintetizzare DNA in vitro 3’ 5’ 5’ 3’ Traccianti utilizzati Nucleotidi marcati con isotopi radioattivi Traccianti utilizzati Nucleotidi marcati con sostanze non radioattive Traccianti utilizzati Nucleotidi marcati con sostanze non radioattive - Fluorocromi (marcatura diretta) - Enzimi (perossidasi, fosfatasi alcalina) - Digossigenina (riconosciuta da anticorpi specifici marcati con fluorocromi o enzimi) - Biotina (riconosciuta da avidina marcata con fluorocromi o enzimi) Traccianti utilizzati Fluorocromi Marcatura di DNA mediante random priming Esempio di ibridazione: FISH (fluorescent in situ hybridization) Come si fa a vedere se un gene è espresso? Purificazione dell’mRNA (non obbligatoria) • L’mRNA rappresenta il 2-4 % dell’RNA totale presente nelle cellule. • L’mRNA può essere purificato mediante cromatografia di affinità con oligo-dT legata a diversi supporti solidi. • L’mRNA purificato prende anche il nome di poly-A+, e rappresenta la base per diverse procedure di analisi di espressione, oltre che per la sintesi del cDNA necessario alla produzione di genoteche. Northern Blotting - 28S 18S 7S + • L’RNA (totale o poly-A+) viene frazionato mediante corsa elettroforetica su gel di agarosio deneturante (il gel contiene formaldeide, e preima della corsa i campioni vengono denaturati in formamide). Questo è necessario per far sì che le molecole di RNA si separino in base al loro peso molecolare, e non in base alla forma. Dopo la corsa si procede come per il Southern blot. Cella elettroforetica per gel di agarosio Northern Blotting Northern Blotting: e dopo il trasferimento? • Saturazione dei siti a cui la sonda potrebbe legarsi in modo non-specifico (macromolecole di vario genere, in particolare si usano proteine e DNA a singolo filamento non marcato) • Ibridazione con sonda marcata • Lavaggi per eliminare la sonda legata debolmente al filtro e non ibridata in modo corretto • Visualizzazione del segnale (ad es. se la marcatura è radioattiva si fa una autoradiografia) Caratteristiche del Northern Blotting • Tecnica non particolarmente sensibile. La sensibilità può essere aumentata notevolmente se invece dell’RNA totale si usa l’RNA polyA+. Infatti il quantitativo di RNA che si può caricare su un gel di agarosio è 50 mg. Se invece di caricare RNA totale carico un uguale quantitativo di poly-A+, posso avere un segnale fino a 50 volte maggiore. • L’uso di mRNA purificato elimina anche la possibilità di ibridazione non-specifica con l’rRNA. • Può essere applicata alla misurazione dell’espressione di un singolo gene • Oltre a misurare i livelli di espressione permette di valutare il peso molecolare dei trascritti, e consente di caratterizzare eventuali isoforme alternative . • Questa tecnica richiede che l’RNA sia il più integro possibile. Anche solo una rottura per molecola di mRNA determina una forte riduzione del segnale specifico. Esempio di Northern Blotting su RNA da tessuti A BCD E mRNA F AAAAAAA Sonda1 Sonda 2 Sonda1 B L K H Sonda2 M B L K H M Ibridazione in situ • Questa metodica si basa sull’ibridazione di sonde marcate direttamente su cellule o tessuti. Si può ibridare su sezioni istologiche o su materiale non sezionato (in quest’ultimo caso si parla whole-mount). • E’ l’unica tecnica che permette di localizzare l’espressione di un dato mRNA a livello delle singole cellule. • Ideale per studiare i geni espressi in un gruppo ristretto di cellule all’interno di un tessuto, o i geni la cui espressione è modulata nel tempo e nello spazio (estremamente utilizzata negli studi di biologia dello sviluppo). Ibridazione in situ Esempi Ibridazione in situ ‘whole mount’ Esempi Misurazione espressione a livello della proteina Western Blotting SDS-PAGE Misurazione espressione a livello della proteina Western Blotting Electroblotting Misurazione espressione a livello della proteina Western Blotting Immunodetection Misurazione espressione a livello della proteina Immunofluorescenza ed immunoistochimica Permettono di valutare in quali cellule e in quali strutture subcellulari sono localizzate le proteine Saggi di ibridazione inversa Sia i macroarrays che i microarrays sono stati sviluppati per soddisfare l’esigenza di misurare contemporaneamente l’espressione di più geni. Entambe le tecnologie si basano sullo stesso principio: 1. Come sonda si usano olgonucleotidi o molecole di cDNA non marcati, immmobilizzati in posizioni precise di un supporto solido A B C D 1 a b g d 2 e z h q 3 i k l m 4 n x o Array p 2. L’array viene ibridizzato con una miscela complessa di molecole marcate rappresentative dell’mRNA espresso dalle cellule in esame mRNA RT Nucleotidi marcati cDNA Macrorray • Le molecole sonda vengono legate a membrane di nylon • Come tracciante viene utilizzata la radioattività • Analisi di qualche decina o poche centinaia di geni Microrray di cDNA (o di oligonucleotidi lunghi) • Le molecole sonda sono cDNA o oligonucleotidi lunghi 70-80 paia di basi, sintetizzati tradizionalmente e egati ad un vetrino da microscopio per mezzo di un processo di stampa a getto. • Su ogni vetrino trovano posto fino a 10000 geni. • Metodica che si usa per comparare le differenze di espressione genica tra due campioni. cDNA Microarray mRNA Reverse transcription Labeled 1st strand cDNA Sample 1 (test) Sample 2 (reference) Hybridization on the cDNA microarray Generazione di una enorme quantità di dati. L’acquisizione dei dati è solo la parte iniziale della procedura. La parte più complicata è l’elaborazione della enorme quantità di dati generati da questi esperimenti, necessaria per rispondere ai quesiti biologici di partenza. I dati più significativi devono essere poi verificati con altri sistemi (northern, real time RT-PCR) Applicazioni Definizione delle basi molecolari e identificazione di nuovi markers prognostici per neoplasie e altre patologie Applicazioni Farmacogenomica e tossicogenomica Misurazione espressione a livello della proteina 2D-Gel electrophoresis
Scaricare