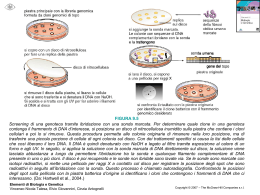
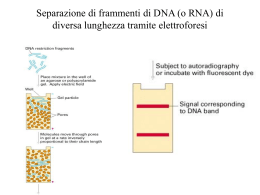
Clonaggio: vettori plasmidici I plasmidi sono molecole di DNA extra-cromosomali che si auto-replicano Sono presenti in natura in batteri, lieviti e alcuni eucarioti superiori, in uno stato di relazione parassitica o simbiontica verso la cellula ospite Molti plasmidi contengono caratteri (geni) che arrecano beneficio alla cellula ospite (es. resistenza a un antibiotico) Molti plasmidi sono in grado di auto-trasferirsi da una cellula all’altra. • VETTORI PLASMIDICI • • • • • • -> 0-10 kb VETTORI l di inserzione -> 0-10 kb VETTORI l di sostituzione -> 9-23 kb VETTORI COSMIDICI -> 30-44 kb VETTORI PAC (crom. artif. P1) -> 130-150 kb VETTORI BAC (crom. artif. batt.)-> fino a 300 kb VETTORI YAC (crom. artif.lievito-> 0.2-2 Mb Il batteriofago l Le genoteche o librerie di DNA: –Libreria genomica: collezione di cloni che include tutto il DNA genomico di una certa specie (es. il genoma umano aploide contiene circa 3x109 coppie di basi, che possono essere contenute in circa 1.5x105 cloni di 20 kb ciascuno) –Libreria di cDNA: collezione di cloni che include tutte le specie di mRNA (trascritte in cDNA) espresse in un dato tessuto, incluso quelle piu’ rare Costruzione di una libreria genomica Costruzione di librerie genomiche utilizzando il fago l come vettore ---GGATCC-----CCTAGG--- ---GATC-----CTAG--- ---G GATCC-----CCTAG G--- BamH1 + BamH1 Sau3A ---G GATCC-----CCTAG G--- --GATC-----CTAG --- --GATC-----CTAG --- Sau3A ---GATC-----CTAG--- Sau3A Costruzione di una libreria di cDNA Analisi dei cloni ottenuti • Crescita dei cloni, purificazione del DNA plasmidico o fagico e analisi con enzimi di restrizione • Sequenziamento • sottoclonaggio Separazione di frammenti di DNA (o RNA) di diversa lunghezza tramite elettroforesi Visualizzazione dei frammenti separati per elettroforesi MAPPE DI RESTRIZIONE Mappare multipli siti di restrizione Separazione di frammenti di DNA (o RNA) di diversa lunghezza tramite elettroforesi IBRIDAZIONE DI ACIDI NUCLEICI • La messa a contatto risultante nell’appaiamento di frammenti a singolo filamento provenienti da due fonti diverse: una sonda (popolazione omogenea di molecole note marcate) e un bersaglio (complessa ed eterogenea popolazione di molecole di acido nucleico). • Usata per determinare la presenza o la quantita’ di acidi nucleici con una sequenza specifica corrispondente a quella della sonda nel campione biologico analizzato. • Le condizioni a cui si effettua la reazione di ibridazione determinano il grado di divergenza delle sequenze analizzate (stringenza). STRINGENZA • La Tm (temperatura di fusione o melting T) caratterizza la stabilita’ dell’ibrido che si forma tra la sonda e il suo filamento complementare. • La Tm e’ critica per determinare la temperatura ottimale per usare sonde oligonucleotidiche come sonde per ibridizzare o come inneschi per la reazione di PCR, o sonde non perfettamente complementari. • La Tm dipende da: – – – – – Lunghezza della sonda Composizione in basi (G+C e A+T) Grado di omologia Concentrazione della sonda e del bersaglio Ambiente chimico: • Concentrazione salina (i cationi monovalenti come ioni Na+ stabilizzano la doppia elica, i denaturanti chimici come formamide o urea la destabilizzano) Ibridazione su membrana DNA a doppio filamento fusione DNA a singolo filamento Il DNA si lega sul filtro filtro Incubare con sonda marcata Lavare via il DNA marcato non ibridizzato autoradiografia Sonde per ibridazione • DNA • RNA Marcate sia - tramite radioattivita’(nucleotidi trifosfati con un fosforo radioattivo) - tramite l’uso di guppi fluorofori (nucleotidi trifosfati coniugati con guppo fluoroforo) Marcatura enzimatica di una sonda a DNA (oligolabeling) Marcatura di una sonda a RNA tramite sintesi in vitro IBRIDAZIONE DI ACIDI NUCLEICI su membrana • DNA->DNA – Southern blot – Ibridazione di colonie batteriche o placche fagiche - screening di librerie • DNA->RNA (Northern blot) • Southern e Northern blot individuano frammenti di DNA o RNA bersaglio gia’ suddivisi in base alle dimensioni tramite elettroforesi su gel Trasferimento secondo Southern (Southern blot) • Il Southern blot permette di effettuare mappaggi di restrizione sia su DNA genomico totale che su DNA ricombinante • di individuare i cosiddetti polimorfismi di lunghezza dei frammenti di restrizione (RFLP) • di individuare le delezioni o inserzioni geniche Mappatura della mutazione che determina l’anemia falciforme, che distrugge un sito MstII e genera un RFLP specifico per la malattia MstII: CCTNAGG Colony o plaque lift e ibridazione
Scaricare