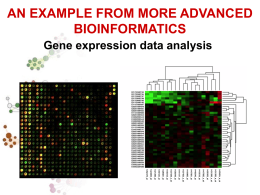
IV GIORNATA SCIENTIFICA DEL DIPARTIMENTO di BIOMEDICINA E PREVENZIONE Studio dell’espressione genica nelle patologie complesse Francesca Amati [email protected] • Nome Cognome: • SSD di afferenza: Francesca Amati MED 03 (Genetica Medica) • Patologie di maggior interesse: • Altre Patologie di interesse: Aterosclerosi e malattie cardiovascolari Cardiopatie congenite, sindromi genomiche, Abortività spontanea ricorrente • Argomenti generali di interesse: Genetica Umana, Genetica Medica,Trascrittomica, Studio degli splicing alternativi , Antisense oligonucleotide therapy, Biomarcatori Molecolari • Metodiche di laboratorio: Biologia molecolare, PCR, RT-PCR, quantitative RT-PCR, Sequenziamento, Next generation sequencing, Microarray • Collaborazioni (ambito): - UNIMI Prof. A. Catapano/Dott. G.D. Norata - Università Tor Vergata Prof. E. Piccione/Prof.ssa Pietropolli - Università Tor Vergata Prof. F. Romeo/Dott. R. Mango - ICREA/Centre de Regulació Genòmica (SPAGNA) Prof. Juan Valcárcel Juárez Aterosclerosi e malattie cardiovascolari Il principale recettore endoteliale per le ox-LDL, LOX-1 (Lectin-like oxidized low-density lipoprotein receptor1), codificato dal gene OLR1, svolge un ruolo chiave nell’aterogenesi; più recentemente, è stata dimostrata anche un’associazione tra l’aumentata attività di LOX-1 e la tumorigenesi. Amati et al., Mol. Ther Nucleic Acids, 2012 Studio dell’espressione genica L’identificazione delle basi genetiche delle patologie umane è una delle principali aree di ricerca medica Quali geni sono espressi in un tessuto ed in un dato momento dello sviluppo? Quanto ciascuno di essi e’ attivo dal punto di vista trascrizionale? C’è differenza, dal punto di vista trascrizionale, tra tessuto “malato” e non? Profilo d’espressione del genoma (TRASCRITTOMA) Microarrays Agilent SureScan Dx Microarray Scanner Applicazioni della tecnologia dei microarrays Differenze nei livelli di espressione genica (mRNA) Differenze nei livelli di espressione di microRNA (miRNA) Differenze nei livelli di espressione delle varie forme di splicing degli mRNAs Determinare l’aumento o la delezione di copie di geni in uno specifico DNA (Comparative genomic hybridization, array-CGH). Spesso si accompagna alla genotipizzazione di SNPs (single nucleotide polymorphisms) Determinare differenze nei livelli di espressione proteica (protein microarrays o protein chip) Identificare fattori di trascrizione attivi in un determinato tessuto o cellula (Ds-DNA microarrays) Identificazione di biomarcatori molecolari utilizzabili per: diagnosi di malattia classificazione e stratificazione dei pazienti risposta ai farmaci identificazione di nuovi target terapeutici Da tenere presente prima di iniziare…… Gli studi di trascrittomica forniscono un importante “fotografia” della quantità di mRNA presente in un dato momento in un sistema vivente Tenere in massima considerazione la “fonte” di mRNA. Altro elemento da tenere in massima considerazione è il gruppo di controllo (o il sistema con cui la nostra popolazione di studio deve essere confrontata) E’ sempre opportuno, una volta applicata la tecnica, effettuare degli esperimenti di validazione dei risultati utilizzando altre tecniche (RT-PCR, in situ hybridization, immunoblots and immunohistochemistry). A causa dell’etereogenità delle patologie complesse, è fondamentale per il ricercatore seguire delle linee guida comuni per la selezione e collezione dei campioni biologici In questo ambito, abbiamo collaborato a diversi progetti sia interni che esterni: Minella D, et al. SOS1 over-expression in genital skin fibroblasts from hirsute women: a putative role of the SOS1/RAS pathway in the pathogenesis of hirsutism. J Biol Regul Homeost Agents. 2011 Giganti MG, et al. A pilot study on the transcriptional response of androgen-and insulin-related genes in peripheral blood mononuclear cells induced by testosterone administration in hypogonadal men. J Biol Regul Homeost Agents. 2011 Amati F, et al., Hif1α down-regulation is associated with transposition of great arteries in mice treated with a retinoic acid antagonist. BMC Genomics. 2010 Nisticò S, et al., Effects of TNF-α and IL-1 β on the activation of genes related to inflammatory, immune responses and cell death in immortalized human HaCat keratinocytes. Int J Immunopathol Pharmacol. 2010 Minella D, et al., Androgen- and insulin-related gene signature using a specific low density oligoarray AndroChip 2 in peripheral blood mononuclear cells in agonists, recreational athletes and sedentary subjects. J Biol Regul Homeost Agents. 2010. Menghini R, et al., MicroRNA217 modulates endothelial cell senescence via silent information regulator 1. Circulation. 2009 Botta A, et al., Gene expression analysis in myotonic dystrophy: indications for a common molecular pathogenic pathway in DM1 and DM2. Gene Expr. 2007 Valentini A, et al. Valproic acid induces neuroendocrine differentiation and UGT2B7 up-regulation in human prostate carcinoma cell line. Drug Metab Dispos. 2007 Amati F, et al., Dynamic changes in gene expression profiles of 22q11 and related orthologous genes during mouse development. Gene. 2007 Bucci B, et al. Fractionated ionizing radiation exposure induces apoptosis through caspase-3 activation and reactive oxygen species generation. Anticancer Res. 2006 Gambardella S, et al., Gene expression profile study in CFTR mutated bronchial cell lines. Clin Exp Med. 2006 Amati F, et al. Gene expression profiling of fibroblasts from a human progeroid disease (mandibuloacral dysplasia, MAD #248370) through cDNA microarrays. Gene Expr. 2004 Prospettive future Con il continuo sviluppo di nuove tecnologie anche il campo dello studio dei profili di espressione genica evolverà (definizione di nuove sonde, incremento della copertura del genoma, maggiore riproducibilità dei risultati) The systems genetics is an emerging discipline that integrates highthroughput expression profiling technology and systems biology approaches for revealing the molecular mechanism of complex traits, and will improve our understanding of gene functions in the biochemical pathway and genetic interactions between biological molecules. GRAZIE…
Scaricare