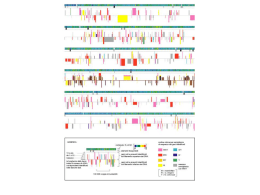
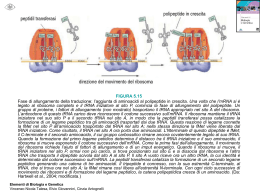
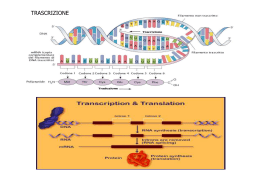
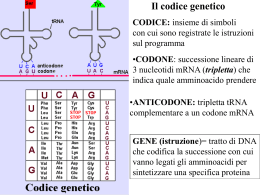
INTERAZIONE TRA mRNA, tRNA e RIBOSOMI mRNA: porta l'informazione della sequenza degli aminoacidi di una determinata proteina. tRNA: ogni tRNA è specifico per il trasporto di un determinato aminoacido – Ribosoma: officina che permette il corretto appaiamento tra mRNA e i diversi tRNA e partecipa attivamente alla formazione della catena proteica. Componenti della traduzione STRUTTURA MOLECOLARE DEL tRNA Nel piano: il filamento 5'→3' assume una forma detta a quadrifoglio con bracci in cui le basi sono appaiate e anse senza appaiamento per basi modificate post-trascrizionalmente. - ANSA D: vi si lega l'enzima aminoacilsintetasi specifico per legare un determinato aminoacido al suo tRNA. - ANSA dell'ANTICODONE: contiene una tripletta complementare e antiparallela al codone che sull'mRNA codifica per un determinato aminoacido. -ANSA del TϕC (ϕ=pseudouracile): necessaria per il legame tra il tRNA e rRNA nella subunità grande del ribosoma -Il tRNA termina al 3' con un codone CCA a cui viene legato l'aminoacido specifico di quel tRNA. Il tRNA nello spazio si ripiega aminoacido in una struttura detta a L rovesciata dove sono bene accessibili e visibili l'ansa dell'anticodone e l'aminoacido legato. anticodone Legame tra aminoacido e tRNA L'aminoacido con il suo gruppo carbossilico (-COOH) si lega all'-OH del C3 del ribosio del nucleotide (A) al 3' del tRNA. I tRNA sono più di 20 (circa 30-40 in procarioti e anche 50 in eucarioti) per cui ad alcuni aminoacidi corrisponde più di un tRNA. Gli enzimi che legano l’aminoacido al rispettivo tRNA (aminoacil-sintetasi) sono 20 come il numero degli aminoacidi. Ognuno trasferisce un’ determinato AA al rispettivo tRNA. STRUTTURA MOLECOLARE DEL RIBOSOMA Ricordiamo che: in Eucarioti gli rRNA vengono prodotti nel nucleolo (tranne il 5S prodotto nel nucleo) dove entrano anche le proteine ribosomiali prodotte nel citoplasma che si assemblano con i rispettivi rRNA per dare i ribosomi in Procarioti tutto ciò avviene nel citoplasma. -Il ribosoma ha una struttura molecolare complessa formata da rRNA e proteine ribosomiali. -I ribosomi sono formati da due subunità (piccola e grande) che si assemblano solo al momento della traduzione. I ribosomi procariotici sono più piccoli dei ribosomi eucariotici: le differenze tra ribosomi procariotici ed eucariotici sono dovute al numero e alle grandezze diverse degli rRNA e al numero e alle grandezze diverse delle proteine. Struttura molecolare dell’ mRNA -Il filamento di mRNA inizia al 5’con il CAP (7-metilguanosina): si legherà all'rRNA 18S durante la traduzione seguito dalla sequenza LEADER che non viene tradotta in proteina -la sequenza codificante inizia sempre con la tripletta AUG e termina con una tripletta detta di STOP. -segue un tratto detto TRAILER (rimorchio) non codificante e termina al 3’ con la coda poliA +1 leader AUG STOP 10-200 nt. CAP CH3-7-G poli-A 50-250 nt. proteina Ciascun gene che codifica per una proteina ha il suo proprio promotore. Trascrizione e traduzione avvengono nello stesso distretto (citoplasma) e sono contemporanee nei Procarioti Trascrizione e traduzione avvengono in distretti diversi e non sono contemporanee negli Eucarioti IL CODICE GENETICO Perché avvenga la traduzione è necessario: passare da un linguaggio fatto di NUCLEOTIDI ad un linguaggio di AMINOACIDI. Gli aminoacidi presenti in tutte le proteine sono 20 mentre le basi sono solo 4. Se ogni base specificasse per 1 amminoacido, avremmo solo 4 aminoacidi specificati 41=4. Se la combinazione di due basi specificasse per 1 aminoacido, avremmo:42=16 aminoacidi specificati. Se la combinazione di tre basi specificasse per 1 aminoacido, avremmo: 43=64 combinazioni per 20 aminoacidi,più che sufficienti per specificare i 20 aminoacidi Il codice pertanto è a TRIPLETTE cioè la combinazione di tre basi specifica per 1 aminoacido. Ogni tripletta è detta codone Esistono 3 triplette non codificano per nessun aminoacido e sono dette codoni di STOP (UAG; UAA; UGA). Poiché ci sono 64-3 (codoni di stop)=61 triplette per 20 aminoacidi si dice che il codice è RIDONDANTE, cioè ogni aminoacido può essere specificato da più di un codone. Es. leucina 6 codoni, valina 4 codoni, triptofano e metionina 1 codone, etc…. CARATTERISTICHE DEL CODICE GENETICO Il codice genetico è a TRIPLETTE cioè la combinazione di tre basi specifica per 1 aminoacido Il codice genetico è RIDONDANTE cioè ogni aminoacido può essere specificato da più di un codone. Il codice genetico NON E' AMBIGUO cioè un determinato codone specifica per un solo aminoacido Il codice genetico è UNIVERSALE cioè uguale per tutti gli esseri viventi tranne alcune eccezioni. Il codice genetico è SENZA PUNTEGGIATURA cioè viene letto linearmente di seguito di tre basi in tre basi e non è SOVRAPPONIBILE (es. la stessa base non può essere letta come l’ultima di un codone e la prima del successivo). MUTAZIONI PUNTIFORMI IN ZONE CODIFICANTI A) CAMBIO di una base: Es. da A a G Conseguenze: 1) nessuna se ad essere colpita è la terza base di un codone è probabile che il nuovo codone codifichi per lo stesso aminoacido. Es. AGU→AGC codificano per Serina 2) cambio dell'aminoacido specificato dal nuovo codone. Es. UCU→CCU si passa da serina a prolina 3) proteina più corta se un codone per l'aminoacido si tramuta in un codone di STOP Es. UUA→UAA dal codone per leucina a un codone di stop 4) proteina più lunga se ad esempio da un codone di stop per mutazione si passa ad un aminoacido qualsiasi B) DELEZIONE o INSERZIONE di una base Cambio della cornice di lettura. Da quel nucleotide (deleto o inserito) la proteina sarà tutta diversa perché a codoni diversi corrisponderanno AA diversi AUG⏐CAA⏐CCC⏐GGA⏐UAA⏐GCU⏐UAA delezione AUG⏐CAA⏐CCC⏐GGU⏐AAG⏐CUU⏐AA… TRADUZIONE INIZIO: -L'mRNA si lega alla SUBUNITA' PICCOLA ribosomiale e al tRNAmet (codone con anticodone) grazie a fattori di inizio e ad 1 GTP→GMP+P+P che dona energia necessaria per il legame. -L'mRNA in eucarioti l'mRNA si lega con il cappuccio al 18S rRNA. -Successivamente si assembla la SUBUNITÀ GRANDE contenente due siti detti A e P per accogliere i tRNA. Solo il 1° tRNAmet si lega direttamente al sito P 1) La traduzione inizia sempre con Metionina il codone per Met èAUG, l’anticodone UAC 2) ALLUNGAMENTO -Il 2° tRNA con l‘AA corrispondente si lega all'mRNA (2°codone/anticodone) e alla subunità grande, nel sito A del ribosoma, grazie a fattori di allungamento e ad 1 molecola di GTP che si scinde donando energia. -Il legame che si forma tra i due AA avviene tra il gruppo carbossilico (-COOH) della metionina e il gruppo amminico (-NH2) del 2° AA (legame peptidico) ad opera della Peptidiltransferasi. -Dopo la formazione del legame peptidico il 1° tRNA, rimasto scarico della metionina, viene allontanato dal sito P. -Avviene quindi la traslocazione: l'mRNA scivola di un codone trascinandosi il 2° tRNA che passa dal sito A al sito P, lasciando il sito A libero per l'attacco del 3° tRNA. Questa traslocazione è mediata da un fattore di traslocazione e dalla scissione di una molecola di GTP. 1) La lettura dell'mRNA procede in direzione 5'→3‘ 2) La sintesi proteica procede dall'estremità N-terminale all'estremità C-terminale del polipeptide 3) Il ciclo si ripete tante volte quanti sono gli AA che devono essere legati. 3) TERMINAZIONE Quando sul sito A si trova un codone di STOP, a cui non corrisponde nessun tRNA, la sintesi si arresta. Inoltre esistono fattori di rilascio che si legano al sito A impedendo comunque l'attacco dei tRNA. La catena polipeptidica si stacca dall'ultimo tRNA grazie ad un enzima (idrolasi) con consumo di una molecola di GTP. Le due subunità ribosomiali si disassemblano. antibiotici (Ammanita falloide) IL VACILLAMENTO DELLE BASI Appaiamenti Il legame tra codone e anticodone è complementare e antiparallelo codone/anticodone Es. 3‘CAG5’ anticodone 5‘GUC3' codone Il numero dei codoni (61) è più grande di quello degli anticodoni. 61 codoni mentre i tRNA sono circa 30-40 in procarioti e anche 50 in Eucarioti) Questo implica che alcune molecole di tRNA con il loro anticodone sono capaci di accoppiarsi a più di un codone. I tRNA hanno una struttura tale da richiedere un appaiamento accurato nelle prime due posizioni del codone (5') e da tollerare un appaiamento scorretto (oscillante) in terza posizione. Questo appaiamento approssimativo rende possibile combinare 20 AA ai loro 61 codoni servendosi per esempio di solo 30/50 molecole di tRNA. Frequentemente al 5' dell'anticodone (corrispondente alla terza base del codone) esiste la base modificata inosina capace di complementarsi sia con A, C, U.
Scarica