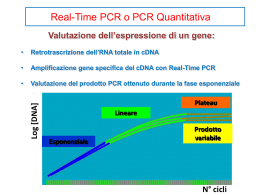
Metodi per la rilevazione e quantificazione della resistenza in patogeni fungini R.M. De Miccolis Angelini, S. Pollastro, S. Toffolatti, A. Vercesi DIPARTIMENTO DI BIOLOGIA E CHIMICA AGROFORESTALE ED AMBIENTALE - DiBCA DIPARTIMENTO DI SCIENZE AGRARIE E AMBIENTALI PRODUZIONE, TERRITORIO, AGROENERGIA Perché? z z z z z z Verificare casi di sospetta resistenza Segnalare la comparsa di forme resistenti Seguire l’evoluzione della resistenza Verificare l’efficacia delle strategie antiresistenza Modulare la scelta dei fungicidi a livello locale Migliorare le conoscenze Obiettivo Sensibilità di base CE50 % Inibizione 100 50 0 MIC (CE100) Curve dose-risposta S Inibizione (%) 100 80 LR 60 HR 40 20 0 0 0,1 1 10 100 Log concentrazione fungicida Distribuzione di ceppi di P. viticola in funzione dell’EC50 a fenamidone EC50 0,1 1 10 100 Risposta di ceppi di Botryotinia fuckeliana a fenhexamid EC50 Resistente FR = EC50 Sensibile Campionamento Campionamento Un numero eccessivo di campioni può essere inutile ai fini del monitoraggio PCR in tempo reale Quantificazione di DNA mutato z Conteggio delle colonie z Germinazione conidica z Crescita micelica Saggi di germinazione sulle oospore 2 1 •Isolamento delle oospore •Risospensione in acqua dopo filtrazione 3 • Incubazione di 1200 oospore a 20°C su: 1. Substrato di controllo (agar-acqua 1%) 2. Agar-acqua addizionato con fungicida • Concentrazione discriminante • Concentrazione crescente Conteggio delle oospore germinate Trascorsi 7-10-14 giorni dall’inoculazione 4 Calcolo delle percentuali di germinazione nr. oospore germinate × 100 G= nr. totale di oospore Calcolo della percentuale di oospore resistenti (RO) RO = Gfungicida × 100 Gagar − acqua Piastre per microtitolazione Composizione del substrato Es: Resistenza alle anilinopirimidine, SDHI Colony growth inhibition (%) Saggi in vitro per la resistenza a QoI 100 80 60 MEA MEA+SHAM 40 20 0,01 0,1 1 10 100 Trifloxystrobin concentration (μg ml-1) Substrato addizionato di inibitori specifici di respirazioni alternative (es. SHAM, n-propyl gallate) z Resistenza associata ad una alterazione genica z Presenza/assenza di mutazioni (SNPs) z Percentuale di allele mutato z Interpretazione del risultato (ploidia,eterocariosi,eteroplasmia) Molecolari Eterocariosi e eteroplasmia Resistente Fungicida DNA mutato DNA ‘wild-type’ Sensibile Tecnica Single-stranded conformational polymorphism PCR (PCR-SSCP) Allele-Specific PCR High-resolution melting detection TaqMan real-time PCR Luminex Molecular Diagnostics Nested PCR-RFLP primer-introduced restriction analysis PCR (PIRA-PCR) rolling circle amplification amplification (RCA-PCR) PCR-restriction fragment length polymorphism (PCR-RFLP) Reverse Transcription PCR Assay Allele-specific probe and primer amplification assay (ASPPAA PCR) Patogeno Riferimento bibliografico Srinivasa et al., 2012; Plant Pathogenic Nahiyan et al., 2011; Bacteria; Fusarium sp.; Patel et al., 2011; Pyrenophora tritici; Veloukas et al., 2011 Botrytis cinerea Cercospora beticola Malandrakis et al., 2011 Botrytis cinerea Billard et al., 2010 Botrytis cinerea; Plasmopara viticola Billard et al., 2012; Samuel et al., 2011; Valsesia et al., 2005 Clinically Relevant Fungi Babady et al., 2011 Botrytis cinerea Saito et al., 2007 Fusarium graminearum Luo et al., 2009 Penicillium marneffei Jiufeng et al., 2010 Botrytis cinerea; Magnaporthe oryzae; Colletotrichum gloeosporioides Mycosphaerella graminicola Botrytis cinerea Kim et al., 2009; Wei et al., 2009; Chung et al., 2009 Guo et al., 2005 Billard et al., 2012 z Impiego di enzimi di restrizione z Analisi del profilo elettroforetico z Discriminazione tra genotipi sensibili (S) o resistenti (R) AluI S R S 1000 pb 500 pb R Primer Allele-Specifici (AS) C3387 G3387 Genotipo Sensibile (S) Genotipo Resistente (R) G G G C S R S S S R R S R S R R Rilevazione della mutazione G143A nel gene cytb 1 2 SOSPENSIONE DI OOSPORE/CONIDI IN ACQUA ESTRAZIONE E QUANTIFICAZIONE DEL DNA TOTALE 3 Sonde Taqman REAL TIME PCR ALLELE SPECIFICA E QUANTIFICAZIONE DELLA % DI ALLELE MUTATO NELLA POPOLAZIONE (MA) Costruzione curve standard per quantificazione di DNA DNA totale DNA mutato Rilevazione della mutazione G143A nel gene cytb Molecolari Real-time PCR – SYBR green Punti critici Metodi standard Punti critici Esempio Botrytis cinerea - Muffa grigia Da usare per Tipo di saggio Proposto da AP Pianta intera X Microtitolazione Microtitolazione Altri X Lachase X Mehl X Microtitolazione Crescita radiale delle colonie SBI SDHI QoI X X X X Sierotzki Stammler Takagaki Comparazione tra metodi esempio: Botrytis cinerea Microtitolazione Fenotipo BosS BosLR BosHR BosMR Germinazione conidica Substrato liquido CE50 0,2 > 10 > 10 0,94 CE95 > 10 > 10 > 10 > 10 Crescita micelica Substrato agarizzato CE50 < 0,01 1-40 > 100 - CMI 0,1 > 100 > 100 - CE50 0,29 > 100 > 100 15,25 CMI 10 > 100 > 100 > 100 esempio: oospore e QoI % Oospore resistenti % Allele mutato 100 80 60 40 20 BBD BAI BBC BAM BAR BAT BAH BAU BAO BAN BAP BAB BAZ BAF BAG BAS BAD BAA BAE 0 Conclusioni La tecnica ideale z In grado di fornire risultati: - realistici - quantitativi - riproducibili - di facile ed univoca interpretazione z Di facile applicabilità (dotazione strumentale, competenze dell’operatore, ecc.) z Economica, ad elevata processività in tempi brevi
Scaricare