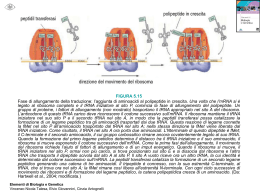
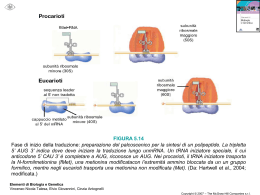
SINTESI PROTEICA Replicazione Trascrizione Traduzione 61 codoni codificanti 3 triplette non senso (STOP) AUG codone di inizio codone per Met Caratteristiche del codice genetico Specificità Specificità Il codice genetico è specifico (non ambiguo): un dato codone codifica sempre e soltanto per un aminoacido Universalità Universalità Il codice genetico è universale, cioè la specificità si è conservata durante l’evoluzione Ridondanza Il codice genetico è ridondante (DEGENERATO): più triplette possono codificare per lo stesso aminoacido Assenza di sovrapposizioni e di una punteggiatura Il codice genetico è letto, a partire da un dato punto di inizio, come una sequenza continua di basi, considerate tre per volta Tre possibili quadri di lettura Conseguenze di una mutazione puntiforme L’appaiamento codone-anticodone è antiparallelo Ipotesi della terza base oscillante: la prima base dell’anticodone (5’) ha un orientamento nello spazio meno definito rispetto alle altre, che consente l’accoppiamento con piu’ di un codone che differiscono per la terza base. Questo spiega perché non ci sono 61 molecole di tRNA tante quante i 61 codoni codificanti. C e A sull’anticodone hanno un legame molo specifico quindi riconoscono solo un codone Se la prima base è U o G il legame è meno specifico, possono essere riconosciuti 2 codoni Se c’è l’inosina I si possono leggere 3 codoni. I componenti necessari alla traduzione: •Gli aminoacidi •Gli RNA transfer •Le aminoacil-tRNA sintetasi •L’RNA messaggero •I ribosomi funzionalmente attivi •I fattori proteici (fattori di inizio, di allungamento e di terminazione o di rilascio) •ATP e GTP come fonti di energia Durante la sintesi proteica sono necessari tutti gli aa a prescindere dalla % relativa. Importanza di un apporto adeguato con la dieta (aa essenziali) •La sintesi proteica avviene in 5 stadi: 1. Attivazione degli amminoacidi 2. Inizio 3. Allungamento 4. Terminazione e rilascio del ribosoma 5. Ripiegamento e modificazioni posttraduzionali I ribosomi Complessi formati da RNA e proteine Sono formati da due subunità: subunità maggiore e subunità minore Nelle cellule eucariote si trovano liberi nel citosol o associati al reticolo endoplasmatico Gli RNA transfer (tRNA) Per ogni aa è necessario almeno uno specifico tRNA. Nell’uomo ci sono almeno 50 specie di tRNA, nei batteri 30-40. Ciò significa che ad alcuni aa corrisponde più di uno specifico tRNA Sono formati da una singola catena di RNA, 73-93 residui. Contengono basi e zuccheri modificati tRNA carico: legato ad un aa tRNA scarico: non legato ad un aa aa attivato: legato a tRNA Strutture caratteristiche Braccio dell’amminoacido Ansa (o braccio) dell’anticodone Braccio D Braccio TψC Alcuni hanno un quinto braccio più corto D: 5,6 diidrouridina ψ: pseudouridina 1-Attivazione degli amminoacidi- Formazione di un aminoacil-tRNA Avviene nel citosol Ciascuno dei 20 amminoacidi viene legato ad un tRNA specifico. La reazione è catalizzata dalla aminoacil-tRNA sintetasi Aminoacil-tRNA sintetasi è specifica per un amminoacido e per un tRNA Se un amminoacido ha più tRNA la stessa aminoacil-tRNA sintetasi catalizza l’attivazione. Esistono due classi di enzimi. Hanno differenze nella struttura primaria e terziaria e nel meccansimo di reazione Mg2+ Amminoacido+tRNA+ATP Amminoacil-tRNA +AMP + PPi La reazione avviene in due passaggi: -si forma un amminoacil-adenilato -il gruppo amminoacilico viene trasferito sul tRNA L’amminoacil adenilato è legato al sito attivo dell’enzima La reazione è irreversibile. legame estere tra il carbossile dell’aa e 3’ OH del riboso dell’adenosina. L’amminoacilazione ha due obiettivi: (1) l’attivazione di un amminoacido per la formazione di un legame peptidico (2) l’attacco dell’amminoacido ad un tRNA che lo porta nella giusta posizione nel polipeptide nascente Aminoacil-tRNA sintetasi è specifica per un amminoacido e per un tRNA Se un amminoacido ha più tRNA la stessa aminoacil-tRNA sintetasi catalizza l’attivazione. La capacità di scegliere il giusto tRNA è importante per la fedeltà della sintesi proteica. Alcune posizioni (blu) sono le stesse in tutti i tRNA Altre posizioni sono punti di riconoscimento per una (arancione) o più (verde) aminoacil-tRNA sintetasi 2- INIZIO Uno specifico amminoacido dà inizio alla sintesi proteica Codone start: AUG Codifica per la Metionina. Tutti gli organismi hanno due tRNA per la Metionina anche se c’è un solo codone che codifica per essa, un tRNA se Met è il primo aa, l’altro se Met è interno. Nei batteri il primo amminoacido è la N-formilmetionina (fMet) Batteri L’inizio della sintesi proteica nei batteri richiede: 1)La subunità 30S del ribosoma 2)L’mRNA 3) l’fMet-tRNA 4) Tre proteine, i Fattori di Inizio (IF1, IF2, IF3) 5) GTP 6) La subunità 50S 7) Mg2+ Il codone di inizio viene diretto verso la sua corretta posizione da un segnale di inizio sull’mRNA chiamato sequenza di Shine-Dalgarno. Questa sequenza viene riconosciuta dal rRNA nella subunità 30S. Questa interazione “blocca” l’mRNA in modo che AUG sia posizionata correttamente nel ribosoma. I ribosomi batterici hanno siti di legame per le molecole di tRNA: A, sito amminoacilico P, sito peptidilico E, sito di uscita Il codone di inizio si posiziona nel sito P Durante la fase di allungamento gli altri amminoaciltRNA entranti si legano prima al sito A e dopo a P ed a E. Si forma un ribosoma 70S funzionale chiamato complesso di inizio Nelle cellule eucariote la formazione del complesso di inizio coinvolge 9 fattori di inizio Batteri 3-ALLUNGAMENTO a) Legame del secondo aminoacil-tRNA Entrano in gioco i Fattori di allungamento, EF-Tu, EF-Ts, EF-G b) Formazione del primo legame peptidico c) Traslocazione Il ribosoma si sposta di un codone verso la terminazione 3’ dell’mRNA. Il dipeptidil-tRNA a sua volta si sposta dal sito A al sito P mentre il tRNA deacilato andrà nel sito E da dove verrà rilasciato nel citosol. Questo spostamento richiede l’intervento di EF-G e energia fornita dall’idrolisi di una molecola di GTP. Dopo la traslocazione il ribosoma ha la posizione A libera per accogliere un nuovo amminoacil-tRNA. Negli eucarioti l’allungamento procede nello stesso modo I fattori di allungamento eucariotici eEF1α, eEF1βγ ed eEF2 hanno funzioni analoghe a quelli batterici. Nel ribosoma eucariotico non c’è il sito E, i tRNA scarichi vengono rilasciati direttamente nel citosol. 4- TERMINAZIONE L’allungamento procede finchè il ribosoma aggiunge l’ultimo aa codificato dall’mRNA. Il segnale è dato da uno dei 3 codoni di stop. Quando il codone di terminazione occupa il sito A, intervengono tre Fattori di terminazione, RF1, RF2 RF3: 1) Idrolisi del legame terminale del peptidiltRNA 2) Rilascio del polipeptide libero e di tRNA scarico 3) Dissociazione del ribosoma. RF1 e RF2 riconoscono i codoni di stop RF3 ha un ruolo non chiarito. Negli eucarioti un solo fattore di rilascio riconosce le tre triplette di stop Il polisoma Nei batteri trascrizione e traduzione sono accoppiate. Negli eucarioti non c’è accoppiamento perchè gli mRNA appena formati devono essere trasferiti dal nucleo nel citoplasma. La sintesi proteica è inibita da antibiotici e tossine La Puromicina ha una struttura simile all’amminoacil-tRNA, si può legare sul sito A e partecipa alla fromazione del legame peptidico. Si forma la peptidil-puromicina che provoca il rilascio del peptide e l’interruzione della traduzione. Le tetracicline bloccano il sito A del ribosoma e impediscono il legame dell’amminoacil-tRNA Il cloramfenicolo blocca la peptidiltrasferasi TOSSINE La Tossina Difterica inattiva eEF2 La Ricina inattiva la subunità 60S
Scaricare