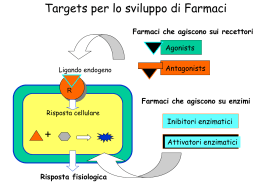
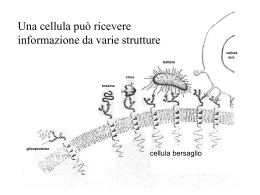
Meccanismi molecolari di trasduzione del segnale. Le vie di trasduzione del segnale sono molto specifiche ed estremamente sensibili. La sensibilita’ delle vie di trasduzione dipende da 3 fattori: -l’affinita’ tra ligando (segnale) e il suo recettore puo’ essere espressa dalla costante di dissociazione Kd , molto spesso con valori intorno a 10-10 M o anche meno; cio’ significa che il recettore puo’ identificare le molecole segnale anche a concentrazioni pM. -la cooperativita’ nelle interazioni ligando-recettore determina una grande variazione nello stato di attivazione del recettore in risposta a una piccola variazione della concentrazione del ligando (effetto della cooperativita’ sul legame dell’ossigeno all’emoglobina) -l’amplificazione mediante una cascata enzimatica inizia con l’attivazione di un singolo enzima associato al recettore del segnale, che a sua volta catalizza l’attivazione di molte molecole di un secondo enzima, ognuna delle quali determina poi l’attivazione di un terzo enzima e cosi’ via. Con queste cascate si possono ottenere amplificazioni di diversi ordini di grandezza in pochi millisecondi. La sensibilita’ dei recettori puo’ essere modulata. -quando un segnale perdura nel tempo, si ha una desensibilizzazione del recettore; quando la concentrazione dello stimolo scende sotto un certo livello, il sistema riacquista la sua sensibilita’. Cosa accade nel sistema di trasduzione visivo quando si passa dalla luce al buio e viceversa? - un’altra proprieta’ importante dei sistemi di trasduzione dei segnali e’ l’integrazione, cioe’ la capacita’ del sistema di ricevere segnali multipli e di produrre una sola risposta approppriata alle necessita’ della cellula o dell’organismo. Vie diverse di trasduzione del segnale convergono l’una con l’altra a diversi livelli generando un’abbondanza di interazioni che serve a mantenere l’omeostasi nelle cellula o nell’organismo. I dettagli molecolari di diverse vie di trasduzione. Il fattore di innesco e’ diverso per ogni sistema, ma le caratteristiche generali delle vie di trasduzione del segnale sono comuni a tutte: un segnale interagisce con il suo recettore; il recettore attivato interagisce con un macchinario cellulare che produce un secondo segnale o una modificazione dell’attivita’ di una proteina cellulare, l’attivita’ metabolica della cellula bersaglio viene alterata, la trasduzione e’ terminata e la cellula ritorna allo stato originario, quello precedente all’arrivo del segnale. Recettori canali ionici Il legame del ligando modifica la conformazione del recettore in modo tale che specifici ioni possono fluire attraverso esso, il risultante movimento ionico altera il potenziale elettrico della membrana cellulare. Recettori con attivita’ enzimatica intrinseca Recettori transmembrana che contengono ed esprimono attivita’ enzimatiche. Quando uno di questi recettori viene attivato da un ligando extracellulare, catalizza direttamente la produzione di un secondo messaggero intracellulare. Recettori senza attivita’ enzimatica intrinseca: accoppiati alla tirosin cinasi Questi recettori sono privi di attivita’ catalitica intrinseca, ma il legame del ligando induce la formazione di un recettore dimerico che interagisce ed attiva una o piu’ tirosina cinasi citosolica. Recettori senza attivita’ enzimatica intrinseca Recettori privi di attivita’ enzimatica, ma che legano e attivano enzimi citoplasmatici che modificano altre proteine, convertendole direttamente in regolatrici dell’espressione genica, oppure attivando una cascata enzimatica che alla fine attiva un gene regolatore. Recettori accoppiati a proteine G Recettori proteici della membrana plasmatica che attivano indirettamente, attraverso proteine che legano il GTP ( protein G), enzimi che a loro volta producono un secondo messaggero. Recettori accoppiati a protein G e loro effettori • • • • • • 7 tratti transmembrana > 2000 recettori Presenti in tutti gli eucarioti Funzioni biologiche diverse Molecole segnale: proteine, peptidi, lipidi e altre piccole molecole Struttura simile Recettori 7TM Il 50% dei farmaci sono attivi su questi recettori (di questi il 25% dei 200 farmaci più usati) Sono state identificate 6 famiglie di questi recettori e di queste le prime 3 sono ben studiate. Famiglie di recettori 7TM • A: include recettori della visione ed olfattivi • B: include solo 25 membri (recettori per glucagone, ormoni gastrointestinali, secretina ecc) • C: piccola famiglia che include recettori del gusto e il recettore metabotropico del glutammato • Circa 200 recettori “orfani” •Sono comunemente definiti come recettori collegati a proteine G S e g n a la z io n e t r a m it e r e c e t t o r i d i s u p e r f ic ie c o lle g a ti a p r o t e in e G e t e r o t r im e r ic h e Proteine G Famiglie di proteine G Proteine G Il genoma umano contiene: 20 geni che codificano per subunità α 5 geni per subunità β 6 geni per subunità γ In teoria almeno 1000 combinazioni diverse. Amplificazione e diversificazione del segnale Le protein G legano i nucleotidi guanidilici, e agiscono come interruttori molecolari attivati dal legame del GTP e inattivati quando questo e’ idrolizzato a GDP. L’idrolisi del GTP e’ catalizzata dalle protein G stesse che posseggono un’attivita’ GTP-asica. RGS proteine regolatrici dell’idrolisi, che legandosi alle protein G attive accelerano l’idrolisi del GTP. Molte protein G sono eterodimeri formati da una subunita’α 45 kD, una subunita’ β 35 kD, e una subunita’ γ 8 kD. Si stima che il genoma umano contenga oltre 1000 geni differenti che codificano recettori a 7 eliche transmembrana, oltre a un numero di geni per le subunita’ α, β e γ sufficiente a permettere l’esistenza di centinaia di differenti proteine G. Le subunita’ α e γ sono ancorate alla membrana da lipidi legati covalentemente alla regione N-terminale della della catena Gα e alla regione C-terminale della catena Gγ. La subunita’ β e’ sempre complessata con la subunita’ γ con cui forma l’eterodimero Gβγ. Nello stato inattivo una proteina G forma un complesso Gα-GDP combinato con una molecola di eterodimero Gβγ che si lega al dominio citosolico di un recettore.Se il recettore viene attivato, un segnale attraversa la membrana attivando il dominio citosolico come conseguenza di una modificazione conformazionale. La proteina G attiva catalizza lo scambio del GDP con il GTP. La proteina G attivata viene quindi liberata dal recettore e si dissocia nei due componenti attivi Gβγ e Gα-GTP, questi trasmettono il segnale a proteine effettrici che producono secondi messaggeri come l’AMP ciclico responsabili delle risposte fisiologiche. L’attivita’ GTP-asica della subunita’ Gα determina la durata della permanenza del segnale nella forma attiva, l’idrolisi del GTP a GDP riporta la subunita’ Gα nella forma inattiva che si lega al dimero Gβγ. La subunita’ Gα (trasducina) ha una catena polipeptidica divisa in 2 domini, 1 dominio GTPasico e 1 dominio α elica collegato al dominio GTP-asico da 2 regioni linker una delle quali segue l’elica α1 mentre l’altra precede il filamento β2 nel dominio GTPasico. Il dominio a elica e’ formato da una lunga α elica di 28 residui che agisce da impalcatura che sostiene 5 eliche piu’ piccole per mezzo di interazioni idrofobe che forniscono una struttura interna rigida in accordo con i dati indicanti che l’intero dominio funziona come un coperchio rigido posto al di sopra del sito di legame del nucleotide e che cambiamenti conformazionali potrebbero agire mediante movimenti a cerniera che provocano l’apertura e la chiusura dell’intero coperchio. Le subunita’ Gα sono estese al loro terminale amminico di circa 30 residui che nella Gα libera sono disordinati mentre assumono una conformazione ad elica quando la subunita’ si trova associata nel trimero Gαβγ. Il dominio catalitico di Gα ha struttura tridimensionale simile alle proteine Ras G=glicina X=aa n.d. S=serina T=treonina K=lisina D=acido aspartico E=acido glutammico N=asparagina Struttura del tipo α/β in cui il foglietto β centrale comprende 6 filamenti β di cui 5 paralleli. Sono presenti 5 α eliche su entrambi i lati del foglietto β. Il GTP e’ legato in una tasca alle estremita’ carbossiliche dei filamenti β. Le regioni loop che partecipano all’attivita’ della proteina sono indicate G1-G5. I loop G1, G3 e G4 presentano rispettivamente le sequenze consenso G-X-X-X-X-K-S/T, D-X-X-E, e N-K-X-D, presenti nelle varie proteine leganti GTP e le subunita’ Gα delle proteine G trimeriche. Il loop G1, detto anche loop legante il difosfato o P-loop, collega il filamento β1 con la elica α1, e’ essenziale per il corretto posizionamento dei gruppi fosfato legandosi ai gruppi fosfato in α e in β del nucleotide guaninico. Il loop G3, che unisce β3 con α2 forma un collegamento tra i sottositi che legano Mg2+ e il fosfato in γ del GTP. Il loop G2 contiene il residuo di treonina conservato sia in Ras che nella subunita’ Gα che lega il Mg2+ importante per l’idrolisi del GTP. I loop G4 e G5 sono coinvolti nel riconoscimento e nel legame della base guanina del nucleotide. Le modificazioni conformazionali che avvengono quando la molecola passa dalla forma attiva a quella inattiva avvengono nella regione interruttore 1 nel loop G2, e interruttore 2 nel loop G3. Quasi tutti gli enzimi che idrolizzano nucleosidi fosfati trifosfati, compresi Ras e Gα, richiedono ioni Mg2+ per l’attivita’ catalitica. Il Mg2+ e’ coordinato a un atomo di ossigeno del fosfato β e γ del GTP e alle catene laterali dei residui Ser 17 e Thr 35 di Ras. Due molecole di acqua completano la coordinazione del magnesio. Passando da Gα-GDP inattiva a Gα-GTP attiva le differenze strutturali interessano le 3 regioni del dominio GTP-asico l’interruttore I e II e l’interruttore III e’ un loop che collega β4 con α3, corrispondente alla regione G4 in Ras. L’elemento che scatena il cambiamento conformazionale della forma GDP alla forma GTP sembra essere la formazione di legami idrogeno tra la proteina e il fosfato γ del GTP accoppiata alla formazione di nuovi legami con lo ione Mg2+ da parti di ligandi della proteina. Interruttore I: la regione viene tirata verso il fosfato γ in modo da portare la catena laterale del residuo di Thr 177 in posizione a formare un legame a idrogeno con l’atomo di ossigeno del fosfato e anche in prossimita’ del Mg2+, dove sostituisce una delle 2 molecole di acqua presenti nella forma legata al GDP. Interruttore II: si forma un legame a idrogeno tra il gruppo NH della catena appartenente al residuo Gly 199 e il fosfato γ del GTP. Il loop contenente la Gly 199 e’ accoppiato all’elica α2 e il movimento di questo loop comporta lo stiramento e la rotazione con conformazione diversa da quella che assume nella forma legata al GDP. Ne consegue che l’estremita’ β3 si sposta da β1 interrompendo due legami a idrogeno. La nuova conformazione e’ stabilizzata da altri legami idrogeno tra β3 e β2 e da ponti salini tra residui di Arg presenti nell’interruttore II e residui di glutammato nella regione III. Interruttore III: la regione non ha interazioni dirette con il fosfato γ ma forma una rete di interazioni altamente conservate con residui presenti nell’interruttore II. I cambiamenti strutturali negli interruttori I e II, innescati dal fosfato γ si propagano indirettamente all’interruttore III. Questi 3 interruttori sono collocati in un’area della subunita’ Gα che e’ implicata in interazioni sia con la subunita’ β di Gβγ sia con le molecole attivatrici della funzione GTP-asica. Le GTPasi: idrolizzano GTP mediante un attacco nucleofilo di una molecola d’acqua Idrolizzano il GTP a GDP e fosfato inorganico mediante un trasferimento diretto all’acqua del fosfato γ senza la formazione di un intermedio legato covalentemente. Questo meccanismo richiede la presenza di una base catalitica che possa sottrarre un protone da una molecola d’acqua producendo un gruppo ossidrilico che attacca l’atomo di fosforo del fosfato γ. Il residuo di Gln della regione dell’interruttore II viene portato in profondita’ vicino al fosfato γ, ed e’ importante per la catalisi quando il suo atomo di ossigeno accetta un protone da una molecola d’acqua mentre l’atomo di azoto della Gln cede un protone al fosfato γ. Il residuo di Gln, insieme a uno di Arg e uno di Thr appartenenti alla regione interruttore I, riduce la carica negativa del fosfato γ attraverso la formazione di legami a idrogeno stabilizzando in questo modo il presunto complesso dello stato di transizione. GEF= fattore di scambio dei nucleotidi guaninici GAP= proteina che attiva la GTPasi. Ras non possiede il residuo di Arg nell’interruttore I che stabilizza la transizione quindi interviene una GAP. La struttura del complesso Ras-GAP mostra che la GAP fornisce un residuo di Arg al sito attivo di Ras.
Scaricare