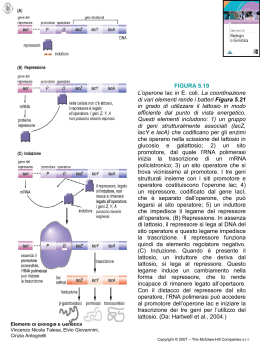
Le principali strategie di regolazione dell’espressione genica nei procarioti Regolazione metabolica Nel genoma di un microorganismo sono presenti migliaia di geni (3000--6000). (3000 Alcuni geni vengono espressi continuamente (geni costitutivi); altri solo quando è necessario (geni regolati). ENZIMI COSTITUTIVI (metabolismo del glucosio) glucosio) ENZIMI INDUCIBILI (prodotti in presenza del substrato es. β–galattosidasi nel metabolismo del lattosio) Regolazione metabolica Gli organismi sono in grado di regolare l’espressione dei propri geni in modo che le proteine, e le altre molecole, vengano sintetizzate nella quantità esatta e nel momento esatto del ciclo cellulare. I MECCANISMI DI REGOLAZIONE Consentono alla cellula di produrre proteine (enzimi) solo quando servono ECONOMIA CELLULARE Meccanismi utilizzati nella regolazione Nella cellula ci sono due strategie principali di regolazione: Controllo dell’attività di un enzima, dopo che la proteina è stata sintetizzata (regolazione post-traduzionale) Controllo della quantità sintetizzata di enzima, variando la quantità di mRNA oppure di proteina prodotti (regolazione della trascrizione e regolazione della traduzione) Controllo dell’attività di un enzima Inibizione dovuta a cambiamenti nell’enzima di tipo: covalente (adenilazione, processamento delle proteine) non covalente (inibizione da feedback o retroazione ; isoenzimi) Inibizione da feedback dell’attività enzimatica Meccanismo tipico della regolazione di intere vie biosintetiche che coinvolgono diversi passaggi enzimatici. Il prodotto finale (es. amminoacido, nucleotide, etc.) è in grado di retroagire sul primo passaggio nella via biosintetica e di regolare la propria biosintesi (autoregolazione). Il prodotto finale inibendo il primo enzima della via inibisce tutta la via biosintetica in quanto non vengono più generati gli intermedi necessari come substrato per gli altri enzimi coinvolti nel processo. L’inibizione da feedback E’ REVERSIBILE: la sintesi riprende quando il prodotto finale si esaurisce Allosteria L’inibizione da feedback è resa possibile da una proprietà dell’enzima definita ALLOSTERIA. Un enzima allosterico possiede un sito attivo che lega il substrato e un sito allosterico dove si lega in modo reversibile l’inibitore. Quando un inibitore si lega al sito allosterico, la conformazione dell’enzima cambia ed il substrato non riesce più a legarsi efficientemente al sito attivo. Quando la concentrazione di inibitore diminuisce, si dissocia dal sito allosterico ed il sito attivo ritorna nella sua forma catalitica. Inibizione da feedback in una via biosintetica ramificata N-acetil glutamato sintetasi g-glutamil chinasi Gli enzimi allosterici sono molto importanti nelle vie biosintetiche ramificate. Prolina e arginina sono sintetizzate a partire dall’acido glutamico. Questi due aminoacidi possono controllare il primo enzima specifico (evidenziati in rosso) per la propria sintesi senza interferire con gli altri, in modo che, ad esempio l’eccesso di prolina non provochi nell’organismo una carenza di arginina. Isoenzimi ed inibizione da feedback 3-deossi-D-arabinoeptulosonato 7-fosfato ISOENZIMI: Proteine diverse che catalizzano la stessa reazione ma soggette a sistemi di regolazione indipendenti SINTESI AMINOACIDI AROMATICI IN E. COLI In E. coli 3 diversi isoenzimi con attività di DAHP sintetasi catalizzano la prima reazione della via biosintetica. Ognuno di questi enzimi è specificamente retroinibito da uno dei tre aminoacidi aromatici. In questo caso è richiesto un eccesso di tutti e tre i prodotti di reazione per spegnere completamente la sintesi di DAHP Modificazioni covalenti degli enzimi I più comuni meccanismi di modificazioni enzimatica covalenti: ADENILAZIONE: legame di un nucleotide adenosinmonofosfato (AMP (AMP)) o adenosindifosfato (ADP (ADP)) FOSFORILAZIONE: legame di un gruppo fosfato (PO (PO42-) METILAZIONE: legame di un gruppo metilico (CH (CH3) Sono processi rapidamente reversibili Il legame del gruppo modificante provoca un cambiamento conformazionale della proteina e può alterarne profondamente l’attività catalitica. La rimozione del gruppo modificante ripristina l’enzima nel suo stato attivo. Regolazione della glutamina sintetasi La glutamina sintetasi (GS) è un enzima che svolge un ruolo chiave nel metabolismo dell’azoto nella cellula. L’attività della GS può essere controllata mediante: Inibizione da feedback concertata: ad opera di 9 composti diversi (aa, prodotti del metabolismo nucleotidico). L’attività enzimatica si riduce proporzionalmente al numero di inibitori presenti: la presenza di tutti e nove gli inibitori determina l’inibizione completa Adenilazione: modificazione covalente La glutamina sintetasi e l’adenilazione PPi ATP GS GS-AMP (1-12) Glutamina AMP ADP Pi Il controllo della GS mediante modificazione covalente è determinato dalla concentrazione di glutamina e di un precursore della glutamina glutamina,, l’a l’a-chetoglutarato chetoglutarato,, un intermedio del ciclo dell’acido citrico. Quando il livello di glutamina nella cellula diminuisce diminuisce,, l’enzima che aggiunge e rimuove i gruppi AMP (enzima PII) viene modificato covalentemente e promuove la deadenilazione della GS, e di conseguenza, un aumento della sua attività. attività. Quando la concentrazione intracellulare di glutamina aumenta, la GS viene fortemente adenilata e la sua attività diminuisce. La glutamina sintetasi e l’adenilazione l’adenilazione Ogni molecola di GS contiene 12 subunità identiche identiche,, e ognuna di queste può essere adenilata.. adenilata Quando l’enzima è completamente adenilato adenilato(contiene (contiene 12 gruppi AMP), è totalmente inattivo, quando è parzialmente adenilato adenilato,, è parzialmente attivo. La glutamina sintetasi e l’adenilazione l’adenilazione I livelli di attività della GS possono essere considerati per la cellula una specie di barometro ad azoto: N GS Livelli di azoto Attività glutamina sintetasi E VICEVERSA Splicing delle proteine Anche nei procarioti esistono alcuni rari geni con introni. Recentemente sono stati individuati numerosi casi in cui l’informazione “extra” viene rimossa a livello della proteina e non dell’RNA. Il processo è stato definito “splicing delle proteine”, e la parte proteica interna che viene rimossa è stata chiamata inteina. Nel processamento della subunità A della DNA girasi di M. leprae, codificata dal gene gyrA, le sequenze fiancheggianti del polipeptide GyrA, chiamate esteine, vengono riunite per formare la forma attiva della proteina, mentre le inteine vengono scartate. Le inteine sono proteasi specifiche self-splicing (autoprocessanti). RUOLO INTEINE? La regolazione della trascrizione Controllo positivo: (operone maltosio) L’elemento di controllo, una proteina regolatrice, promuove il legame dell’RNA polimerasi determinando un aumento della sintesi di mRNA Controllo negativo: repressione (operone arginina) ed induzione (operone (operone lattosio) L’elemento di controllo, il repressore, inibisce la sintesi dell’mRNA Le proteine che legano il DNA Le interazioni tra proteine ed acidi nuclei sono essenziali per la replicazione, la trascrizione, la traduzione, nonché per la regolazione di tali processi. 1. 2. INTERAZIONI ASPECIFICHE INTERAZIONI SEQUENZASEQUENZASPECIFICA Le sequenze ripetute invertite costituiscono spesso siti specifici di legame per le proteine. Le proteine che interagiscono con il DNA sono spesso omodimeri, composte da due catene polipetidiche identiche. Su ogni catena polipeptidica c’è un dominio di legame al DNA che si inserisce nel solco maggiore e lungo l’impalcatura di fosfato e un dominio di interazione proteina-proteina che tiene insieme le due catene del dimero. Sequenza nucleotitica della regione operatore dell’operone lattosio legata dal repressore lac Struttura delle proteine che legano il DNA Classi di domini proteici di fondamentale importanza per la formazione del corretto legame con il DNA: Helix-turn Helixturn--helix (elica(elica-giro giro--elica) Zinc finger (dito di zinco) Leucine zipper (cerniera di leucine) Struttura helixhelix-turn turn--helix Il motivo strutturale helix helix--turn turn--helix possiede: Un’ elica di riconoscimento (aelica) che interagisce con il DNA Una breve sequenza di tre aa aa,, il primo dei quali è generalmente una glicina con la funzione di “girare la proteina”, ovvero di curvarla a gomito Un’elica stabilizzante, stabilizzante, che stabilizza la prima interagendo con essa idrofobicamente e partecipando alla formazione del dimero. Il riconoscimento di sequenze specifiche nel DNA avviene tramite interazioni non covalenti che comportano la formazione di legami idrogeno ed interazioni di van der Waals. Nei Batteri ci sono diverse proteine con questo motivo: tra cui molti repressori (sistemi lac e trp di E. coli; repressore del batteriofago l) Oltre 250 proteine con questo motivo si legano al DNA per regolare la trascrizione in E. coli. Modello delle substrutture proteiche che si trovano nelle proteine eucariotiche che legano il DNA Nel motivo zinc finger tra gli aa che legano lo ione Zn2+ vi sono sempre almeno due residui di cisteina (C), mentre gli altri residui sono generalmente istidine (H). Nel motivo leucine zipper i residui di leucina sono regolarmente ripetuti ogni sette aa formando una struttura simile ad una cerniera lampo. La cerniera di leucine serve a tenere unite due eliche di riconoscimento che interagiscono con il DNA Il controllo negativo della trascrizione Nei batteri sono noti diversi meccanismi per il controllo della sintesi di un enzima e tutti sono ampiamente influenzati dall’ambiente nel quale l’organismo cresce ed in particolare dalla presenza o assenza di piccole molecole specifiche (come quelle che legano il DNA) per controllare la trascrizione o più raramente la traduzione. La repressione e l’induzione rappresentano le forme più semplici di regolazione che controllano l’espressione genica a livello della trascrizione. Repressione dell’attività enzimatica La repressione enzimatica è un fenomeno ampiamente diffuso nei batteri e viene utilizzato nel controllo della sintesi di vari enzimi coinvolti nella biosintesi di aminoacidi, purine e pirimidine. In quasi tutti i casi è il prodotto finale di una particolare via biosintetica a reprimere gli enzimi della stessa. Si può ovviamente intuire quanto sia importante questo processo per il microrganismo, dato che gli permette di non sprecare energia sintetizzando enzimi non necessari. Gli enzimi coinvolti nella sintesi dell’ arginina sono sintetizzati soltanto se l’arginina è assente nel mezzo di crescita; l’aggiunta di arginina nel mezzo reprime la loro sintesi. E’da notare, come questo sia un effetto specifico, in quanto la sintesi di tutti gli altri enzimi continua regolarmente. Induzione dell’attività enzimatica Un fenomeno complementare alla repressione è l’induzione di un enzima, enzima, ovvero la sintesi di un particolare enzima solo quando è presente il suo substrato. Gli enzimi coinvolti nel catabolismo del carbonio o di altre fonti di energia sono spesso inducibili. Tipico esempio è dato dall’enzima b -galattosidasi galattosidasi,, coinvolto nella utilizzazione del lattosio, che non viene sintetizzato se lo zucchero è assente nel mezzo, mentre la sintesi inizia non appena al terreno viene aggiunto lattosio. Anche in questo caso si può facilmente comprendere il valore di tale meccanismo, dal momento che esso permette all’organismo di non sintetizzare un enzima fino a quando non gli sia necessario. L’induttore ed il corepressore EFFETTORI Induttore: sostanza che dà inizio all’induzione di un enzima Corepressore:: sostanza che reprime la sintesi di un enzima Corepressore Gli induttori e i corepressori in genere agiscono in modo indiretto, indiretto, combinandosi con specifiche proteine di regolazione che a loro volta influenzano la sintesi di mRNA mRNA.. Non tutti gli effettori sono substrato o prodotti finali degli specifici enzimi regolati. Alcuni analoghi di queste sostanze sono in grado di provocare l’induzione o la repressione di un enzima senza esserne il substrato. L’ L’isopropiltiogalattoside isopropiltiogalattoside (IPTG), per esempio, è un induttore della bgalattosidasi,, pur non essendo idrolizzato dall’enzima stesso. galattosidasi Regolazione dell’operone arginina Nel caso di un enzima reprimibile, il corepressore (per esempio l’arginina) si lega alla proteina repressore specifica (il repressore dell’arginina). Il repressore è una proteina allosterica la cui conformazione può venire modificata in seguito al legame con il corepressore. Una volta modificato, il repressore può legarsi a una regione specifica di DNA vicina al promotore del gene stesso, la regione operatore che deve il suo nome all’operone, gruppo di geni la cui espressione è sotto il controllo di un singolo promotore. Se il repressore si lega all’operatore, la sintesi di mRNA è bloccata perché l’RNA polimerasi non si può legare o non può procedere nella trascrizione e tutte le proteine codificate da quell’mRNA policistronico saranno represse. Regolazione dell’operone lattosio Anche l’induzione di un enzima può essere controllata da un repressore; in questo caso il repressore è attivo in assenza dell’induttore e capace quindi di bloccare la sintesi di mRNA mRNA.. Quando invece si aggiunge l’induttore, questo si lega al repressore inattivandolo, rimuovendo il blocco trascrizionale e permettendo la sintesi dell’mRNA dell’mRNA.. Nel caso dell’operone lac, il repressore è costituito dal repressore lac e l’induttore dall’allolattosio Dal momento che il ruolo dei repressori è inibitorio (reprimono la sintesi dell’mRNA dell’ mRNA), ), il tipo di regolazione che li coinvolge viene spesso definito controllo negativo. negativo. Regolazione dell’operone maltosio In assenza di induttore (maltosio) In presenza di induttore (maltosio) I geni necessari per l’utilizzazione del maltosio sono dispersi in diversi operoni, ognuno dei quali possiede un sito di legame dell’attivatore. Quando diversi operoni si trovano sotto il controllo primario della stessa proteina costituiscono un regulone. Il catabolismo del maltosio in E. coli è un tipico esempio di sistema sottoposto a regolazione positiva.. positiva Gli enzimi necessari all’utilizzazione del maltosio in E. coli sono sintetizzati solo dopo l’aggiunta di maltosio al mezzo. La sintesi di questi enzimi è controllata a livello trascrizionale da una proteina attivatrice che non può legarsi al DNA se prima non si è legata al maltosio, il suo induttore. Il legame dell’attivatore al DNA induce un cambiamento della sua struttura e permette alla RNA polimerasi di iniziare la trascrizione. trascrizione. La sequenza specifica che serve da sito di legame per l’attivatore si chiama sito di legame dell’attivatore. Il controllo positivo La proteina attivatrice può anche interagire direttamente con l’RNA polimerasi, sia quando il sito di legame dell’attivatore è vicino al promotore, sia quando è lontano, situazione che richiede la formazione di un’ansa per poter stabilire i contatti necessari. La proteina attivatrice, una volta legatasi al DNA, aiuta l’RNA polimerasi sia a riconoscere il promotore sia ad iniziare la trascrizione. Ad esempio, l’attivatore può indurre un cambiamento nella struttura del DNA, in genere una curvatura, permettendo così all’RNA polimerasi di stabilire contatti precisi con il promotore per iniziare la trascrizione. La regolazione globale Spesso, in risposta ad una variazione ambientale, un organismo ha bisogno di regolare simultaneamente geni diversi. I meccanismi di regolazione che rispondono a segnali ambientali sono definiti sistemi di controllo globale. globale. Nell’ambiente in cui crescono le cellule batteriche, infatti, possono essere presenti diverse fonti di carbonio utilizzabili e sarebbe uno spreco indurre, gli enzimi per il catabolismo del lattosio o del maltosio quando le cellule stanno utilizzando una fonte di carbonio più facilmente metabolizzabile. La repressione da catabolita Il problema viene risolto da uno dei sistemi di regolazione globale definito repressione da catabolita. catabolita. Nella repressione da catabolita la sintesi di numerosi enzimi viene inibita quando le cellule crescono in un terreno che contiene una fonte di energia ottimale come il glucosio. La repressione da catabolita è stata anche definita effetto glucosio,, perché questa è stata la prima sostanza per la quale si è glucosio dimostrato il fenomeno; anche se è noto che diverse fonti di carbonio determinano questo tipo di repressione. Crescita diauxica in presenza di una miscela di glucosio e lattosio Una conseguenza della repressione da catabolita è la crescita diauxica che si osserva quando due fonti di energia sono presenti contemporaneamente nel mezzo. La repressione da catabolita permette alle cellule di utilizzare subito la migliore fonte di carbonio, infatti, i batteri crescono più rapidamente in presenza di glucosio. Nella cresita diauxica, diauxica, l’organismo cresce prima utilizzando una fonte di energia (ad esempio il glucosio), e segue poi un lungo intervallo di tempo prima che la crescita ricominci utilizzando la seconda fonte (ad esempio lattosio). Quando un microrganismo cresce in presenza di una miscela di glucosio e lattosio, finchè il glucosio è presente nel mezzo, la b galattosidasi,, codificata dal gene lacZ galattosidasi dell’operone lattosio e responsabile della utilizzazione di questo zucchero, non viene sintetizzata ed il microrganismo utilizza solo il glucosio lasciando intatto il lattosio. Quando il glucosio è esaurito, la repressione da catabolita finisce e dopo un certo intervallo si osserva sintesi di bgalattosidasi e inizia la crescita basata sul lattosio come fonte di carbonio. La repressione da catabolita La repressione da catabolita si basa sul controllo della trascrizione mediato da un attivatore ed è quindi un tipo di controllo positivo. Nel caso di enzimi reprimibili da parte del catabolita, il legame della RNA polimerasi al DNA avviene solo quando si è legata un’altra proteina, chiamata attivatore proteico del catabolita (CAP), che a sua volta può legarsi al DNA solo se prima si è legato l’AMP ciclico, un elemento chiave in numerosi sistemi controllo. L’entrata del glucosio nella cellula inibisce la sintesi dell’AMP ciclico e stimola il suo trasporto al di fuori della cellula con riduzione della sua concentrazione intracellulare ed inibizione del legame della RNA polimerasi ai promotori sensibili a questo tipo di controllo. La repressione da catabolita, quindi, è in realtà dovuta alla mancanza di AMP ciclico nella cellula e può essere superata aggiungendo tale composto al terreno. terreno. La repressione da catabolita Nonostante questo tipo di regolazione possa sembrare un semplice sistema di controllo positivo, ognuno degli operoni controllati da CAP è anche soggetto a regolazione da parte dei suoi regolatori specifici. Poiché la repressione da catabolita modula l’espressione di sistemi di regolazione non correlati essa rappresenta uno dei principali esempi di regolazione globale. Finchè è presente glucosio viene impedita l’espressione di tutti gli altri operoni catabolici soggetti a questo elemento di controllo globale. Affinché la trascrizione possa avvenire, devono essere rispettate due condizioni: 1. il ivello di AMP ciclico deve essere abbastanza elevato, in modo da permettere alla proteina CAP di legarsi al sito di legame specifico (controllo positivo); 2. deve essere presente un induttore, come il lattosio, in modo che il repressore del lattosio non blocchi la trascrizione legandosi all’operatore (controllo negativo). Regolazione dell’operone lattosio Il primo gene strutturale di questo operone , lacZ, codifica l’enzima b-galattosidasi che degrada il lattosio. L’operone contiene altri due geni (lacY e lacA) coinvolti nel metabolismo del lattosio. In figura è mostrata la regione regolatrice dell’operone. I due siti dell’operatore (dove si lega il repressore lac) sono sequenze ripetute invertite. Sequenze ripetute invertite si ritrovano anche nel sito di legame di CAP. E’ indicata la posizione delle sequenze -35 e la Pribnow box che fanno parte del promotore. Viene indicata anche la posizione di due sequenze cruciali dell’mRNA (Shine-Dalgarno ed il codone d’inizio). L’attenuazione dell’operone triptofano in E. coli Questo operone è soggetto in realtà a diversi tipi di regolazione: il primo enzima della via, l’antranilato sintetasi è infatti, sottoposto all’inibizione da feedback da parte del triptofano, triptofano, ed inoltre la trascrizione dell’operone è sotto il controllo del repressore del triptofano. triptofano. L’operone triptofano contiene i geni strutturali per cinque proteine della via biosintetica del triptofano, oltre al promotore e alle sequenze regolatrici localizzate all’inizio dell’operone. Oltre al promotore P e all’operatore O c’è una sequenza leader L che codifica un polipeptide contenente residui di triptofano ripetuti in tandem vicino alla sua estremità terminale e operante come attenuatore. Se nella cellula è il triptofano è abbondante, il peptide leader verrà sintetizzato, determinando la terminazione della trascrizione del restante operone trp, che comprende i geni strutturali per gli enzimi biosintetici; se invece il triptofano è scarso, il peptide leader, che è ricco in triptofano, non sarà prodotto, e la trascrizione della restante parte dell’operone potrà aver luogo. L’attenuazione dell’operone triptofano Per spiegare questo fenomeno bisogna tener conto che nelle cellule procariotiche i processi di traduzione e trascrizione avvengono simultaneamente: mentre la trascrizione di regioni a valle è ancora in corso, è già iniziata la traduzione delle regioni trascritte. L’attenuazione ha luogo perché una parte dell’mRNA neosintetizzato si ripiega a formare un’ansa a doppia elica, grazie alla presenza di sequenze complementari, seguita da una sequenza di uracili che segnalano alla RNA polimerasi di cessare l’attività trascrizionale. Se il triptofano è presente in abbondanza, il ribosoma potrà tradurre l’intero peptide leader (codificato dalle regioni 1 e 2), e così la regione 2 non potrà appaiarsi con la regione 3. Le regioni 3 e 4 si appaieranno a formare una struttura ansaansa-stelo, che costituirà un sito di pausa della trascrizione. Poi trovandosi a valle di questa sequenza un tratto ricco in uracile, si avrà la terminazione. L’attenuazione dell’operone triptofano Se invece il triptofano è scarso, il ribosoma rallenterà in corrispondenza del codone triptofano; la sosta del ribosoma in questa posizione permetterà la formazione di una struttura ansaansastelo alternativa (regione 22-regione 3) che non rappresenta un segnale di terminazione e previene efficacemente la formazione del vero terminatore. L’RNA polimerasi oltrepasserà il sito di terminazione che non si è correttamente ripiegato e inizierà la trascrizione dei geni strutturali del triptofano I sistemi di regolazione a due componenti Molti dei sistemi di regolazione tramite i quali i batteri sono in grado di rilevare i segnali ambientali e fornire una risposta sono costituiti da due componeneti. Un sistema classico a due componenti è costituito da: 1. Una proteina sensore (chinasi sensore), locaizzata nelle membrana cellulare e in grado di autofosforiularsi in risposta a un segnale ambienetale 2. Una proteina regolatrice della risposta, generalmente una proteina di legame al DNA che regola la trascrizione in modo + o – a seconda del sistema La regolazione della chemiotassi Il grado di metilazione delle proteine MCP controlla la loro capacità di rispondere ad una sostanza chimica e determina il processo dell’ADATTAMENTO.
Scaricare