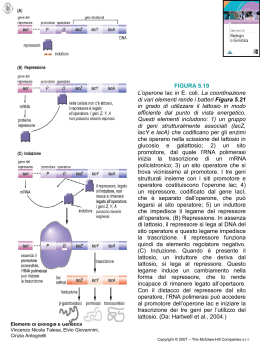
La Matematica delle Cellule Corno Andrea V BLT Anno Scolastico 2006/2007 Perché i Modelli Matematici per la Biologia? • Strumenti di analisi di dati • Elaborazioni di “leggi”(che possono essere predittive) • Modellizzazione (dinamica) della realtà • Costruzione e formalizzazione di teorie La Cellula della Biologia Classica Il dogma principale della cellula: DNA mRNA Proteine 1. Duplicazione di DNA 2. Trascrizione di mRNA 3. Traduzione in amminoacidi Duplicazione di DNA La Cellula della Biologia Cellulare Computazionale • Ramo sperimentale della Biologia Moderna • Studia e analizza le diverse interazioni tra le molecole biologiche (soprattutto le proteine) • Capire come le proteine interagiscano tra di loro, quali siano le connessioni che stabiliscono e come esse variano in funzione degli stimoli ambientali aiuta a comprendere meglio il funzionamento di una cellula nel suo insieme. Nuova Biologia, nuovo Dogma • Il funzionamento di una cellula è dato dall’insieme delle interazioni tra macromolecole. • Diverse interazioni tra macromolecole costituiscono una rete, definita NETWORK Il network nel dettaglio Gli elementi di un network sono: Nodi Rappresentano le macromolecole coinvolte nell’insieme di interazioni del network Frecce Indicano le interazioni presenti tra le macromolecole: • reazioni di attivazione () • reazioni di inibizione (--| ) Da Fenomeno Biologico… a Modello Matematico 1. Analisi del fenomeno biologico da studiare 2. Rappresentazione del sistema in esame in un modello matematico 3. Simulazione del funzionamento del modello matematico 4. Predizioni e/o conclusioni Operone Lac in termini computazionali Analisi Traduzione Simulazione Conclusioni Approfondimento... Controllo dell’espressione genica • Espressione genica: traduzione dell’informazione genica dai geni alle proteine (dal genotipo al fenotipo) • Il controllo di tale processo permette alle cellule di specializzarsi, producendo determinate proteine solo quando necessarie • Il controllo è attuato da un insieme di relazioni tra macromolecole, e ciò è oggetto di studio della Biologia Cellulare Computazionale 1. Analisi fenomeno biologico Operone: insieme specifico di geni che vengono regolati insieme, tramite processi di attivazione/inibizione. Uno tra i primi operoni studiati nella storia della Biologia, da Jacques Monod, fu l’Operone Lac (o Operone Lattosio) che fornisce enzimi per l’uso del lattosio da parte del batterio E. Coli. Struttura schematica Operone-Lac Gene regolatore Geni per l’utilizzo Promotore Operatore del lattosio Sequenza di nucleotidi, daaenzimi cui Geni Sequenza corrispondenti nucleotidica di nucleotidi, a riconosciuta tre cuiverrà si sintetizzato il regolatrice repressore importanti legherà dalla la per proteina RNA-polimerasi funzioni strettamente attiva correlate con il lattosio (per esempio, il trasporto) Senza lattosio… NESSUNA SINTESI DI ENZIMI Con lattosio… SINTESI DI ENZIMI (in minime quantità) Con lattosio, ma carenza di glucosio… SINTESI DI ENZIMI (in notevoli quantità) 2. Creazione del modello matematico Dall’osservazione del fenomeno biologico, è possibile definire i “ruoli” dei nodi del futuro network: – Il repressore inibisce la produzione degli enzimi necessari – Il lattosio inibisce il repressore, favorendo quindi la produzione degli enzimi – In carenza di glucosio, viene attivato un complesso che aumenta la produzione di enzimi Valori basali Valori massimi Lattosio REP Enzima Glucosio cAMP + Crp 0 or 1 1 1 0 or 1 1 1 1 2 1 1 3. Simulazione Osserviamo come funziona l’operone Lac in termini computazionali: Casi Lattosio Glucosio Enzima a 0 0 0 b 0 1 0 c 1 1 1 d 1 0 2 Caso a) abcde a = Lattosio ; b = repressore ; c = enzima d = glucosio ; e = cAMP+Crp Caso b) abcde a = Lattosio ; b = repressore ; c = enzima d = glucosio ; e = cAMP+Crp Caso c) abcde a = Lattosio ; b = repressore ; c = enzima d = glucosio ; e = cAMP+Crp Caso d) abcde a = Lattosio ; b = repressore ; c = enzima d = glucosio ; e = cAMP+Crp 4. Conclusioni • In ogni caso, i valori della tavola delle verità compaiono nel rispettivo stato stabile; si e’ dimostrato, inoltre, che e’ necessario almeno il lattosio per produrre l’enzima. • Sono stati riscontrati stati stabili • Non sono state riscontrate oscillazioni tra diversi stati Possiamo fidarci del nuovo dogma? I vantaggi: • Capire come agisce una determinata macromolecola all’interno di un sistema di regolazione può essere utile per la creazione di farmaci specifici, o per capire come si sviluppano determinate patologie (per esempio i tumori) • Il binomio “Biologia computazionale – laboratorio” possiede un notevole potenziale, se sfruttato al meglio Possiamo fidarci del nuovo dogma? Eventuali difficoltà: • Numero elevato di variabili necessità di semplificazione • Sostituzione del modello alla realtà, con rischio di falsificazione • Estrapolazione dal semplice al complesso • Mancanza di corretta pianificazione degli esperimenti in funzione della modellazione La Biologia Cellulare Computazionale ha trovato impiego in laboratori internazionali di ricerca: in Gran Bretagna, in Svizzera, negli USA e anche in Italia (presso IFOM – Istituto FIRC di Oncologia Molecolare di Milano). Nuove sfide e nuove conoscenze attendono la Biologia sperimentale in questo campo. Si prospetta, quindi, un futuro innovato da nuove scoperte e da nuovi saperi, un futuro che la Ricerca costruisce giorno dopo giorno. Fonti: • • • • • • • • • • Kaplan D. , Glass L. – Understanding Nonlinear Dynamics – SpringerVerlag Tyson J. J. , Chen K. , Novak B. – Network dynamics and cell physiology (2001) Gonzales A. , Naldi A. , Sánchez L. , Thieffry D. , Chaouiya C. – GINsim: A software suite for the qualitative modelling, simulation and analysis of regulatory networks (2005) Sánchez L. , Thieffry D. – Dynamical modelling of pattern formation during embryonic development (2003) Thieffry D. , Thomas R. – Qualitative analysis of gene networks Christopher P. Fall, Eric S. Marland, John M. Wagner, John J. Tyson – Computational Cell Biology Enciclopedia on-line Wikipedia (it.wikipedia.org) Il genoma umano – DVD, supporto integrativo di Le Scienze Federico Tibone e BioMEDIA Associates – Biologia della Cellula (cd-rom) – 2001 – Zanichelli Editore Sito web IFOM - Istituto FIRC di Oncologia Molecolare (www.ifom-firc.it)
Scaricare