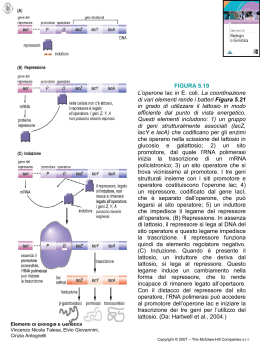
Struttura dei geni batterici La replicazione è semiconservativa QuickTime™ e un decompressore TIFF (Non compresso) sono necessari per visualizzare quest'immagine. La replicazione è semidiscontinua QuickTime™ e un decompressore TIFF (Non compresso) sono necessari per visualizzare quest'immagine. La replicazione è bidirezionale QuickTime™ e un decompressore TIFF (Non compresso) sono necessari per visualizzare quest'immagine. (Cairns, 1961) Struttura dei geni batterici RNA polimerasi l’elica 3’-5’ del DNA funge da stampo per la sintesi dell’RNA (5’-3’) nucleotidi inseriti in base alle regole della complementarità RNA: ribosio invece del desossiribosio e U al posto di T la sequenza “codificante” del DNA è uguale a quella del messaggero promotore: sequenza presente a monte di tutti i geni, riconosciuta dal fattore sigma dell’RNA polimerasi, necessaria per l’inizio della trascrizione. La sequenza è conservata promotori riconosciuti dal fattore sigma-70 di E. coli mutazioni “down”: diminuisce l’efficienza del promotore TTGACa TAtAaT lettera maiuscola = basi più conservate (presenti in quella posizione rispetto al +1 in quasi tutti i casi lettera minuscola = basi presenti in molti casi ma non sempre regolazione dell’espressione genica mediante l’intervento di fattori sigma alternativi geni “heat-shock”: CCCCCC - 15/17 basi - CCCC promotore riconosciuto dal fattore sigma32 attivato solo in caso di aumento della temperatura. I geni che hanno questo promotore vengono espressi (trascritti) SOLO IN PRESENZA DI QUESTO SPECIFICO FATTORE SIGMA geni “spo” in B. subtilis sono silenti e vengono trascritti solo durante la sporulazione per la presenza di fattori sigma specificamente attivati in sporulazione SEQUENZE PALINDROMICHE: sequenze ripetute e invertite 5’- TGCA- 3’ 3’- ACGT - 5’ lette allo stesso modo su entrambi i filamenti del DNA (nella stessa direzione) rispetto all’asse di simmetria le sequenze sono specularmente complementari 5’-ATCCGGAT-3’ 3’-TAGGCCTA-5’ palindromi perfette = palindrome perfetta a 8 coppie di basi siti di restrizione (endonucleasi specifiche) 5’-ATCCAAGTACGGAT-3’ PALINDROME IMPERFETTA 3’-TAGGTTCATGCCTA-5’ (terminatori della trascrizione) QuickTime™ e un decompressore TIFF (LZW) sono necessari per visualizzare quest'immagine. Terminatore forte o rho-indipendente: -struttura secondaria stabile e forte per la presenza di molte G e C nello “stem” - sequenza di tante U al 3’ della struttura secondaria (che facilita la separazione dell’ibrido DNA/RNA) QuickTime™ e un decompressore TIFF (LZW) sono necessari per visualizzare quest'immagine. Terminatore forte o rho-indipendente Terminatore debole o rho-dipendente Terminatore debole o rho-dipendente Ordinare i seguenti promotori in base alla loro forza partendo da quello più forte: a) TTAACC ...........17 basi............CCTAAG b) TTGACC ................".................TATATT c) TTGACA.................".................TATAAT d) CTGACA.................".................CATAAT sequenza consenso: TTGACa....................TAtAaT Indicare quale delle seguenti palindromi è imperfetta ATGCAT TACGTA ATGCTA TACGAT TTACAACCCTGTAA AATGTTGGGACATT Scrivere la sequenza di un ipotetico terminatore forte riportando la struttura che esso assume sull'RNA 5’- tcCACCGGCTGCCGaagtgtttaCGGCAGCCGGTGtttttttt - 3’ 3’-agGTGGCCGACGGCttcacaaatGCCGTCGGCCACaaaaa..-5’ g t g a a t t t a G C C G C G G C T A C G G C G C C G C G A T tcC GUUUUUU La sequenza sotto riportata rappresenta un terminatore della trascrizione. 1) Indicare se si tratta di un terminatore forte o debole motivando la risposta 2) riportare la struttura che tale sequenza assume una volta trascritta GAACTCTAGCCCGGCTTAGCCxxxxxxxxxGGCTAAGCCGGGCTTTATATTTT CTTGAGATCGGGCCGAATCGGxxxxxxxxxCCGATTCGGCCCGAAATATAAAA Struttura dei geni batterici rRNA tRNA attivazione degli aminoacidi formazione dell’aminoacil-AMP QuickTime™ e un decompressore TIFF (LZW) sono necessari per visualizzare quest'immagine. N-formil-metionil-tRNA (tRNA iniziatore nei batteri) (archea metionil-tRNA) Sintesi proteica: fasi di inizio •Interazione sub. minore del ribosoma con mRNA (complementarità rRNA16S e sequenza SD) •Legame del tRNA iniziatore (N-formil-metionina) alla tripletta di inizio AUG • legame sub. maggiore del ribosoma 50S Fattori di inizio (IF-1, IF-2, IF3) GTP per fornire energia tRNA iniziatore Interazione sub minore + mRNA (16S + sequenza SD) SEQUENZA DI SHINE-DALGARNO: legame con il 16S della sub. minore del ribosoma rRNA 16S SD Tripletta di inizio AUG Sequenza codificante Ribosoma: sito A = sito “accettore” (interazione con tRNA carico) Sito “P” = formazione del legame peptidico con l’aa “nuovo” Sito “E” = sito di uscita del tRNA “scarico” -Legame aa2-tRNA al sito A -Legame aa1 (sito P) con aa2 (sito A) Legame peptidico tra il gruppo NH2 dell’aa “nuovo” (sito A) e il gruppo COOH dell’aa precedente (sito P). Il peptide si accresce di un aa. Il tRNA del sito P si “scarica” e si allontana (sito E). Il ribosoma trasloca e il tRNA che porta la catena di aa si troverà nel sito P. Il sito A è vuoto e presenta una nuova tripletta per il legame con il corrispondente tRNA carico La traslocazione è catalizzata dall’idrolisi del GTP. La formazione del legame peptidico non richiede altra energia perche il legame tra tRNA e aa è ad alta energia TERMINAZIONE DELLA SINTESI PROTEICA UAA, UGA, UAG = codoni di stop (non codificanti) RF = fattori di rilascio Accoppiamento trascrizionetraduzione (procarioti) RNA + ribosomi QuickTime™ e un decompressore TIFF (LZW) sono necessari per visualizzare quest'immagine. DNA QuickTime™ e un decompressore TIFF (LZW) sono necessari per visualizzare quest'immagine. 1 prodotto 2 o più prodotti gene operone operone: insieme di più sequenze codificanti controllate da un unico promotore promotore SD SD SD Regolazione dei geni batterici • a livello trascrizionale, con un controllo che può riguardare l’inizio della sintesi dell’mRNA o la terminazione precoce del processo • a livello traduzionale, con meccanismi che riguardano principalmente la fase di inizio del processo geni regolati negativamente espressione inducibile REPRESSORE sito operatore geni regolati negativamente espressione reprimibile REPRESSORE geni regolati positivamente Esempio: l'operone lacZYA ENZIMI PER L’UTILIZZO DEL LATTOSIO QuickTime™ e un decompressore TIFF (LZW) sono necessari per visualizzare quest'immagine. espressione inducibile (LATTOSIO = INDUTTORE) RBS REPRESSORE (LacI) REPRESSORE INATTIVO INDUTTORE (lattosio) OPERONE LAC TRASCRITTO Crescita con glucosio e lattosio La presenza del glucosio inibisce l’espressione dell’operone glucosio lacZYA non si esprime glucosio + lattosio lacZYA si esprime a livelli molto bassi lattosio lacZYA si esprime a livelli alti la presenza di glucosio ha un effetto negativo sull'espressione di lacZYA + glucosio = bassi livelli intracellulari di cAMP - glucosio = alti livelli intracellulari di cAMP lacZYA necessita di un attivatore = proteina CAP (o CRP) legata al cAMP RBS GLUCOSIO ASSENTE Il P destinato al glucosio attiva l’adenilato ciclasi (alti livelli di cAMP) CRP ATTIVO AMP CICLICO REPRESSORE ATTIVO (-lattosio) PROTEINA CAP REPRESSORE INATTIVO (+lattosio) RNA pol. -LATTOSIO: Repressore attivo Trascrizione repressa AMP CICLICO REPRESSORE ATTIVO (-lattosio) PROTEINA CAP REPRESSORE INATTIVO (+lattosio) RNA pol. +LATTOSIO -GLUCOSIO : Repressore inattivo cAMP alti livelli Proteina CAP attiva Trascrizione alta (utilizzo del lattosio) RNA pol. AMP CICLICO REPRESSORE attivo (-lattosio) PROTEINA CAP REPRESSORE INATTIVO (+lattosio) +LATTOSIO + GLUCOSIO Repressore inattivo cAMP basso Proteina CAP inattiva Trascrizione bassa (viene usato prima tutto il glucosio) AMP CICLICO REPRESSORE ATTIVO (-lattosio) PROTEINA CAP REPRESSORE INATTIVO (+lattosio) RNA pol. - lattosio: REPRESSORE ATTIVO, NO TRASCRIZIONE + lattosio: REPRESSORE INATTIVO, SI TRASCRIZIONE +glucosio:NO AMPc, NO ATTIVATORE, TRASCRIZIONE BASSA (utilizzo preferenziale del glucosio) - glucosio: SI AMPc, SI ATTIVATORE, TRASCRIZIONE ALTA (utilizzo del lattosio) + Glucosio P EIII Adenilato ciclasi Glucosio Glucosio-P cAMP
Scaricare