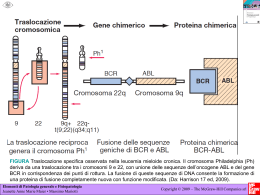
Organizzazione del genoma umano Genoma nucleare 3200Mb Geni e sequenze annesse 1200Mb Geni 48Mb Pseudogeni DNA Intergenico 2000Mb Sequenze correlate 1152Mb frammenti genici LINE 640Mb Ripetizioni Intersperse 1400Mb Introni, UTR Microsatelliti 90Mb Elementi LTR 250Mb SINE 420Mb Trasposoni a DNA 90b Altre regioni intersperse 600Mb Varie 510Mb Organizzazione del genoma umano Genoma nucleare 3200Mb DNA Intergenico 2000Mb Ripetizioni Intersperse 1400Mb Microsatelliti 90Mb LINE 640Mb Elementi LTR 250Mb SINE 420Mb Trasposoni a DNA 90b Altre regioni intersperse 600Mb Varie 510Mb Esistono diversi modi di classificazione delle sequenze genomiche: 1) Numero di copie ➣ Sequenze a copia singola ➣ Sequenze mediamente ripetute ➣ Sequenze altamente ripetute 2) Velocità evolutiva ➣ Sequenze ad alta velocità evolutiva ➣ Sequenze ad bassa velocità evolutiva 3) Capacità Codificante➣ Sequenze codificanti ➣ Sequenze non codificanti 4) Plasticità ➣ Sequenze con alto grado di plasticità ➣ Sequenze con basso grado di plasticità Esistono diversi modi di classificazione delle sequenze genomiche: 1) Numero di copie ➣ Sequenze a copia singola ➣ Sequenze mediamente ripetute ➣ Sequenze altamente ripetute Costituite da sequenze geniche singole o poco ripetute 2) Velocità evolutiva ➣ Sequenze ad alta velocità evolutiva ➣ Sequenze ad bassa velocità evolutiva 3) Capacità Codificante➣ Sequenze codificanti ➣ Sequenze non codificanti 4) Plasticità ➣ Sequenze con alto grado di plasticità ➣ Sequenze con basso grado di plasticità Esistono diversi modi di classificazione delle sequenze genomiche: 1) Numero di copie ➣ Sequenze a copia singola ➣ Sequenze mediamente ripetute ➣ Sequenze altamente ripetute da elementi da geni ripetuti, in tandem o 2)Costituite Velocità evolutiva ➣ trasponibili Sequenze ade alta velocità evolutiva interspersi (rDNA e tDNA) ➣ Sequenze ad bassa velocità evolutiva 3) Capacità Codificante➣ Sequenze codificanti ➣ Sequenze non codificanti 4) Plasticità ➣ Sequenze con alto grado di plasticità ➣ Sequenze con basso grado di plasticità Esistono diversi modi di classificazione delle sequenze genomiche: 1) Numero di copie ➣ Sequenze a copia singola ➣ Sequenze mediamente ripetute ➣ Sequenze altamente ripetute 2)Costituite Velocità evolutiva ➣ Sequenze altatandem velocità aevolutiva da sequenze ripetutead in formare DNA ➣ Sequenze ad bassa velocità evolutiva satellite: a) DNA microsatellite triplette 3)b)Capacità Codificante➣ satellite Sequenzetelomerico;CNV; codificanti DNA minisatellite ➣ Sequenze non codificanti c) DNA satellite 4) Plasticità ➣ Sequenze con alto grado di plasticità ➣ Sequenze con basso grado di plasticità od intersperso come le sequenze Sine e Line Microsatelliti e Minisatelliti I microsatelliti sono costituiti da unità di ripetizione lunghe da 1 a 10 pb, ripetute in tandem 10-20 volte, che formano raggruppamenti molto corti, <150pb, di tipo (A)n, (CA)n, (CGG)n, ecc. Sono anche detti SSR (simple sequence repeats). Le ripetizioni possono essere perfette o presentare piccole variazioni. I minisatelliti sono costituiti da unità più lunghe (da 11 a 100pb) ripetute in tandem 20-50 volte che formano raggruppamenti di lunghezza fino a 20kb Gli SSR costituiscono circa il 3% del genoma umano. Sono molto importanti nello studio delle malattie genetiche in quanto mostrano un elevato grado di polimorfismo nella popolazione umana. 7 Gli SSR possono formarsi attraverso un meccanismo di scivolamento della replicazione Gli SSR sono presenti con una frequenza di almeno uno ogni circa 2 kb del genoma. • Si originano da vari meccanismi tra cui il più importante è lo scivolamento della DNA polimerasi durante la replicazione. Microsatelliti: Genetic Fingerprint Caratteristiche degli SSRs • Polimorfismo di lunghezza: DNA fingerprinting • Spesso adoperati come marcatori genetici per la mappatura di geni associati a patologie. Microsatelliti e malattie genetiche I microsatelliti, ed in particolare le ripetizioni di triplette sono associati a varie malattie genetiche (vedi Cummings and Zoghbi, ARGHG 2000). 10 Metodi Bioinformatici per l’individuazione di SSR http://tandem.bu.edu/trf/trf.h tml Esistono diversi modi di classificazione delle sequenze genomiche: 1) Numero di copie ➣ Sequenze a copia singola ➣ Sequenze mediamente ripetute ➣ Sequenze altamente ripetute 2) Velocità evolutiva ➣ Sequenze ad alta velocità evolutiva ➣ Sequenze ad bassa velocità evolutiva Costituite da sequenze non codificanti libere di evolvere poiché, 3)apparentemente Capacità Codificante➣ prive di Sequenze funzione codificanti ➣ Sequenze non codificanti 4) Plasticità ➣ Sequenze con alto grado di plasticità ➣ Sequenze con basso grado di plasticità Esistono diversi modi di classificazione delle sequenze genomiche: 1) Numero di copie ➣ Sequenze a copia singola ➣ Sequenze mediamente ripetute ➣ Sequenze altamente ripetute 2) Velocità evolutiva ➣ Sequenze ad alta velocità evolutiva ➣ Sequenze ad bassa velocità evolutiva 3) Capacità Codificante➣ Sequenze codificanti Costituite da sequenze codificanti sottoposte a pressione evolutiva ➣ Sequenze non codificanti 4) Plasticità ➣ Sequenze con alto grado di plasticità ➣ Sequenze con basso grado di plasticità Esistono diversi modi di classificazione delle sequenze genomiche: 1) Numero di copie ➣ Sequenze a copia singola ➣ Sequenze mediamente ripetute ➣ Sequenze altamente ripetute 2) Velocità evolutiva ➣ Sequenze ad alta velocità evolutiva ➣ Sequenze ad bassa velocità evolutiva 3) Capacità Codificante➣ Sequenze codificanti ➣ Sequenze non codificanti 4) Plasticità ➣ Sequenze con alto grado di plasticità ➣ Sequenze con basso grado di plasticità Esistono diversi modi di classificazione delle sequenze genomiche: 1) Numero di copie ➣ Sequenze a copia singola ➣ Sequenze mediamente ripetute ➣ Sequenze altamente ripetute 2) Velocità evolutiva ➣ Sequenze ad alta velocità evolutiva ➣ Sequenze ad bassa velocità evolutiva 3) Capacità Codificante➣ Sequenze codificanti ➣ Sequenze non codificanti 4) Plasticità ➣ Sequenze con alto grado di plasticità ➣ Sequenze con basso grado di plasticità Esistono diversi modi di classificazione delle sequenze genomiche: 1) Numero di copie ➣ Sequenze a copia singola ➣ Sequenze mediamente ripetute ➣ Sequenze altamente ripetute 2) Velocità evolutiva ➣ Sequenze ad alta velocità evolutiva ➣ Sequenze ad bassa velocità evolutiva 3) Capacità Codificante➣ Sequenze codificanti ➣ Sequenze non codificanti 4) Plasticità ➣ Sequenze con alto grado di plasticità ➣ Sequenze con basso grado di plasticità Con il termine di plasticità del genoma si intende la sua capacità di modificarsi senza mutare Ripetizioni in tandem: Duplicazioni segmentali Le duplicazioni segmentali sono ripetizioni di segmenti genomici comunemente osservate in genomi di animali e piante, non riconducibili a elementi trasponibili, di lunghezza ≥10 kbp e identità ≥90%. Queste possono essere anche molto estese come nel cromosoma Y umano che presenta un blocco ripetuto di 1.45 Mbp. La porzione eucromatica del genoma umano contiene circa il 5,3% di duplicazioni segmentali che sono classificate in : -duplicazioni inter-cromosomiche, segmenti che si sono duplicati tra cromosomi non omologhi; -duplicazioni intra-cromosomiche, segmenti duplicati all’interno di un particolare cromosoma. Duplicazione segmentali Gap Ripetizioni in tandem: Duplicazioni segmentali Le duplicazioni segmentali sono localizzate in prevalenza nelle regioni adiacenti ai centromeri, mentre raramente coinvolgono le regioni più distali di ciascun braccio dei cromosomi. Sono di grande interesse in campo medico in quanto sono regioni che mostrano una particolare predisposizione a riarrangiamenti con conseguenti effetti fenotipici. Sono note varie malattie genetiche correlate a queste regioni (es. sindrome DiGeorge, Charcot-Marie-Tooth, etc.). Copy Number Variations (CNV) Le duplicazioni segmentali sono una importante fonte di variabilità genetica tra individui nella popolazione umana. Dato che si estendono su più geni, portano alla variazione del numero di copie di determinati geni tra diversi individui. E’ oggi possibile fare studi su larga scala di queste varianti strutturali del genoma mediante specifiche piattaforme di microarray. Uno studio su 270 individui di 4 popolazioni ha identificato circa 1500 CNVs. I CNV sono responsabili di variazioni del livello di espressione genica e possono essere associati a specifici tratti fenotipici e/o patologici (es. HIV, cancro della mammella, autismo, malattie auto-immuni). Il DNA Il DNA in cellula non è mai “nudo”, a sempre complessato con una serie di proteine a formare la cosiddetta CROMATINA Compattamento DNA doppia elica 2 nm 0 Collana di nucleosomi 11 nm ~3 Solenoide 30 nm ~6 Anse su armatura 300 nm ~24 Cromatidio (Rosette) 700 nm ~20 ~8600 Telomere IL CROMOSOMA EUCARIOTICO Origin of replication Origin of replication Kinetochore proteins Centromere Origin of replication SONO MOLECOLE DI DNA LUNGHE E LINEARI UNA MOLECOLA DÌ DNA PUO’ ESSERE CONSIDERATA UN CROMOSOMA SE HA: ORIGINE DÌ REPLICAZIONE (+ DÌ UNA) CENTOMERO (UNO) Origin of replication Genes Repetitive sequences Telomere TELOMERO (DUE) Il CENTROMERO è un complesso locus genetico, cis-agente, responsabile della corretta segregazione dei cromosomi in mitosi ed in meiosi. Esso rappresenta: 1) il sito di attacco dei microtubuli del fuso; 2) La regione di coesione tra cromatidi fratelli; 3) Il motore meccano-chimico responsabile del movimento dei cromosomi. Dal un punto di vista citologico il CENTROMERO appare come un dominio strutturale distinto nel cromosoma metafasico (Costrizione Primaria) dovuta alla mancanza dell’ultimo passaggio di superavvolgimento della cromatina. Il CENTROMERO è composto da almeno tre distinti domini strutturali: 1) Dominio del Cinetocoro (lungo la superficie esterna); 2)Dominio Centrale; 3)Dominio di Appaiamento (lungo la superficie interna). Dominio del Cinetocoro E’ una struttura proteica formata da almeno tre distinte regioni: 1) disco interno (giustapposto al dominio centrale) 2) disco centrale 3) disco esterno La Corona Fibrosa, è visibile sul disco esterno quando i microtubuli sono assenti Organizzazione del DNA satellite ai centromeri The DNA sequence of centromeres differs between species, but the presence and function of CENPA and its homologues at kinetochores is highly conserved. a | S. cerevisiae centromere function depends on a region that contains three conserved elements (I, II, III), to which Cse4 localizes. B| S. pombe centromeres contain a unique central core, which Cnp1 localizes to, flanked by conserved inverted inner and outer repeats. C|The minichromosome Dp1187, with the only defined Drosophila melanogaster centromere, consists of a core of 5-bp satellites and transposons, flanked by other repetitive DNA (red). D|Human centromeres consist of alpha-satellite DNA tandemly arranged into higher-order repeats. CENPA localizes to a portion of these arrays. CENPB (red), a centromeric alpha-satellite-binding protein, is present at both centromeres. CENPA (green) locates to only the active centromere of the dicentric chromosome. e|Caenorhabditis elegans kinetochores assemble along the length of each chromosome. Right, at metaphase, the centromeric histone HCP-3 (green) is present on the poleward-facing side of chromatids. The schematics show the location of different centromere-region proteins with respect to the DNA sequence. a | Prometaphase Caenorhabditis elegans holocentric chromosomes contain inner and outer kinetochore proteins distributed in clusters. b | Drosophila melanogaster centromere proteins. c | Schizosaccharomyces pombe centromere proteins bind either the inner centromere (the centromere core and inner repeats), or the flanking heterochromatin. a|The S. cerevisiae centromere probably consists of a single, Cse4containing nucleosome that gives rise to the kinetochore. b|The S. pombe centromere consists of multiple Cnp1-containing nucleosomes that are flanked by heterochromatin. The invertedrepeat structure of the centromere-flanking regions indicate that the centromere region might form a stem–loop structure97, perhaps stabilized by heterochromatic proteins, such as homologues of HP1 and SU(VAR)3-9. c|The larger and more complex centromeres of Drosophila melanogaster and humans possibly contain repeats of similar 'loops', or related higher-order structures, that are concentrated at specific chromosomal regions. Because tandem, rather than inverted, repeats exist in fly and human centromeric DNA, they might not form stem–loop structures; the DNA might, for example, spiral through a centromeric higher-order structure. Flanking heterochromatin might form a boundary that limits the spreading of centromeric chromatin in monocentric chromosomes. Such boundaries might distinguish monocentric from holocentric centromeres. d|C. elegans holocentric chromosomes might contain more repeat units at multiple sites, covering most of the chromosome. In these models, the higher-order structure of centromeric chromatin is conserved, and might be required to 'present' the centromeric chromatin to the outer face of condensed. Telomere IL CROMOSOMA EUCARIOTICO Origin of replication Origin of replication Kinetochore proteins Centromere Origin of replication SONO MOLECOLE DI DNA LUNGHE E LINEARI UNA MOLECOLA DI DNA PUO’ ESSERE CONSIDERATA UN CROMOSOMA SE HA: ORIGINE DI REPLICAZIONE (+ DI UNA) CENTOMERO (UNO) Origin of replication Genes Repetitive sequences Telomere TELOMERO (DUE) Il TELOMERO Il telomero è la regione terminale del cromosoma composta di DNA altamente ripetuto. Il telomero ha la duplice funzione: 1) funge da estremità vera del cromosoma lineare; 2) si oppone all’accorciamento del cromosoma stesso come risultato dei cicli replicativi del genoma. Contrariamente alle sequenze ripetute (DNA satellite) centromeriche, che sono estremamente variabili da specie a specie, le sequenze ripetute che formano il telomero hanno una sequenza molto simile tra loro L’enzima telomerasi è in grado di catalizzare la reduplicazione dei telomeri; il core dell’enzima è costituito da una proteina chiamata TERT (telomerase reverse trascriptase) e da TERC (telomerase RNA component) un piccolo RNA. In sintesi la telomerasi è una trascrittasi inversa che utilizza il suo RNA come stampo per allungare l’estremo 3’ grazie ad un secondo intervento della DNA polimerasi. Si suppone che l’espressione della telomerasi sia repressa nelle cellule somatiche, ma non nelle cellule germinali. Ciò significa che nelle prime le estremità telomeriche siano soggetto ad una continua erosione e l’accorciamento telomerico risulta essere un evento fondamentale che conduce alla senescenza della stessa cellula. La riattivazione della telomerasi in queste cellule è uno dei passaggi chiave nella trasformazione neoplastica. Recentemente nell’uomo è stata individuata una proteina denominata TRF1 capace di legarsi ai telomeri. La sua assenza provoca l’allungamento dei telomeri, mentre la sua sovraespressione ne provoca l’accorciamento.
Scaricare