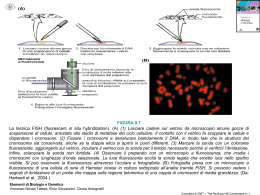
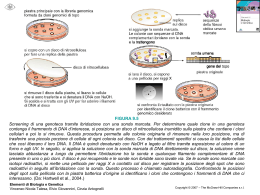
MARCATURA ISOTOPICA Radioisotopi più usati per marcare gli acidi nucleici Isotopo 3H 32P 33P 35S Emivita 12.4 anni 14.3 giorni 25.5 giorni 87.4 giorni Tipo di emissione beta beta beta beta - Energia di emissione 0.019 MeV 1.710 MeV 0.248 MeV 0.167 MeV Marcatura con 3H :nucleotide con isotopo in posizioni diverse Marcatura con 32P, 33P: 1) nucleotidi con l’isotopo nel gruppo fosfato in (marcatura interna); 2) un nucleotide con l’isotopo nel gruppo fosfato in che viene sostituito dal P del nucleotide terminale (marcatura terminale con chinasi) Marcatura con 35S: inserito nucleotide con l’isotopo fosfato in posizione 35S al posto dell’O- del gruppo MARCATURA INTERNA DEL DNA MEDIANTE “RANDOM PRIMING” Klenow: attività endonucleasica 5’ no esonucleasica 5’ 3’ (distruggerebbe primer) si esonucleasica 3’ 5’ 3’ MARCATURA INTERNA DEL DNA MEDIANTE “RANDOM PRIMING” MARCATURA TERMINALE DEL DNA MEDIANTE POLINUCLEOTIDE CHINASI MARCATURA NON ISOTOPICA - Diretta: incorporazione di nucleotidi modificati contenenti un fluoroforo (gruppo chimico che emette fluorescenza se esposto a certe lunghezze d’onda) - Indiretta: incorporazione di un nucleotide al quale è stato legato un gruppo indicatore (R); individuazione dell’ibridazione della sonda per riconoscimento di (R) da parte di un “anticorpo” (A) che viene evidenziato con un marcatore (M) Digossigenina (steroide vegetale) e biotina (vitamina B7) hanno un gruppo indicatore (incorporato nell’acido nucleico) che può essere riconosciuto da ligandi specifici. Esempi: La biotina viene legata dall’avidina (glicoproteina presente nell’albume dell’uovo) legata a marcatore fluorescente saggio fluorimetrico; La digossigenina viene legata da un anticorpo monoclonale coniugato con la fosfatasi alcalina saggio enzimatico con conversione di un substrato incolore Fluorofori per la marcatura non isotopica diretta dUTP Fluorescin-dUTP (verde) Rhodamine-dUTP (rosso) Marcatura non isotopica indiretta Marcatura non isotopica indiretta Deossiuridina trifosfato (dUTP) modificata con Biotina, ibridazione con Avidina accoppiata con marcatore fluorescente Sonda DNA complessata con perossidasi di rafano (Horseradish peroxidase, HP) + luminolo: sviluppo di chemioluminescenza rilevata con autoradiogramma Tipi di sonda utilizzati per l’ibridazione degli acidi nucleici: DNA, RNA, Oligonucleotidi Tipi di ibridazione: Southern blot (in genere: marcatura isotopica) Northern blot (in genere: marcatura isotopica) Ibridazione di filtri con colonie (in genere: marcatura isotopica) Ibridazione in situ di cromosomi per mappatura citogenetica (ora non isotopica) Ibridazione in situ di tessuti (ora non isotopica) Ibridazione di filtri con spot di DNA da PCR (dot blot) IBRIDAZIONE DI UN SOUTHERN BLOT Specificità dell’ibridazione: Temperatura di ibridazione Concentrazione salina Temperatura lavaggi IBRIDAZIONE DI UN NORTHERN BLOT IBRIDAZIONE DI FILTRI CON COLONIE IBRIDAZIONE IN SITU su campione intero Espressione di un gene per la crescita dei fibroblasti nell’embrione di pollo. Trascritti marcati con sonda antisenso marcata con digossigenina e rilevati con anticorpi antidigossigenina accoppiati a fosfatasi alcalina MAPPATURA CITOGENETICA CON IBRIDAZIONE IN SITU DI SONDE MARCATE Procedura sperimentale: - Mitosi fissate su vetrino - Parziale deproteinizzazione con proteasi - Denaturazione ad alte temperature (90-100°C) - Ibridazione con sonda denaturata e marcata con isotopo radioattivo o colorante fluorescente - Lavaggi - Esposizione con lastra autoradiografica e sviluppo Svantaggi dell’uso di tritio: - Utilizzo del radioattivo - Bassa risoluzione (diffusione della radiazione ed alto background) - Bassa sensibilità MAPPATURA CITOGENETICA CON IBRIDAZIONE IN SITU MAPPATURA CITOGENETICA CON IBRIDAZIONE IN SITU - Localizzazione diretta di sonde su cromosomi metafasici - Posizionamento contemporaneo di più sonde lungo i cromosomi - Localizzazione di sonde rispetto a bande e strutture conservate (centromeri e telomeri) Fluorescent In Situ Hybridization: FISH Ibridazione in situ fluorescente - Metodo diretto: Marcatura della sonda con nucleotidi coniugati a fluorocromi - Metodo indiretto: Riconoscimento della sonda con una seconda sonda marcata con fluorocromi Visualizzazione diretta al microscopio a fluorescenza - FISH SU CROMOSOMI METAFASICI (Risoluzione 1 Mb) - FISH SU CROMOSOMI INTERFASICI (Risoluzione tra 500-50 Kb) - FISH SU CROMOSOMI IN INTERFASE ARTIFICIALE (700 - 5 Kb) (Fiber FISH) MICROSCOPIA A FLUORESCENZA dUTP Fluorescin-dUTP (verde) Rhodamine-dUTP (rosso) FISH Ibridazione con sonda che copre il sito di rottura cromosomica FISH multicolore - Utilizzo sostanza fluorescenti con diverso spettro di emissione (FITC, fluorescina isotiocianato, Texas Red, rodamina) - Utilizzo contemporaneo di sonde marcate con diversa localizzazione lungo il cromosoma CHROMOSOME PAINTING - Sonde: collezione di frammenti specifici di ogni cromosoma - Ogni cromosoma risulta fluorescente ed ha un colore diverso - Utilizzo di un microscopio a fluorescenza ed analisi digitale dell’immagine - Cariotipo molecolare (spectral karyotype SKY) CHROMOSOME PAINTING Sonde: collezione di frammenti specifici di un singolo cromosoma Cromosomi fluorescenti con colori diversi Utilizzo di un microscopio a fluorescenza ed analisi digitale dell’immagine Chromosome painting FISH multicolore
Scaricare