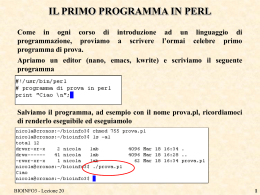
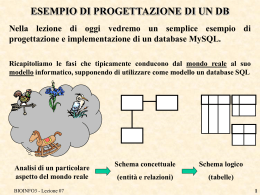
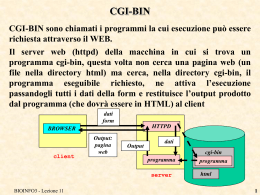
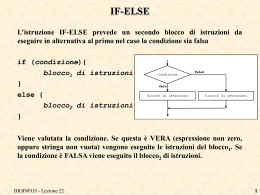
INTERAZIONE CON UN SERVER MYSQL
Quando abbiamo parlato dei database relazionali SQL vi avevo già accennato
che l’interazione (invio di comandi e ricezione di risultati) poteva avvenire in
due modi:
•Direttamente attraverso un client interprete di comandi.
Ed è quanto abbiamo fatto finora a lezione e nelle esercitazioni, richiedendo,
dalla linea di comando UNIX, l’esecuzione del client mysql. I comandi sono
digitati direttamente e il programma client colloquia, attraverso la rete, con il
server, utilizzando il protocollo TCP/IP (il protocollo standard per comunicare
attraverso internet)
•Attraverso il cosiddetto “embedding” in un linguaggio di programmazione.
In questo caso si utilizza l’intermediazione di “librerie” del linguaggio, che
permettono al programma di colloquiare con il server. Il programma chiede
l’esecuzione di alcune funzioni fornendo loro il comando SQL da eseguire
sotto forma di stringa di testo.
BIOINFO3 - Lezione 34
1
MODULI DI PERL (es. BIOPERL)
Per il Perl esiste un gran numero di librerie (dette “moduli”)
anche per molte altre cose, ad esempio anche per la
BIOINFORMATICA (il cosiddetto BIOPERL). Suggerisco a chi
ne fosse interessato e alla fine del corso volesse approfondire
l’argomento di consultare il sito www.bioperl.org.
Questa libreria aggiunge al Perl la
possibilità di svolgere in modo semplice
le più comuni (e ripetitive) operazioni
che normalmente si effettuano in
programmi bioinformatici: esecuzione
di programma (BLAST, PSI-BLAST,
GENSCAN…), interpretazione dei
risultati ed estrazione di informazioni (il
cosiddetto “PARSING” o, italianizzato,
“PARSERIZZAZIONE”), gestione di
database di sequenze, tipicamente a flatfile, conversione tra vari formati dei dati
BIOINFO3 - Lezione 34
2
EMBEDDING DI MySQL in PERL
Ma torniamo al modulo di Perl per MySQL.
Per avvalersi di questa libreria, che aggiunge al linguaggio alcune funzioni per
interagire con server MySQL è necessario utilizzare l’istruzione:
use Mysql;
Molti moduli vengono già distribuiti insieme al Perl, altri si possono installare
successivamente, scaricandoli dal sito di chi li ha realizzati.
Bisogna precisare che i moduli non vanno a modificare il linguaggio
aggiungendo nuove istruzioni, ma utilizzano degli strumenti evoluti forniti dal
linguaggio stesso (abbiamo già accennato a funzioni o ad esempio utilizzando
la programmazione ad oggetti, di cui però non parleremo in questo corso)
BIOINFO3 - Lezione 34
3
CONNESSIONE AL SERVER
Una volta dichiarato che si intende usare la libreria del Perl per MySQL è
necessario collegarsi al server MySQL desiderato ed a un database specifico
gestito dal server. L’istruzione è del tipo
Mysql->connect(server,database,user,password);
Server. L’indirizzo del server. Ad esempio nelle esercitazioni avete usato e
userete sibilla.cribi.unipd.it. Si può scrivere semplicemte “sibilla” se il
programma verrà eseguito in un computer della sottorete in cui il server
sibilla è conosciuto. Se invece il programma gira in un computer fuori dalla
sottorete si deve indicare l’indirizzo completo “sibilla.cribi.unipd.it” o anche
l’indirizzo IP (“147.162.3.226”). Se il programma è sulla stessa macchina che
ospita il server si può usare “localhost”.
Database. Il nome del database (es. “btbm-1”).
User e password. Questi vengono decisi dall’amministratore del database per
consentire l’accesso al database solo agli utenti autorizzati. Nel nostro caso
non sono stati definiti per cui si usa “” in entrambi i casi
BIOINFO3 - Lezione 34
4
CONNESSIONE AL SERVER
L’istruzione di connect restituisce un “puntatore” al database che dovrà essere
assegnato ad una variabile per poter successivamente operare con quel
database.
E’ possibile aprire più connessioni contemporaneamente a database diversi
(anche su server diversi) ed ovviamente in ciascun caso si farà riferimento ad
una variabile diversa.
$db=Mysql->connect(“sibilla”,”test”,””,””);
$db2=Mysql->connect(“sibilla”,”btbm-2”,””,””);
BIOINFO3 - Lezione 34
5
CONNESSIONE AL SERVER
Vediamo un esempio di connessione fallita…
In questo caso non esisteva il database “test20” sul server cronos!
BIOINFO3 - Lezione 34
6
CONNESSIONE AL SERVER
… ed una connessione riuscita.
In questo caso il programma non muore con l’istruzione die, ma stampa il
valore della variabile $db, giusto per farvi vedere come essa viene gestita
internamente dal Perl. Il numero è un indirizzo di memoria ove il programma
organizza in una struttura dati opportuna (oggetto) tutte le informazioni che
gli servono per gestire questa connessione.
BIOINFO3 - Lezione 34
7
OPERAZIONI SUL DATABASE
Avendo a disposizione la variabile che rappresenta la connessione con il DB ora è
possibile inviare al database qualunque comando SQL semplicemente scrivendolo
in una stringa ed invocando la funzione query su quella variabile, usando tale
stringa come parametro
$db->query(“…comando SQL…”);
Ad esempio le istruzioni
$query=“insert into enzimir values(‘EcoRI’,’GAATTC’,1)”;
$db->query($query);
effettuano l’inserimento di un record nella tabella enzimir.
Il procedimento è identico per le istruzioni di update e delete.
BIOINFO3 - Lezione 34
8
OPERAZIONI DI SELECT
Finora abbiamo visto le operazioni di SQL che non restituiscono risultati.
Vediamo cosa succede con l’operazione di select, che invece restituisce una
tabella di risultati, formata in generale da diverse righe.
$q=“select * from enzimir”;
$r=$db->query($q);
Tutte le righe di risultato restituite dal server sono assegnate alla variabile $r,
che sarà a sua volta un puntatore ad una zona di memoria dove Perl gestirà
opportunamente questo oggetto complesso. Non dovrete assolutamente
preoccuparvi del valore di $r, così come di quello di $db, che, ripeto, sono
gestiti dal Perl, ma semplicemente usare questa variabile nel modo che tra
poco vedremo.
$r
BIOINFO3 - Lezione 34
9
RISULTATO DI UNA SELECT
Ancora il valore è un puntatore ad un “oggetto” (struttura dati complessa)
contenuta ad un certo indirizzo di memoria
BIOINFO3 - Lezione 34
10
ESTRAZIONE DEI RISULTATI
I risultati vengono “estratti” dalla variabile $r usando una funzione chiamata
“fetchhash”. Ogni volta che viene invocata questa funzione restituisce la riga
successiva di risultati. Quindi la prima volta che viene chiamata, fetchhash
restituisce la prima riga di risultati, la seconda volta la seconda riga e così via
fino all’ultima riga.
La funzione fetchhash restituisce in un array associativo (ancora loro…) la
riga di risultati. L’array associativo ha come chiavi i nomi delle colonne della
tabella restituita dalla query e come valori i valori corrispondenti in quella
riga.
%f=$r->fetchhash;
%f
nome
sequenza
EcoRI
GAATTC
BIOINFO3 - Lezione 34
sitotaglio
1
11
CICLO DI ESTRAZIONE RISULTATI
Tipicamente si effettua un ciclo per estrarre tutte le righe di
risultati
while (%f=$r->fetchhash;){
#
UTILIZZO DELLA RIGA CORRENTE DI RISULTATI (%f)
}
Il ciclo continua fino a quando la fetchhash riesce ad estrarre
righe di risultati da $r assegnandole a %f. Se la select non
restituisce nessuna riga non si entra nemmeno nel while. Esaurite
le righe di risultati la fetchhash restituisce un array associativo
vuoto a %f e pertanto la condizione %f che controlla il while
diventa falsa, facendolo terminare.
BIOINFO3 - Lezione 34
12
Notare come ad ogni
ciclo venga estratta
una riga diversa dei
risultati. Nell’ultimo
ciclo la fetchhash
resituisce un array
associativo vuoto che
fa terminare il while
BIOINFO3 - Lezione 34
13
Join di tabelle
select orfconsensus.name , orfconsensus.chromStart,
orfconsensus.chromEnd, orfannotation.description
from orfconsensus, orfannotation
where orfconsensus.name='PBPRA0671' and
orfconsensus.name=orfannotation.name
$q=“select orfconsensus.name as name , orfconsensus.chromStart as Start,
orfconsensus.chromEnd as End, orfannotation.description as description
from orfconsensus, orfannotation
where orfconsensus.name='PBPRA0671' and
orfconsensus.name=orfannotation.name”;
$r=$db->query($q);
while(%f= $r->fetchhash){
$nome=$f{‘name’};
$Start=$f{‘Start’};
$End=$f{‘End’};
$Description=$f{‘description’};
}
BIOINFO3 - Lezione 34
14
BIOINFO3 - Lezione 34
15
ESEMPIO
Si vuole scrivere un programma Perl eseguito via WEB (CGI) che stampi in
una tabella HTML tutti i record della tabella degli enzimi di restrizione
BIOINFO3 - Lezione 34
16
ESEMPIO
BIOINFO3 - Lezione 34
17
ESEMPIO
Vediamo cosa succede se il programma non trova server o database
BIOINFO3 - Lezione 34
18
ESEMPIO
Proviamo a scrivere anche una pagina web da cui richiamare, clickando su
un link ipertestuale, l’esecuzione della pagina
BIOINFO3 - Lezione 34
19
RIEPILOGO
•Moduli (librerie) del Perl
•Embedding di MySQL in Perl
BIOINFO3 - Lezione 34
20
Scaricare