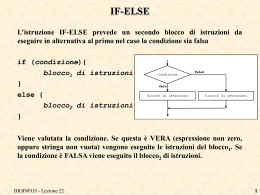
Universita` di Padova Laurea Biologia Molecolare Corso di Bioinformatica III (A.A. 2006-2007) Docente: Dr. Nicola Vitulo Dipartimento di Biologia, CRIBI Tel. 0498276165 Email: [email protected] BIOINFO3 - Lezione 1 1 Calendario Esercitazioni: 1 Marzo 14:00 -18:00 MySql 7 Marzo 14:00 -18:00 HTML 20 Marzo 14:00 -18:00 PERL 23 Marzo 14:00 -18:00 MySql / PERL 30 Marzo 14:00 -18:00 CGI-BIN Competenze informatiche necessarie per il corso: nessuna Tipologia d`esame: scritto (2h) Libri di testo: appunti di lezione (grup.cribi.unipd.it/~nicolav/bioinfoIII_padova/) BIOINFO3 - Lezione 1 2 LA BIOINFORMATICA COS’È LA BIOINFORMATICA? Esistono diverse definizioni..... BIOINFORMATICA= APPLICAZIONE DELL’INFORMATICA ALLA GESTIONE E ALL’ANALISI DEI DATI BIOLOGICI BIOINFO3 - Lezione 1 3 DATI BIOLOGICI Quali sono i dati biologici? Principalmente i dati di sequenza di acidi nucleici e proteine, prodotti in modo sempre più massiccio dai progetti di sequenziamento sistematico (tecnologie sempre piu` sofisticate). Negli ultimi anni vi è stata poi un’invasione di dati relativi ad esperimenti di microarray BIOINFO3 - Lezione 1 4 2007 BIOINFO3 - Lezione 1 5 La bioinformatica e` una branca della biologia in rapida evoluzione, e altamente interdisciplinare in quanto usa tecniche e concetti che derivano dall`informatica, statistica, matematica, chimica , biochimica, fisica. National Center for Biointecnology Information (NCBI) definisce la bioinformatica: la bioinformatica e` la scienza nella quale biologia, informatica e tecnologia dell`informazione si uniscono in un`unica disciplina. Esistono tre importanti sottodiscipline all`interno dell`informatica: BIOINFO3 - Lezione 1 6 1. Sviluppo di nuovi algoritmi e statistiche con i quali valutare le relazioni tra i membri di un ampio data set. 2. Analisi ed interpretazione di vari tipi di dati che includono sequenze aminoacidiche e nucleotidiche, domini proteici, e strutture proteiche. 3. Sviluppo ed implementazione di tool, strumenti, che permettato un efficiente accesso e gestione dei differenti tipi di informazione. BIOINFO3 - Lezione 1 7 National Institute of Health (NIH) Bioinformatica: ricerca, sviluppo o applicazione di strumenti computazionali e di approcci che permettano di espandere e migliorare l`uso di dati biologici inclusi quegli strumenti per l`acquisizione, l`organizzazione, l`archiviazione, l`analisi e la visualizzazione di tali dati. Biologia Computazionale: sviluppo e applicazione di metodi analitici e teoretici, modelli matematici e tecniche di simulazione per lo studio di sistemi biologici. BIOINFO3 - Lezione 1 8 QuickTime™ e un decompressore TIFF (Non compresso) sono necessari per visualizzare quest'immagine. QuickTime™ e un decompressore TIFF (Non compresso) sono necessari per visualizzare quest'immagine. BIOINFO3 - Lezione 1 9 Applicazioni della bioinformatica Computational biology Genomics: la genomica rappresenta l`analisi o la comparazione dell`intero genoma di una o piu` specie. Proteomics: la proteomica consiste nello studio delle proteine - localizzazione, struttura e funzione. Identificazione, caratterizzazione e quantificazione di tutte le proteine coinvolte un un particolare metabolismo, di un organello, cellula, tessuto, organo o organismo. Pharmacogenomics: applicazione degli approcci genomici e tecnologie mirate all`indentificazione dei target delle droghe. Studia in che modo i geni influenzano la risposta ad una droga, sia a livello di popolazione che a livello molecolare BIOINFO3 - Lezione 1 10 Pharmacogenetics: studia in che modo variano le azioni e le reazioni alle droghe. Gli individui rispondono in modo differente al trattamento alle droghe; la maggior parte di questa variabilita` ha basi genetiche. Chemical informatics: memorizzazione, recupero, analisi di informazioni chimiche. Chemometrics: applicazione della statistica all`analisi dei dati chimici. Structural bioinformatics: analisi delle strutture delle macromolecole. Comparative genomics: comparazione del genoma di due o piu`differenti orgnismi. Functional genomics: integrando dati provenienti da sequenziameto di genomi, microarray, proteomica, descrive il funzionamento e l`interazione dei geni. BIOINFO3 - Lezione 1 11 DIMENSIONE “OMICS” I dati biologici hanno guadagnato da tempo il suffisso “-OME” (Genome, Proteome, Trascriptome, Metabolome, Bibliome, Interactome….) e le discipline che li gestiscono e analizzano sono diventate “-OMICS” (Genomics, Proteomics… analisi su larga scala) A chi fosse interessato segnalo il sito http://www.genomicglossaries.com/content/omes.asp che elenca le –ome e gli –omics esistenti BIOINFO3 - Lezione 1 12 Un po` di storia.. Il primo database di dati biologici fu costruito pochi anni dopo che le prime sequenze proteiche cominciarono a diventare disponibili. La prima sequenza proteica ottenuta , di 51 residui, fu l`insulina bovina nel 1956. Circa 10 anni piu` tardi si ottenne la prima sequenza di acidi nucleici, l` alanine rRNA di lievito. Alla fine degli anni `70, Margareth Dayhoff raccolse tutte le sequenze disponibili per creare il primo database biologico (NBRF, National Biomedical Research Foundation). Agli inizi degli anni `80 in Europa l`EMBL promuoveva la creazione dell` EMBL-database, banca dati di sequenze di DNA e RNA. La prima release fu rilasciata nel 1981 e conteneva 519 entries BIOINFO3 - Lezione 1 13 Parallelamente negli Stati Uniti veniva prodotto un archivio simile: banca dati da cui si e` originato GenBank, la cui prima release fu resa pubblica nel 1982. Nel 1986 venne realizzata la banca dati giapponese DDBJ. Accordo tra GeneBank, EMBL e DDBJ per lo scambio giornaliero di dati. Seconda meta` degli anni 80 realizzazione delle prime banche dati specializzate come PROSITE -> innesco per la realizzazione di banche dati sempre piu` specializzate. Sistemi di retrieval: SRS (EBI) e ENTREZ (NCBI). BIOINFO3 - Lezione 1 14 Metodologie bio-computazionali associate alle procedure di confronto di biosequenze per la ricerca di regioni di similarita`. Nel 1970 Needlaman e Wunsch pubblicano l`algoritmo per la ricerca del miglior allineamento globale tra due sequenze. Nel 1971 Gibbs e McIntyre pubblicano un metodo basato sulla matrice basato dot-plot che permetteva la visualizzazione regioni di similarita` piu` o meno stringente , utilizzato poi in numerosi algoritmi di analisi comparative. Nel 1981 Smith e Watermann pubblicano l`algoritmo per il miglior allineameno locale tra due sequenze. Nel 1983 Wilbur e Lipmann pubblicano un algoritmo per la ricerca di similarita` in banca dati e nel 1985 viene pubblicato FASTA, seguito poi nel 1990 da BLAST (Altshul) BIOINFO3 - Lezione 1 15 In parallelo furono sviluppati numerosi metodi per la ricerca di motivi, per la caratterizzazione di sequenze genomiche di regioni codificanti proteine. Per quello che riguarda gli studi di evoluzione molecolare fondamentale e` stata nel 1965 la pubblicazione da parte di Zuckerkandl e Pauling dell`ipotesi dell` “orologio molecolare” (relazione di proporzionalita` diretta tra tempo di divergenza e numero di sostituzioni tra proteine omologhe). 1966 Dayhoff metodo della Massima Parsimonia per l`analisi delle proteine , esteso nel 1977 da Fitch all`analisi delle sequenze nucleotidiche. Metodo di Zucker per la predizione di strutture di RNA e il metodo di Fasman per strutture secondarie proteiche. BIOINFO3 - Lezione 1 16 Esigenza di avere i programmi che implementatano i vari algoritmi in per l`analisi dei dati organizzati in un a logica omogenea e interfacciati con i database di dati biologici: GCG (Genetic Computer Group, Oxford) EMBOSS: prodotto dalla comunita` EMBnet, scaricabile gratuitamente dalla rete (http://www.embnet.org, http://emboss.sourceforge.net/download/) Phylip: pacchetto per analisi di evoluzione molecolare. 1987 : Perl (Practical Extraction Report Language) is released by Larry Wall. 1991: Linus Torvalds announces a Unix-Like operating system which later becomes Linux. 1995: The Haemophilus influenzea genome (1.8 Mb) is sequenced. The Mycoplasma genitalium genome is sequenced. BIOINFO3 - Lezione 1 17 1996: The genome for Saccharomyces cerevisiae (baker's yeast, 12.1 Mb) is sequenced. The Prosite database is reported by Bairoch, et.al. Affymetrix produces the first commercial DNA chips. 1997: The genome for E. coli (4.7 Mbp) is published. 1998: The genomes for Caenorhabditis elegans and baker's yeast are published. The Swiss Institute of Bioinformatics is established as a non-profit foundation. Craig Venter forms Celera in Rockville, Maryland. 2000: The genome for Pseudomonas aeruginosa (6.3 Mbp) is published. The A. thaliana genome (100 Mb) is secquenced. The D. melanogaster genome (180Mb) is secquenced. 2001: The human genome (3,000 Mbp) is published. .... BIOINFO3 - Lezione 1 18 LA BIOINFORMATICA OGGI Si tratta di una disciplina in rapida evoluzione: i libri di testo non sono in grado di tenere il passo con le novità e con i moltissimi database e programmi pubblicati di continuo. Per rimanere aggiornati l’unica possibilità è la rete. Esistono siti specializzati su particolari argomenti (es. Individuazione dei geni, text mining, systems biology..) che cercano (a fatica) di tenere un indice delle pubblicazioni, dei database e dei programmi dedicati a quel particolare ambito ristretto. Fondamentali sono i siti delle riviste scientifiche che accolgono (dopo un lungo e profondo processo di “peer reviewing”) le pubblicazioni. BIOINFO3 - Lezione 1 19 I DUE ASPETTI DELLA BIOINFORMATICA GESTIONE DEI DATI → DATABASE ANALISI DEI DATI → COMPUTATIONAL BIOLOGY BIOINFO3 - Lezione 1 20 DATABASE E COMPUTATIONAL BIOLOGY DATABASE Memorizzazione accurata, organizzazione, indicizzazione e mantenimento di informazioni biologiche COMPUTATIONAL BIOLOGY Qui la lista è lunghissima e sempre in evoluzione. Vi cito solo alcune delle possibili analisi dei dati di cui si occupa la computational biology: •ricerca di similarità tra sequenze (ricerca di omologia funzionale) (dovrebbe essere chiara la differenza tra similarità ed omologia) •ricerca di geni nelle sequenze di DNA •ricerca di motivi funzionali nel DNA (es. siti di binding per fattori di trascrizione) e nelle proteine (domini) •analisi dei genomi •allineamento multiplo di sequenze e analisi filogenetica •analisi di dati strutturali 3D DI PROTEINE •analisi dei risultati di esperimenti con microarray BIOINFO3 - Lezione 1 21 GLI STRUMENTI CHE VEDREMO NEL CORSO GESTIONE DATI MySQL HTML Perl ANALISI DATI MySQL: linguaggio per definizione e gestione database HTML: linguaggio per la definizione di pagine web (accesso ai database e ai programmi attraverso Internet) Perl: linguaggio di programmazione BIOINFO3 - Lezione 1 22 Mysql : http://dev.mysql.com/downloads/mysql/5.0.html Perl: http://www.activestate.com/Products/ActivePerl/ BIOINFO3 - Lezione 1 23
Scaricare