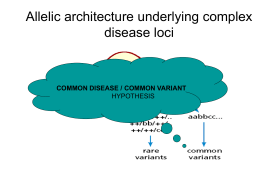
Lezione 11-12 Martedì 23-XI-2010 ore 14:00-15:00 aula 6a Genomica corso di laurea a.a. 2010-2011 biotecnologie industriali i polimorfismi in tutte le specie interesse per la biodiversità coltivazioni ed allevamenti riducono i polimorfismi per effetto della selezione delle specie e varietà più convenienti ed utili per la produzione studio dei polimorfismi come marcatori neutrali per l’analisi di popolazioni ribaltamento fenotipo / genotipo / fenotipo studio dei polimorfismi associati ai fenotipi nella genomica parliamo di wgs nella specie umana WGS wide genome screening perchè ci sono i polimorfismi e perchè si conosce l’intero genoma gli studi sulla specie umana derivano dal fatto che sono utili sia a livello di conoscenze di base di biologia che per l’applicazione alla patologia negli organismi modello di laboratorio si fanno altri tipi di esperimento che sull’umano non si può come nei topi transgenici ecc. analisi del genoma tramite wgs (wide genome screening) cosa significa e come si fa devono essere noti i marcatori SNPs o VNTR con una densità nota, distribuzione omogenea ed alta densità (≠ risoluzione) ultimi metodi a densità media di un marcatore / 500bp più marcatori più risoluzione necessità di nuove tecniche J.N.Hirschhorn & M.J.Daly Nature Reviews Genetics vol.6; Febr.2005 Genome-wide association studies for common diseases and complex traits analisi genomiche approccio globale sull’intero genoma sull’uomo: analisi di associazione con fenotipi vari approcci mappare geni o loci: per malattie genetiche e tratti quantitativi tramite: la strategia di studio dei geni candidati e genome wide studies in entrambe i casi si usa “linkage mapping” o genome wide association studies quando si usa un metodo e quali vantaggi hanno con quali obbiettivi analisi della variabilità genetica di popolazione linkage disequilibrium LD associazione con marcatori e triadi o famiglie (iniziata per malattie Mendeliane) analisi di associazione dei fenotipi e patologie confronto casi controlli brevetti sui genomi? si possono brevettare solo strutture artificiali, caso mai un test, ma non sequenze o organismi si può discutere sul micoplasma sintetico non sulle varianti alleliche naturali si è dovuti arrivare a definire patrimonio dell’umanità, ciò che ovviamente è di tutti beni inalienabili: aria e acqua, chi li inquina dovrebbe ripulirli i limiti degli studi di linkage grande utilità per studi di malattie monogeniche Mendeliane uso di markers che segregano nelle famiglie col gene della malattia per definizione malattie monogeniche hanno alta penetranza i marcatori della malattia cosegregano entro 10-20 Cm (ci sono poche varianti polimorfiche a causa della selezione negativa) è stato applicato anche per malattie non monogeniche con regioni con forte linkage per varianti che aumentano la suscettibilità alla malattia: infiammaz.intest, Diabete 1, schizofr. successi parziali che spiegano solo parte (non arrivano oltre a tre volte il rischio) essendo multifattoriali con 30x di rischio due diverse metodologie wide genome scr. association: analisi frequenze caso controllo analisi di lnkage disequilibrium: famiglie grandi, triadi, popolazioni inbred, popolazioni omogenee per definizione sappiamo che c’è linkage tre marcatori con 6 alleli: A a B b C c aplotipi con freq uguali, se in disequilibrio aumenta la frequenza di uno degli aplotipi rispetto agli altri esiste disequilibrio per effetto della mutazione recente al’interno della stessa coorte motivazione delle carenze - bassa ereditabilità dei tratti genetici più complessi - mancanza di loci (markers) a meno di 10 cM per avere informazione più completa - definizione precisa del fenotipo - inadeguatezza del piano di indagine sperimentale - gli studi devono essere molto estesi (molti campionifamiglie) - complicazione nel verificare molti tratti genetici quantitativi che partecipano solo in una piccola parte al fenotipo patologico, i singoli polimorfismi possono avere solo un peso limitato nel determinare il rischio della malattia - varianti comuni possono aumentare il rischio di due volte o meno e non essere sotto selezione negativa forte basse frequenze non visibili per linkage questi marcatori con un aumento di rischio di sole due volte o meno si possono osservare solo su grandi popolazioni e quindi non su linkage in famiglie per la bassa frequenza che hanno. sarebbero necessari studi su milioni di gemelli o famiglie nessun studio di genome wide linkage ha evidenziato nel diabete type 2 la regione PPARG che è significativa alleli comuni e ache rari partecipano alle malattie comuni ed ai tratti quantitativi, linkage ha bassa efficienza a trovare alleli comuni a bassa penetranza necessità di strategie alternative linkage mapping e gw association “linkage mapping” efficientissimo per malattie monogeniche Mendeliane le malattie monogeniche hanno una selezione negativa forte perciò varianti rare (morbidità e mortalità alte e precoci), alta penetranza per definizione, i markers cosegregano entro 1020 cM successo in alcune malattie multifattoriali: Inflam Bowel dis (IBD); Shizofrenia, Diabete tipo I analisi dei geni candidati Ethnic difference in patients with type 2 diabetes mellitus in inter-East Asian populations: a systematic review and meta-analysis focusing on gene polymorphism. Takeuchi M, Okamoto K, Takagi T, Ishii H. J Diabetes. 2009 Dec;1(4):255-62. BACKGROUND: We previously reported that the fasting serum insulin level was significantly lower in Japanese patients than in Korean and Chinese patients, and showed evidence that a difference in the dietary component would be one of the most influential factors for the ethnic difference. However, it is well known that type 2 diabetes mellitus (T2DM) results from the interaction between genetic predispositions and environmental risk factors. Therefore, we investigated ethnic differences by focusing on gene polymorphism, possibly related to T2DM in Japanese, Korean, and Chinese subjects. METHODS: Data sources included MEDLINE and EMBASE between January 2001 and October 2008. We conducted a search for articles containing minor allele frequency (MAF) in the gene polymorphisms of peroxisome proliferator-activated receptor-γ (PPARG), inward-rectifying potassium channel Kir6.2 (KCNJ11), Calpain 10 (CAPN10), and transcription factor 7-like 2 (TCF7L2). The pooled odds ratio was calculated by using a fixed-effects model with the Mantel-Haenszel method after confirming statistical evidence of homogeneity across the ethnicities using the Breslow-Day test. RESULTS: The Breslow-Day test revealed that there were no statistically significant differences between ethnicities in pooled odds ratios for the gene polymorphisms in PPARG (P = 0.828), KCNJ11 (P = 0.194), CAPN10 (P = 0.090), and TCF7L2 (P = 0.376). Also, pooled odds ratios of each gene polymorphism in East Asians were 0.645 for PPARG (P = 0.000), 1.168 for KCNJ11 (P = 0.000), 0.967 for CAPN10 (P = 0.759), and 1.386 for TCF7L2 (P = 0.000). CONCLUSION: The results of this study and our previous studies suggest that behavioral and environmental risk factors have a more significant impact on ethnic difference in East Asian patients with T2DM compared with genetic predispositions. analisi dei geni candidati selezionati o da regioni di linkage o da evidenze che mostrano il rischio tecnica più comune è il risequenziamento del gene candidato nei pazienti e controlli si cerca la variante o il set di varianti che sono più frequenti o assenti nei soggetti ammalati spesso vengono sequenziate le regioni codificanti ed esoni analisi di geni candidati sistema alternativo al LD per appartenenza a regioni in LD per evidenze che riguardano/influenzano la patologia analisi più comune con resequencing di pazienti e controlli (ricerca delle varianti che sono più presenti o assenti nella patologia) analisi spesso ristretta a regioni codificanti o a singolo esone come il gene recettore per la “melanocortin-4” implicato in una piccola percentuale dell’obesità giovanile alcuni alleli introvabili con LD alleli a bassa suscettibilità non possono essere trovati a meno di screening con 106 individui es. variante Pro 12 Ala del gene proliferativo recettore g attivato del perossisoma PPARG rischio doppio per diabete tipo 2 mai potrebbe essere trovato per LD (link diseq) solo con screening su oltre un milione di soggetti ragionevole credere che le malattie comuni siano determinate da alleli comuni, rari e tratti con effetto quantitativo, necessaria strategia complementare esempio Cohen and colleagues have successfully applied the resequencing approach to high-priority candidate genes in which severe loss-of function variants cause Mendelian disorders of lipid metabolism; they found that these genes also harbour less severe but still relatively rare missense variants that are associated with high, but not extreme, levels of high-density lipoprotein. Rare loss-of-function mutations in ANGPTL family members contribute to plasma triglyceride levels in humans. Romeo S, Yin W, Kozlitina J, Pennacchio LA, Boerwinkle E, Hobbs HH, Cohen JC. J Clin Invest. 2009 Jan;119(1):70-9. Common Single-Nucleotide Polymorphisms Act in Concert to Affect Plasma Levels of High-Density Lipoprotein Cholesterol. Spirin V, Schmidt S, Pertsemlidis A, Cooper RS, Cohen JC, Sunyaev SR. Am J Hum Genet. 2007 Oct 19;81(6). [ Multiple rare variants in NPC1L1 associated with reduced sterol absorption and plasma low-density lipoprotein levels. Cohen JC, Pertsemlidis A, Fahmi S, Esmail S, Vega GL, Grundy SM, Hobbs HH. Proc Natl Acad Sci U S A. 2006 Feb 7;103(6):1810-5. association studies Association studies using common allelic variants are cheaper and simpler than the complete resequencing of candidate genes, and have been proposed as a powerful means of identifying the common variants that underlie complex traits. In their simplest form, association studies compare the frequency of alleles or genotypes of a particular variant between disease cases and controls. Alternative approaches include using family-based controls to avoid the potential problem of population stratification. case control studies confronto di frequenze alleliche tra i soggetti patologici o con 1 fenotipo e le frequenze della popolazione di controllo problemi di stratificazione: come si devono scegliere le 2 popolazioni da controllare? I paesi oltreoceano hanno popolazioni miste (melting pot) e devono essere pesate le componenti della popolazione di controllo di riferimento e non tutti gli africani o gli europei sono uguali, cioè non basta prendere dei neri e dei bianchi come riferimento. Esiste una statistica apposta per questo: tests di caso-controlli una meta analisi fatta su dati di letteratura pubblicati: (andatevi a vedere cosa è una meta-analisi) Ethnic difference in patients with type 2 diabetes mellitus in inter-East Asian populations: a systematic review and meta-analysis focusing on gene polymorphism. Takeuchi M, Okamoto K, Takagi T, Ishii H. J Diabetes. 2009 Dec;1(4):255-62. METHODS: Data sources included MEDLINE and EMBASE between January 2001 and October 2008. We conducted a search for articles containing minor allele frequency (MAF) in the gene polymorphisms of peroxisome proliferator-activated receptor-γ (PPARG), inward-rectifying potassium channel Kir6.2 (KCNJ11), Calpain 10 (CAPN10), and transcription factor 7-like 2 (TCF7L2). The pooled odds ratio was calculated by using a fixed-effects model with the Mantel-Haenszel method after confirming statistical evidence of homogeneity across the ethnicities using the Breslow-Day test. candidate gene association limits Candidate-gene association studies have identified many of the genes that are known to contribute to susceptibility to common disease. Such studies are greatly facilitated by using indirect LINKAGEDISEQUILIBRIUM (LD)-based methods. However,candidate-gene studies rely on having predicted the identity of the correct gene or genes, usually on the basis of biological hypotheses or the location of the candidate within a previously determined region of linkage.Even if these hypotheses are broad (for example, involving the testing of all genes in the insulin-signalling pathway), they will, at best, identify only a fraction of genetic risk factors, even for diseases in which the pathophysiology is relatively well understood.When the fundamental physiological defects of a disease are unknown, the candidate-gene approach will clearly be inadequate to fully explain the genetic basis of the disease. genome wide association approach definizione: studio di associazione causale di varianti genetiche con una rassegna del genoma. Non ci sono preconcetti sulla regione genomica delle varianti. Il metodo sfrutta la forza dell’associazione senza avere una ipotesi sull’identità del gene causale. E’ un metodo non “bias” (sapete cosa vuol dire?) cioè privo di una preferenza di scelta, anche in presenza di evidenze convincenti contrarie sulla funzione e localizzazione dei geni causali. Deve essere un metodo capace di trovare appunto i geni che potrebbero sfuggire ad una indagine del tipo gene-candidato in cui si suppone l’associazione di un metabolismo ai suoi geni correlati come predisponenti. Qui è l’opposto: ricerca dei geni non correlabili sulla base delle evidenze note. base statistica per WGS Estimating haplotype frequencies by combining data from large DNA pools with database information. We assume that allele frequency data have been extracted from several large DNA pools, each containing genetic material of up to hundreds of sampled individuals. Our goal is to estimate the haplotype frequencies among the sampled individuals by combining the pooled allele frequency data with prior knowledge about the set of possible haplotypes. Such prior information can be obtained, for example, from a database such as HapMap. We present a Bayesian haplotyping method for pooled DNA based on a continuous approximation of the multinomial distribution. The proposed method is applicable when the sizes of the DNA pools and/or the number of considered loci exceed the limits of several earlier methods. In the example analyses, the proposed model clearly outperforms a deterministic greedy algorithm on real data from the HapMap database. With a small number of loci, the performance of the proposed method is similar to that of an EMalgorithm, which uses a multinormal approximation for the pooled allele frequencies, but which does not utilize prior information about the haplotypes. The method has been implemented using Matlab and the code is available upon request from the authors. Gasbarra D, Kulathinal S, Pirinen M, Sillanpää MJ. University of Helsinki, Helsinki. IEEE/ACM Trans Comput Biol Bioinform. 2011 Jan-Mar;8(1):36-44. metodi disponibili per analisi throughput perchè Genome Wide ass. studies “approaches to mapping the genes that underlie common disease and quantitative traits fall into two categories: CANDIDATE-GENE studies, which use either association or resequencing approaches, and genome-wide studies, which include both LINKAGE MAPPING and genome-wide association studies. The approaches and their advantages and disadvantages are summarized in TABLE 1. In this review,we discuss these approaches and present arguments as to why genome wide association studies might be advantageous for identifying the genetic variants associated with common disease. One fundamentally different approach, ADMIXTURE MAPPING**, is not discussed here but has been described elsewhere7–10.” ** studi su campioni di popolazioni mescolate es: America (USA, Brasile ecc.) il problema è il controllo di riferimento nelle patologie non Mendeliane o fenotipi multi fattoriali bassa penetranza: aumenta la necessità di ampiezza del campione per la significatività della statistica ricerca in coorti ad origine limitata e popolazioni isolate es. Sardegna, Giappone, Foresta Amazzonica ecc. analisi dei geni identificati per confronto esempio dei gemelli Infl Bowel Disease solo 2x il rischio per fattori noti il calcolo totale del rischio è 30x quindi il n. di fattori ignoti è molto grande quali cause: bassa ereditabilità di tratti genetici complessi copertura parziale dei markers con microsatelliti definizione imprecisa dei fenotipi protocolli di studio non adeguatamente potenti necessità di studi estesi di geni candidati (più difficili?) da regioni di 10 cM (~106 bp) al gene dalla regione di linkage per arrivare al gene analisi dei geni presenti possibili geni candidati per funzione sequenziamento e ricerca degli alleli nel caso di mutazioni non ci potrà essere disequilibrium il sistema è anche poco efficiente per trovare le varianti genetiche che hanno poco effetto sulla malattia effetti quantitativi tratti con effetti quantitativi con architetture complesse influenzano il fenotipo con la somma e le interazioni tra fattori ambientali e genetici le singole varianti alleliche hanno poca influenza le varianti comuni freq >1% possono dare suscettibilità resistono alla selezione negativa per la bassa influenza ne sono stati trovati con un aumento di rischio = 2x limiti degli studi gene candidato l’ipotesi biologica, fisiopatologica, testare tutti i geni del pathway insulin-signalling dipendente identificazione solo di una frazione dei fattori genetici di rischio nonostante le buone conoscenze biologiche in assenza di conoscenze fondamentali fisiopatologiche della patologia in esame il sistema è inadeguato (cercare l’ago nel pagliaio senza la calamita o metal detector) studi di associazione in assenza di certezze fisiopatologiche della malattia l’analisi dei geni candidati non è possibile uso di varianti alleliche comuni meno caro del risequenziamento efficiente per evidenziare regioni complesse analisi per confronto delle varianti caso/controllo uso alternativo del controllo con analisi famiglia/malato wgs per associazione si cercano le regioni associate “in linkage” con gli alleli possibili dei geni implicati (niente a che fare con LD) bassa penetranza per la presenza di molti fattori analisi di campioni sempre maggiori per gli alleli rari assenza di presunte regioni preferenziali, no bias assenza di evidenze della collocazione e funzione dei geni coinvolti miglioramento delle tecniche essenziale per sveltire analisi molto estese database di 96 dbSNPs umani con frequenze > 1% costi attuali ~0.001 $ x genotipo = 500 $ per individuo varie tecniche adottate, la maggior parte con PCR e ibridazione su chip array presenza di aplotipi e LD “ENCODE” encyclopedia of DNA elements esistenza di lunghi tratti di genoma in LD alta predittività degli SNPs vicini (o recenti o senza ricombinazione) conosciute le regioni in LD (aplotipi) si riducono gli SNPs da analizzare si deve analizzare la porzione di genoma a basso LD nascita della Hap Map data poche centinaia di migliaia di SNPs per mappare l’intero genoma mappa degli aplotipi determina le regioni di LD e blocchi di genoma con gli stessi alleli che non ricombinano nelle popolazioni antiche come in Africa ci sono meno LD sono necessari più SNPs (c’è stato più tempo per ricombinazione, i gruppi etnici del resto del mondo partono da alcuni colli di bottiglia con meno alleli e meno aplotipi approccio missenso scelta degli SNPs missenso in regioni codificanti 1-2 SNPs in media per gene riduce a 30-60 x103 il n.tot. di SNPs la ricerca di tutti gli SNPs dei geni prevede invece il resequencing bidirezionale di 3x105 esoni x 48 soggetti ~306 sequenziamenti possibile solo con i nuovi metodi di sequenziamento massivo possibili paradossi: linkage di SNP missenso con variante di regione regolativa (RR) es. CTLA4 cytotoxic T-lymphocyte-associated protein 4 Thr17Ala in LD con RR polimorfica non codificante in associazione + forte con autoimmunità metodi esistenti (alcuni con brevetto) Selected commercially available high-throughput genotyping platforms Company Method of allele discrimination Method of detection Number of assays detected simultaneously Third Wave PCR, cleavase Fluorescence; plate reader Sequenom PCR, primer extension Mass spectrometry 7–12 ABI PCR, primer extension Fluorescence; gel electrophoresis 48 Illumina Oligo ligation, generic PCR Fluorescence; tags on beads 1.536 Parallele Gap closure, generic PCR Fluorescence; tags on array 10.000 Affymetrix Generic PCR, hybridization Fluorescence; hybridization to array 10,000–100,000 Perlegen PCR, hybridization Fluorescence; hybridization to array 100,000+ 1 (multiplexed 100-fold at PCR stage only) approcci diversi su topo esperimento per fare topi transgenici in ogni parte del genoma trascritta e tradotta (esoni) creare una collezione (library) di cellule embrionali ricombinanti in ogni gene la prospettiva nel restante 95% del genoma genoma si ricomincia da capo nell’interattoma va inserito il genoma e si allargano le prospettive nuove tecniche, metodi, strumenti da chi è diretto il movimento della cromatina cercare di capire come si muove (attiva o passiva) analisi in vivo con microscopia i movimenti fuori dal territorio cromosomico controllati da actina-miosina in topi transgenici [Curr Biol. 2006 Apr 18;16(8):825-31.] effetto “looping out” dal territorio cromosomico dipendente dal tipo cellulare: Hoxd di topo ha il “looping” sull’asse antero-posteriore ma non negli abbozzi degli arti, effetto di ricollocamento nel territorio nucleare del crms X dopo inattivazione di Xist, ma i territori crms non sono barriere per la trascrizione da parte della pol. II tabella 1 GWS *candidate-gene studies; ‡ genome wide studies
Scaricare