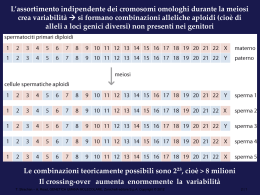
Nella Genetica Umana l’oggetto di studio è sia il fine che il mezzo Difficoltà nello studio della GU: non è possibile programmare gli incroci le fratrie sono di piccole dimensioni il tempo di generazione dello sperimentatore è uguale a quello dell’organismo oggetto di studio Aspetti positivi: le conoscenze riguardanti l’uomo sono molto vaste un fenotipo, anche se raro, è difficile che non capiti all’osservazione esiste una documentazione storica sia per gli individui che per le popolazioni MAPPATURA GENETICA Costruzione di mappe genetiche, cioè di mappe in cui la posizione relativa dei geni (e la distanza tra di essi) viene stabilita attraverso la stima delle frequenze di ricombinazione. Si basano sul fatto che : la frequenza di ricombinazione tra due geni è funzione della distanza fisica che li separa. Il requisito minimo per mappare due geni l’uno rispetto all’altro è che di ENTRAMBI si conoscano almeno due alleli locus A alleli A1 e A2 locus B alleli B1 e B2 Perché ci interessa mappare il genoma? interesse di tipo evolutivo applicazioni pratiche il restringimento della regione cromosomica in cui mappa un gene-malattia costituisce il primo passo nella sua identificazione l’individuazione della regione cromosomica in cui mappa un gene-malattia ed il linkage con altri marcatori trova un’immediata applicazione nella consulenza genetica (diagnosi prenatale, diagnosi presintomatica, diagnosi dello stato di portatore sano) MAPPATURA GENETICA NELL’UOMO METODO DEI LOD SCORE (Morton 1955) ► E’ in grado di distinguere tra associazione e indipendenza ►Non dipende necessariamente dalla fase degli alleli del doppio eterozigote (cis o trans) ► Permette di combinare dati provenienti da famiglie diverse ►In caso di associazione non assoluta è in grado di stimare la frazione di ricombinazione METODO DELLA MASSIMA VEROSIMIGLIANZA (ML = Maximum Likelihood) Metodo che viene applicato quando non è possibile verificare direttamente la veridicità di un’ipotesi Data un’ipotesi A e un certo risultato R la verosimiglianza di A viene calcolata come probabilità che si verifichi R se A fosse vera Esempio: abbiamo un sacchetto contenente 3 tipi di monete in ugual numero: • monete con testa su entrambe le facce (TT) • monete con croce su entrambe le facce (CC) • monete con croce su una faccia e testa sull’altra (CT) Dobbiamo stabilire che tipo di moneta peschiamo senza poterla guardare ma effettuando 4 lanci possibili risultati della serie di 4 lanci e loro probabilità nel caso in cui la moneta sia: 4T 3 T, 1 C 2T, 2C 1T, 3C 4C TT 1 0 0 0 0 TC 1/16 4/16 6/16 4/16 1/16 CC 0 0 0 0 1 Risultato ottenuto (R) = 4T R ci fa scartare l’ipotesi CC e ci fa ritenere più verosimile l’ipotesi TT rispetto alla TC. Ma quanto più verosimile? Dato il risultato R: •Verosimiglianza di TT •Verosimiglianza di TC •Verosimiglianza di CC 1 1/16 0 L’ipotesi TT è 16 volte più verosimile dell’ipotesi TC Rapporto di verosimiglianze TT:TC ‘a priori’ 1:1 ‘a posteriori’ 16:1 Questo metodo si può applicare anche quando il rapporto tra le verosimiglianze ‘a priori’ è diverso da 1:1 Esempio sappiamo che le monete TT sono 20 volte più numerose delle TC A. VEROSIMIGLIANZA ‘A PRIORI’ A FAVORE DI TT = 20:1 B. R = 4 T, cioè VEROSIMIGLIANZA ‘A POSTERIORI’ A FAVORE DI TT = 16:1 C. VEROSIMIGLIANZA GLOBALE A FAVORE DI TT = = (20 x 16) : 1, cioè 320 : 1 Quanto deve essere la verosimiglianza a favore di TT per accettare questa ipotesi come vera? Prima di iniziare l’esperimento si decide una soglia allo scopo di: 1) ridurre al minimo i casi in cui si accetta per buona un’ipotesi che invece è sbagliata; 2) ridurre al minimo i casi in cui si lascia senza risposta il problema in esame Il valore della soglia dipende soprattutto da quanto gravi sarebbero le conseguenze di una decisione errata MAPPATURA GENETICA NELL’UOMO METODO DEI LOD SCORE (Morton 1955) ► E’ in grado di distinguere tra associazione e indipendenza ► Non dipende necessariamente dalla fase degli alleli del doppio eterozigote (cis o trans) ► Permette di combinare dati provenienti da famiglie diverse ► In caso di associazione non assoluta è in grado di stimare la frazione di ricombinazione METODO DEI LOD SCORE, come si procede: 1. reperimento delle famiglie informative (requisito minimo almeno un genitore deve essere doppio eterozigote) e costruzione dei pedigree; 2. determinazione del genotipo di tutti gli individui per uno o più marcatori polimorfici; 3. individuazione e conta dei gameti parentali e dei ricombinanti; 4. valutazione della verosimiglianza ‘a posteriori’ sulla base del risultato osservato, e questo per una serie di ipotesi di linkage, cioè di valori di ricombinazione (); 5. calcolo, per ciascuna frazione di ricombinazione, dell’ODD (= verosimiglianza dell’ipotesi / verosimiglianza dell’ipotesi di indipendenza); 6. calcolo del logaritmo degli ODD (LOD) calcolati al punto 5; 7. costruzione del grafico dei LOD METODO DEI LOD SCORE: 1) Reperimento di famiglie informative UN GENITORE DEVE ESSERE DOPPIO ETEROZIGOTE PER I DUE LOCI MEIOSI INFORMATIVE E NON INFORMATIVE Locus marcatore A, alleli A1, A2 ; Locus malattia (ignoto); malattia Autosomica Dominante individuo sano individuo malato A) NON INFORMATIVA A1 A1 A2 A2 A1 A2 Gli alleli del marcatore in omozigosi nel padre non possono essere distinti Queste famiglie NON sono MAI informative MEIOSI INFORMATIVE E NON INFORMATIVE B) NON INFORMATIVA A1 A2 A1 A2 La figlia ha ereditato dal padre l’allele A1, ma può averlo ereditato insieme ad A1 OPPURE insieme ad A2 A1 A2 C) INFORMATIVA A1 A2 A1 A2 La figlia ha ereditato dal padre A1 INSIEME all’allele malattia A1 A1 I genitori della famiglia B) sono uguali a quelli della C), ma nel primo caso la famiglia è informativa, nel secondo no MEIOSI INFORMATIVE E NON INFORMATIVE D) INFORMATIVA A1 A2 A3 A4 A1 A4 La figlia ha ereditato A1 dal padre insieme all’allele malattia Queste famiglie sono SEMPRE informative Per poter identificare senza ambiguità i gameti parentali e i ricombinanti è necessario avere informazioni su almeno 3 generazioni II-1 ha ricevuto dalla madre l’allele malattia del locus malattia E l’allele A1 del locus marcatore NR NR NR NR NR R II-1 ha ricevuto dalla madre l’allele malattia ma non sappiamo se abbia ricevuto dalla madre l’allele A1 o l’allele A2 del marcatore I loci A e B 1) sono associati? cioè θ < 0.5 questa ipotesi è costituita da n ipotesi, una per ciascuno degli n valori compresi tra 0 e 0.5 oppure 2) sono indipendenti? cioè θ = 1 – θ = 0.5 ? Per cercare di rispondere a questa domanda applichiamo il metodo dei LOD SCORE Il risultato R è rappresentato dal numero dei gameti P e NP probabilità di un gamete P = 1 – θ probabilità di un gamete NP = θ tutte le ipotesi 1 (associazione) prevedono che P > NP l’ipotesi 2 (indipendenza) prevede che P = NP Famiglia 1 5 P e 2 R a) Verosimiglianza di una dell’ipotesi 1 (associazione con θ = 0.01) (1 – θ)5 θ2 = 0.57 = 7.8125 x 10-3 b) Verosimiglianza dell’ipotesi 2 (indipendenza, quindi θ = 0.5) (1 – θ)5 θ2 = 0.992 x 0.015 = 9.5099 x 10-5 ODD = Rapporto di questi 2 valori a/b = 0.0122 LOD = log ODD = - 1.9146 LOD SCORE Z (0) = LOD SCORE Z (0.01) = - 1.9146 LOD SCORE Z (0.05) = - 0.6062 LOD SCORE Z (0.10) = - 0.1216 LOD SCORE Z (0.15) = 0.1065 ECC. IN QUESTO ESEMPIO IL LOD SCORE MASSIMO SI RAGGIUNGE IN CORRISPONDENZA DEL VALORE DI RICOMBINAZIONE = 0.286 (0.2884) Con questi dati si costruisce un grafico dei LOD VALORI DI LOD CRITICI (valori soglia) LOD + 3 Ipotesi di linkage accettata verosimiglianza ‘a posteriori’ a favore del linkage 1000:1 verosimiglianza ‘a priori’ che due loci siano linked 1:50 (a sfavore dell’associazione) verosimiglianza globale 1000:50, cioè 20:1 LOD - 2 Ipotesi di linkage scartata verosimiglianza ‘a posteriori’ a favore dell’indipendenza 100:1 verosimiglianza ‘a priori’ a favore dell’indipendenza 49:1 verosimiglianza globale a favore dell’indipendenza 5000:1 Esempi di curve di lod score 1 Evidenza di linkage assoluto 2 Evidenza di linkage per θ = 0.23 3 Linkage escluso per valori di θ < 0.12 4 Risultato non conclusivo per tutti i valori di θ Il marcatore ideale per studi di mappatura genetica deve essere: altamente polimorfico; analizzabile con una tecnica semplice e a basso costo; analizzabile su un materiale biologico facilmente reperibile; Marcatori ideali sono STR (Simple Tandem Repeats) e SNP (Single Nucleotide Polymorphism) Metodo dei LOD SCORE: range di efficienza: Massima per linkage assoluti. Teoricamente è in grado di scoprire qualsiasi grado di linkage, ma in pratica non è così per scoprire gradi di associazione modesti (= elevata frequenza di ricombinazione) è necessaria una quantità di dati non realisticamente ottenibile. Esempio con 25 gameti informativi si può arrivare a dimostrare che due loci sono linked solo se la frequenza di ricombinazione tra di essi non supera il 10% Se non ci sono ricombinanti 10 meiosi informative sono sufficienti a fornire prova di linkage. Se = 0.3 sono necessarie 85 meiosi informative per dare una prova convincente dell’esistenza di linkage Con la mappatura genetica si può restringere la regione in cui si trova un gene malattia a qualcosa dell’ordine di 1-2 cM (equivalenti a 1-2 Mb) Con il metodo dei LOD SCORE si producono gruppi di associazione (o di sintenia). L’assegnazione ad un particolare cromosoma è possibile solo se un (o più) marcatore facente parte del gruppo di associazione è stato assegnato ad uno specifico cromosoma
Scaricare