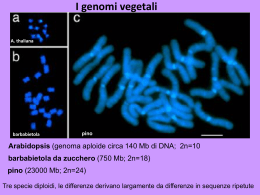
Insegnamento di Genomica Corsi di Laurea Specialistica in: Biotecnologie Agro-industriali Biologia Molecolare e cellulare docenti: Silvia Fuselli ([email protected]) Vincenza Colonna Inga Prokopenko Morena de Bastiani Definizioni • Genomica: Scienza che si interessa della determinazione e dello studio, a livello molecolare, dell’intera sequenza di DNA di un organismo. • Genoma: L’intero contenuto genico di un organismo vivente Genetica Molecolare Informatica 1940-1950 Collegamento tra DNA ed ereditarietà Primi computer 1950-1960 Struttura del DNA Primo computer commerciale 1960-1970 Codice genetico Chip 1970-1980 Sequenziamento DNA Internet collega 4 università americane 1980-1990 PCR PC 1990-oggi Progetto genoma umano Internet collega tutto il mondo Bioinformatica • Informatica applicata alla biologia (molecolare) con diversi scopi: – Generazione di dati: • Interpretazione di dati di sequenziamento • “Assemblaggio” di sequenze e genomi – Organizzazione di dati: • I dati sono memorizzati in banche dati di pubblico dominio • Devono essere accessibili per parole chiave o comunque in modo “semplice” • Devono essere “collegati” tra loro in modo logico: da un cromosoma devo potere risalire ai geni mappati sul cromosoma, dai geni alle proteine che codificano, e così via Bioinformatica • Ma anche analisi (alcuni esempi): – Analisi delle sequenze nucleotidiche per la localizzazione di geni ed elementi di regolazione. – Analisi delle sequenze proteiche allo scopo di riconoscerne la funzione biologica. – Studio delle interazioni intermolecolari tra proteine e acidi nucleici allo scopo di riconoscerne la funzione e sviluppare nuovi farmaci. – Studio dell’evoluzione di geni e genomi per la loro classificazione funzionale e filogenetica – Studio dell’espressione dei geni in diverse condizioni – Sviluppo di tecniche diagnostiche innovative – Progettazione e applicazione di modelli che simulino i processi biologici cellulari Genomica Determinazione e studio dell’intera sequenza del DNA di un organismo. Identificazione e studio dell’espressione dei geni (associati a malattie e non) in essa contenuti. Trascrittomica Proteomica Identificazione, studio e analisi funzionale dell’intero contenuto di mRNA di una cellula. Studio sistematico della struttura (primaria, secondaria, terziaria), della funzione e delle interazioni tra proteine. Studio di serie di processi biochimici legati tra loro da relazioni funzionali. Reti metaboliche Es:. Processi chimici e biochimici che portano alla morte cellulare nelle patologie tumorali 5 agosto 2005 – da ensembl 5 maggio 2006 – da ensembl Ensembl release 53 - Mar 2009 Obiettivi fondamentali della Genomica Tutti i “progetti genoma” condividono un gruppo comune di scopi: • Stabilire un database ed un’interfaccia di ricerca, reciprocamente integrati e disponibili su Internet • Ottenere e combinare mappe fisiche e genetiche del genoma • Generare ed ordinare sequenze genomiche e sequenze di geni espressi (allineamento contig, segnali di sequenze espresse: EST) • Identificare ed annotare tutti i geni codificati da un determinato genoma • Costruire atlanti di espressione genica (analisi dei microarray) • Accumulare dati funzionali, identificando le caratteristiche biochimiche, cellulare e fisiologiche dei geni (Genomica funzionale, Farmacogenomica). • Caratterizzare la diversità di sequenza del DNA attraverso lo studio della distribuzione dei polimorfismi, analisi del LD (Genetica forense, Genetica delle popolazioni) • Fornire le risorse per eseguire comparazioni tra i genomi (Genomica comparativa) Programma • Anatomia dei genomi – I genomi dei procarioti – I genomi degli eucarioti (I genomi nucleari, I genomi degli organelli) • Studio dei genomi – Enzimi (nucleasi,ligasi,ecc.), Clonazione, Vettori (BAC, YAC, ecc.), PCR • La mappatura dei genomi – Mappatura genetica (RFLP, SSLP, SNPs ecc.) – Mappatura fisica (restrizione, FISH, STS, ecc.) • Metodi per il sequenziamento del DNA – Metodi di sequenziamento – Assemblaggio dei contigua • Funzionamento dei genomi – I domini della cromatina – Modificazioni della cromatina e l’espressione del genoma • Progetto Genoma umano • Evoluzione dei Genomi Diversi tessuti umani sono formati da cellule che si comportano in modo diverso (nel corpo umano ci sono circa 6x1013 cellule) Ogni cellula contiene una esatta copia del genoma (che non è altro che l’intera sequenza del DNA dell’organismo) All’interno del nucleo della cellula ci sono i cromosomi: 22 paia di autosomi e un paio di cromosomi sessuali XY Nei cromosomi si possono individuare porzioni di DNA codificante chiamati geni Dogma Centrale L’ espressione dell’informazione genetica raccolta nelle molecole di DNA, avviene in due stadi: –(i) trascrizione, durante la quale il DNA è trascritto in mRNA –(ii) traduzione, durante la quale l’ mRNA è tradotto per produrre la proteina associata DNA mRNA proteine Espressione Differenziale Se ogni cellula contiene una copia dell’intero genoma, e le cellule sono di diversi tipi (cellule muscolari, cellule cardiache, cellule della pelle, cellule del sangue …), Che cosa le rende differenti ? Espressione genica differenziale, cioè: quando, dove, e in che quantità ogni gene è espresso. Il controllo dell’espressione genica può essere esercitato ad ogni tappa del processo che termina con la sintesi proteica ma avviene prevalentemente a livello della prima tappa, ovvero a livello della trascrizione del DNA in RNA messaggero. Anatomia dei genomi EUCARIOTI: nucleo, mitocondri (cloroplasti) • • • • piante animali funghi protozoi PROCARIOTI • eubatteri (gram +, gram -, cianobatteri) • archeobatteri Anatomia dei genomi eucariotici Componenti nucleari e mitocondrali del genoma umano Genoma nucleare: 3,2 Gb composto da 46 molecole di DNA lineare: La molecola più corta è di ~ 47Mb e la più lunga è di ~ 245 Mb Genoma mitocondriale: molecola di DNA circolare di 16.569 bp; Ci sono ~ 800 mt per cellula e ~10 copie di mtDNA per mitocondrio = 8000 molecole di mtDNA Il genoma mitocondriale umano Sequenziato nel 1981 (Anderson et al.) Control region 16.569 bp Il Genoma umano in numeri • 23 paia di cromosomi; • 2 metri di DNA; • 3,200,000,000 bp (3,2 Gb; 3,200 Mb; 3,200,000 kb); • 20,000-25,000 geni. ??????????? Dicembre, 2003 Maggio, 2005 Maggio, 2004 10% di sequenze ripetute non assegnabili perché appartengono a telomeri, centromeri ecc. Science. 1996 Jun 21;272(5269):1755-62. The complete 685-kilobase DNA sequence of the human beta T cell receptor locus. Rowen L, Koop BF, Hood L. Department of Molecular Biotechnology, University of Washington, Seattle 981957730, USA. The human beta T cell receptor (TCR) locus, comprising a complex family of genes, has been sequenced. The locus contains two types of coding elements-TCR elements (65 variable gene segments and two clusters of diversity, joining, and constant segments) and eight trypsinogen genes --that constitute 4.6 percent of the DNA. Genome-wide interspersed repeats and locusspecific repeats span 30 and 47 percent, respectively, of the 685-kilobase sequence. A comparison of the germline variable elements with their approximately 300 complementary DNA counterparts reveals marked differential patterns of variable gene expression, the importance of exonuclease activity in generating TCR diversity, and the predominant tendency for only functional variable elements to be present in complementary DNA libraries. 65 Vß; 2 cluster DJC con 1 Dß, 6-7 Jß, 1 Cß Un segmento del genoma umano 50 Kb Locus: Recettore ß delle cellule T Cromosoma 7 685 Kb Cosa contiene questa regione di 50 kb, parte del locus che codifica il β T-cell receptor? • un gene: TRY4 (5 esoni, 4 introni) • due segmenti genici: V28 e V29-1, discontinui • uno pseudogene: TRY5 • 52 “genome-wide repeat sequences”: 4 tipi tutti presenti (LINEs, SINEs, elementi LTR, trasposoni) • due microsatelliti • il rimanente 50% della sequenza è composto di DNA a singola copia, non genico e non ripetitivo, a funzione ignota GENOMI EUCARIOTI • tutti sono suddivisi in 2 o + molecole lineari • tutti gli eucarioti hanno anche genomi mitocondriali, alcuni anche cloroplastici • le caratteristiche generali sono comuni, l’unico aspetto molto variabile è la grandezza • il n° di cromosomi non è legato alla complessità della specie, né alla grandezza del genoma (es. salamandra ha un genoma 30 volte più grande dell’uomo ed è distribuito su un n° di cromosomi che è la metà di quello umano) Contenuto in paia di basi del genoma aploide Le dimensioni dei genomi dei vari organismi sono molto variabili. L’ordine di grandezza però coincide in un certo modo con la complessità dell’organismo: •eucarioti semplici (funghi) hanno genomi piccoli, •eucarioti superiori (vertebrati o piante a fiore) hanno genomi grandi AMEBA?!?!? paradosso del valore C Paradosso del valore C manca correlazione tra la complessità di un organismo e la grandezza del suo genoma La grandezza del genoma è correlata al numero di geni in esso contenuti??? Paradosso del numero dei geni es: genoma di lievito = 12 Mb genoma umano = 3200 Mb 0.004 x genoma umano uomo ha ~ 25.000 geni lievito dovrebbe avere 25.000 x 0.004= 100 geni in realtà ne ha 5.800 la correlazione tra le dimensioni del genoma di un organismo ed il numero di geni in esso contenuto non è affatto esatta! esatta Specie Dimensioni del genoma (Mb) N° geni approssimativo Eucarioti S. Cerevisiae C. elegans Arabitopsis thaliana (erba galletta) D. Melanogaster Homo Sapiens 12,1 97 125 180 3200 5800 19000 25500* 13600 27000 Batteri Mycoplasma genitalium Streptococcus pneumoniae Vibrio cholerae EI Tor N16961 Mycobacterium tubercolosis H37Rv Escherichia coli K12 Yersinia pestis CO92 Pseudomonas aeruginosa PA01 0,58 2,16 4,03 4,41 4,64 4,65 6,26 500 2300 4000 4000 4400 4100 5700 Archea Methanococcus jannaschii Archaeoglobulus fulgidusla) 1,16 2,18 1750 2500 (*) circa 8000 geni sono specifici per la fotosintesi Ipotesi: i genomi più grossi presentano un numero di geni maggiore Cr. 7 Cr. 3 rispetto a quello umano il segmento del genoma di lievito • contiene più geni (26 per proteine e 2 per tRNA) • i geni discontinui sono molto pochi: solo 239 introni in tot (nell’uomo > di 300.000) • ci sono poche genome wide repeats: una LTR (Ty2) e 4 LTR troncate (sequenze delta). In questo caso LTR sono 16% del tot, ma in realtà, su tutto il genoma, sono solo il 3.4% (nell’uomo il 44%, in drosofila il 12%) soluzione: i genomi degli organismi meno complessi hanno i geni strettamente impacchettati, e per questo risultano più piccoli. Sembra che le genome-wide repeats abbiano ruolo importante nel determinare il grado di compattezza di un genoma in mais sembrano costituire ~ 50% del genoma sta emergendo che nei genomi di alcune specie queste repeats hanno proliferato massicciamente questo spiegherebbe perché genomi di specie molto simili possono essere anche molto diversi per grandezza Riflette l’eterogeneità degli eventi evolutivi che hanno portato al modellamento dei diversi genomi
Scaricare