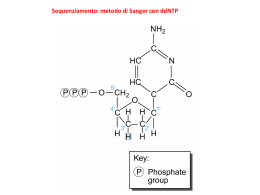
Sequenziamento: metodo di Sanger con ddNTP Sequenziamento con dideossonucleotidi Sequenziamento automatizzato del DNA Clonaggio: Inserto Vettore + Ligazione Trattamento del vettore con fosfatasi AATTC G G CTTAA pAATTC G G CTTAAp fosfatasi DNA ligasi pAATTC G G CTTAAp EcoRI GAATTC CTTAAG GAATTC CTTAAG Vettori plasmidici (pBR322) Vettori plasmidici (pUC18) •Ceppo di E.coli Lac Z mutante •Vettore pUC LacZ parziale •Ceppo di E.coli Lac Z mutante •Vettore LacZ interrotto dal clonaggio Complementano Non c’è complementazione β-galattosidasi ATTIVA Metabolizzato Terreno + X-Gal β-galattosidasi Non metabolizzato INATTIVA X-Gal: 5-Bromo-4-Cloroindolil-β-galattoside (incolore) β-galattosidasi ATTIVA β-galattosidasi INATTIVA Colonie Blu Colonie bianche 1° ciclo di PCR DNA genomico 5’ 3’ 3’ Primer reverse 5’ 3’ Primer forward DNA genomico 5’ 3’ 5’ ETEROGENEITA’ DEL TEMPLATO : ETEROZIGOSI PER UNA DELEZIONE C A G C A G C A G C A G C A C C T C A G C A G C A G C A G C A C C T C A G G G G F P M C/C G/- C/- LOSS OF HETEROZYGOSITY RNA sequencing Dopo trascrizione inversa 6 8 6 7 7/- 8 CTT 7/- -/- Figure 1: Illumina Flow Cell Figure 2: Prepare Genomic DNA Sample DNA Figure 3: Attach DNA to Adapters Surface DNA Dense lawn of primers Figure 4: Bridge Amplification Adapters Figure 5: Fragments Become Double Stranded Attached terminus Figure 6: Denature the Double-Standed Molecules Attached terminus Free terminus Attached Attached Figure 7: Complete Amplification Figure 8: Determine First Base Clusters Laser Figure 9: Image First Base Figure 10: Determine Second Base Figure 11: Image Second Chemistry Cycle Figure 12: Sequencing Over Multiple Chemistry Cycles Figure 13: Align Data Laser GCTGA...
Scaricare