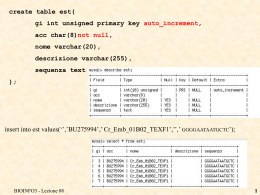
DATABASE PRIMARI Cosa sono i database primari di acidi nucleici? Sono i contenitori di tutte le sequenze prodotte nel mondo e rese disponibili alla comunità scientifica. Memorizzano essenzialmente la sequenza e poche altre informazioni generiche associate (laboratorio di sequenziamento, data, specie, descrizione…) EMBL → Europa GENBANK → USA DDBJ → Giappone I tre database si aggiornano quotidianamente scambiandosi i dati ricevuti durante la giornata. BIOINFO3 - Lezione 2 1 DATABASE COMPOSTI Problemi dei database primari •formato dei dati •accuratezza dei dati •ridondanza I database composti prendono i dati da più sorgenti, generalmente con formati diversi e costruiscono un nuovo database specializzato, in genere pulito e non ridondante Esempio: NRDB (database non ridondante di sequenze di acidi nucleici) BIOINFO3 - Lezione 2 2 Problema della ridondanza ✔ ✔ ✔ ✔ Molti database proteici e nucleotidici contengone sequenze appartenenti a famiglie geniche o versioni di geni omologhi di organismi differenti. Molti gruppi possono sottomettere la stessa sequenza. Differenti approcci sperimentali possono produrre sequenze simili (ad esempio da genomico e da cDNA). Splicing alternativi. BIOINFO3 - Lezione 2 3 L`utilizzo di database ridondanti comporta almeno tre potenziali fonti di errore: ✔Se un dataset contiene larghe famiglie di sequenze strettamente correlate, le analisi statistiche soffriranno di un bias dovuto a sovrastime di caratteristiche peculiari di quella famiglia ✔Apparenti correlazioni in differenti posizioni specifiche delle sequenze possono essere un artefatto dovuto ad un bias nella composizione del campione. ✔Se un dataset e` usato per predizioni di certe caratteristiche, e le sequenze per calibrare la predizioni sono troppo correlate tra loro, l`apparente capacita` predittiva puo` essere sovrastimata. (riconosce il particolare, non il generale). BIOINFO3 - Lezione 2 4 DATABASE SECONDARI Contengono il risultato di analisi eseguite sulle sequenze contenute nei database primari. Esempio. Da SWISSPROT, database primario di sequenze di amminoacidi, sono costruiti i database secondari PROSITE (pattern e profili) e PFAM (domini) BIOINFO3 - Lezione 2 5 INTERROGAZIONI DELLE BANCHE DATI RICERCHE TESTUALI Restituiscono i record di un database che soddisfano i criteri richiesti (mediante utilizzo di parole chiave) Es. “horse liver alcohol dehydrogenase” restituisce risultati specifici per questo enzima. Verranno cercate tutte entry contenenti le 4 parole chiave (“horse AND liver AND alcohol AND dehydrogenase”). operatori booleani-> AND , OR, NOT, &, | , ! RICERCHE PER SIMILARITÀ Restituiscono le sequenze di un database più simili ad una sequenza fornita come query (BLAST, FASTA) BIOINFO3 - Lezione 2 6 SISTEMI INTEGRATI (RICERCA TESTUALE) Esistono dei sistemi integrati che permettono di interrogare, attraverso il web, in modo semplice ed intuitivo le banche dati biologiche. I tre sistemi principali sono: ENTREZ → associato a GENBANK SRS → associato a EMBL DBGET → associato a DDBJ 2 FORM DI QUERY 1 SISTEMA INTEGRATO PAGINA DI RISPOSTA 5 PC UTENTE RETE BIOINFO3 - Lezione 2 4 DB1 3 … DBn COMPUTER „SERVER“ REMOTO 7 ANCORA DATABASE! CLASSIFICAZIONE In generale, considerando la loro natura, possiamo classificare i database in almeno 2 differenti classi principali FLAT-FILE RELAZIONALI Vedremo in dettaglio entrambi i tipi BIOINFO3 - Lezione 2 8 FLAT-FILE Come dice il nome, anche un semplice file di testo può costituire un DATABASE. All’interno del file esistono delle sequenze di caratteri che permettono di individuare i singoli record I record rappresentano l’unità di memorizzazione del database All’interno di un record esistono delle parole chiave che permettono di individuare i campi di quel record In genere esiste un campo (ID, identificatore, chiave…) che identifica univocamente i record del DB (non possono esistere 2 record con lo stesso valore nel campo chiave!) BIOINFO3 - Lezione 2 FLAT-FILE RECORD separatore RECORD separatore RECORD campo1 campo2 campo3 valore1 valore2 valore3 RECORD 9 UN ESEMPIO Quanti record sono? (quale è il separatore di record?) Quali campi ha ciascun record? (quali sono i separatori e gli identificatori di campo?) Quale è il campo chiave? Che valori assume? ID PARENT ID RANK GC ID SCIENTIFIC NAME SYNONYM SYNONYM SYNONYM // ID PARENT ID RANK GC ID MGC ID SCIENTIFIC NAME // ID PARENT ID RANK GC ID MGC ID SCIENTIFIC NAME // : : : : : : : : 28877 28876 no rank 1 IDIR agent Infectious Disease of Infant Rats Rotavirus (GROUP B / STRAIN IDIR) infectious diarrhea of infant rats agent IDIR : : : : : : 55279 6607 family 1 5 Idiosepiidae : : : : : : 82764 82761 family 1 5 Idoteidae BIOINFO3 - Lezione 2 10 UN ALTRO ESEMPIO MGI:11945 Ablim1 actin-binding LIM protein GDB:7173461 MGI:87902 Acta1 actin, alpha 1, skeletal muscle GDB:120535 ACTA1 58 MGI:87909 Acta2 actin, alpha 2, smooth muscle, aorta GDB:125197 ACTA2 59 MGI:87904 Actb actin, beta, cytoplasmic GDB:118964 ABLIM1 3983 ACTB 60 ABLIM1 3983 Quale è il separatore di record? Quale è l’identificatore (separatore) di campo? MGI:11945 Ablim1 actin-binding LIM protein GDB:7173461 MGI:87902 Acta1 actin, alpha 1, skeletal muscle GDB:120535 ACTA1 58 MGI:87909 Acta2 actin, alpha 2, smooth muscle, aorta GDB:125197 ACTA2 59 MGI:87904 Actb actin, beta, cytoplasmic GDB:118964 ACTB 60 Le righe (record) sono separate dal carattere NEW-LINE (vai a capo) I campi sono separati dal carattere TAB (tabulazione) BIOINFO3 - Lezione 2 11 INDICIZZAZIONE Un flat-file biologico può contenere migliaia o milioni di record. Sarebbe assurdo, ogni volta che si ha bisogno di un record, scandire tutto il file dall’inizio alla fine per ricercarlo (ad esempio cercando nel campo ID il valore 82764) Viene fatta allora una indicizzazione, cioè si preparano degli indici, con tutti i possibili valori dei vari campi (o almeno dei principali) e la posizione corrispondente nel file Gli indici sono dei file molto più piccoli. Le ricerche sono effettuate solo sugli indici e risultano quindi più veloci. Gli indici in generale vengono costruiti ordinati BIOINFO3 - Lezione 2 12 ESEMPIO Indicizzare il seguente flat-file contenente i dati anagrafici di alcune persone. Quanti record? Quali campi? Qual’è il campo chiave? 1 > ID=1 NOME=MARIO SESSO=M 33 > ID=2 NOME=LUIGI SESSO=M 66 > ID=3 NOME=MARIO SESSO=M 100 > ID=4 NOME=MARIA SESSO=F BIOINFO3 - Lezione 2 ID 1:1 2:33 3:66 4:100 LUIGI:33 NOMI MARIA:100 MARIO:1,66 F:100 SESSO M:1,33,66 13 SRS Anche SRS funziona nel modo che abbiamo appena visto. I singoli database sono indicizzati e le ricerche sono effettuate sugli indici. DB1 4 2 FORM DI QUERY 1 … SRS PAGINA DI RISPOSTA PC UTENTE RETE BIOINFO3 - Lezione 2 Indici DB1 3 DBn 6 0 5 Indici DBn COMPUTER „SERVER“ REMOTO 14 SVANTAGGI Ogni volta che si modifica, o si aggiunge, o si cancella un record è necessario ripetere l’indicizzazione Il sistema è usato solo per le ricerche. Non esiste un sistema diretto per modificare il database BIOINFO3 - Lezione 2 15 DATABASE RELAZIONALI Esiste un DBMS (DataBase Management System) che si preoccupa di gestire fisicamente l’aggiunta, la modifica e la cancellazione dei record e la gestione degli indici. Non ci interessa il modo in cui ciò avviene realmente Il DBMS funge da interfaccia verso il database, in una tipica configurazione CLIENT-SERVER. Il server è residente su un computer remoto, mentre i client sono in generale altri computer richieste risultati CLIENT BIOINFO3 - Lezione 2 RETE DBMS DB SERVER 16 ORGANIZZAZIONE CLIENT-SERVER E’ un’organizzazione piuttosto comune e naturale nell’informatica. Esiste una risorsa, gestita da un server ed un certo numero di utenti (client), che necessitano della risorsa. In genere i client si mettono in coda per accedere alla risorsa. Quando il server è libero ascolta la richiesta del primo cliente in coda, se possibile la esegue restituendo al client quanto richiesto Alcuni esempi di client-server della vita comune? BIOINFO3 - Lezione 2 17 SQL Structured Query Language E’ sicuramente il DBMS più diffuso. SQL è uno standard di cui esistono alcune implementazioni ORACLE (commerciale) MySQL (free) SQL e` un linguaggio ANSI (American National Standars Institute) standard per l`accesso e la manipolazione di database. I comandi SQL sono usati per recuperare, aggiornare, immagazzinare dati all`interno di un database. Sfortunatamente esistono differenti versioni di SQL, ma per rispettare gli ANSI standard, devono supportare la maggior parte delle parole chiave in un modo simile ( come SELECT, UPDATE, DELETE, INSERT, WHERE ...) BIOINFO3 - Lezione 2 18 SQL PIU’ IN DETTAGLIO SQL è un linguaggio attraverso cui è possibile comunicare col database (DBMS=interfaccia tra database ed utente) SQL permette di: Definire la struttura del database (struttura dei dati). E’ un DDL (Data Definition Language) Interagire con i dati, manipolarli. E’ un DML (Data Manipulation Language) In questa settimana vedremo meglio entrambi gli aspetti BIOINFO3 - Lezione 2 19 DATABASE RELAZIONALE Un DATABASE RELAZIONALE è un insieme di TABELLE (table), in origine chiamate relazioni, con un qualche collegamento logico tra di esse. Una tabella di un DB relazionale è l’equivalente di un flat-file tabelle database relazionale La progettazione di un DB è guidata dal mondo reale. In genere si vuole creare un modello di qualcosa (banca, traffico aereo, sistema biologico,…). Si tratta di capire quali entità devono essere rappresentate ed in genere si costruisce una tabella per ciascuna di esse BIOINFO3 - Lezione 2 20 RECORD Una tabella è un contenitore di RECORD Se la tabella rappresenta un’entità del mondo reale, ogni record rappresenta un’istanza di quell’entità Esempio delle automobili. Se definiamo una tabella di automobili, avremo un record per ogni modello di automobile Se definiamo una tabella di sequenze di proteine, avremo un record per ogni sequenza di proteine Possiamo pensare ad una tabella come formata da tante righe. Ogni riga rappresenta un record della tabella BIOINFO3 - Lezione 2 telethonin record actin titin calmodulin Tabella “proteine” 21 CAMPI A sua volta ogni record è composto da un certo numero di campi (fields). Ogni campo rappresenta un attributo dell’entità da modellare. Un campo può contenere valori solo di un certo tipo (numeri interi, numeri reali, date, stringhe di caratteri,…) Ad esempio il nome del modello di automobile, il suo prezzo, l’anno di produzione ecc… In una tabella di un db relazionale tutti i record sono formati dagli stessi campi. Eventualmente un campo di un record potrà anche non essere definito. In questo caso si usa il valore speciale NULL (nei flat-file invece determinati campi potevano tranquillamente essere non definiti per un record). Se i record rappresentano le righe di una tabella, i campi ne rappresentano le colonne BIOINFO3 - Lezione 2 22 ESEMPIO DI UNA TABELLA id marca modello prezzo 1 FIAT 126 3500 2 FIAT 500 4000 3 LANCIA beta NULL \ Esercizio 5. Dato il flat-file qui a fianco convertirlo in una tabella di un database relazionale. Dare un nome alla tabella ed un nome per ogni campo. Raffigurare tutti i record in una matrice (una riga per ogni record, una colonna per ogni campo) BIOINFO3 - Lezione 2 > ID=1 NOME=MARIO SESSO=M > ID=2 NOME=LUIGI > ID=3 NOME=MARIO SESSO=M > ID=4 NOME=MARIA SESSO=F 23 BIOINFO3 - Lezione 2 24
Scaricare