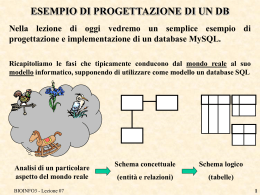
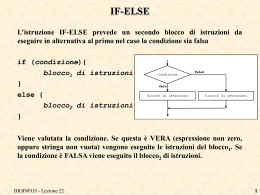
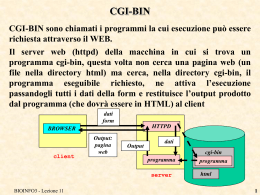
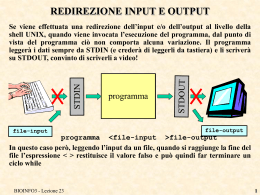
ESERCIZIO
Dato un programma con la sola istruzione:
$a=“Hasta la vista!”
Quanto vale length($a) ?
15
substr($a,0) ?
Hasta la vista!
substr($a,$b) ?
Hasta la vista!
length(substr($a,$c,1) ) ?
1 (ovvio!)
substr($a,lenght($a)-1) ?
!
index ($a,’ ‘) ?
5
index ($a,’ ‘) ?
-1
BIOINFO3 - Lezione 38
1
ESERCIZIO (TIPO ESAME?)
Dato un programma con la sola istruzione:
$a=10;
Quanto valgono $b e $a dopo $b=++$a;
?
$a=11 e $b=11
Quanto valgono $b e $a dopo un ulteriore
$b=$a++; ?
$a=12 e $b=11
Quanto vale $c dopo $c=“Formula $b”; ?
$c=“Formula 11”
Quanto vale $d dopo $d=‘Formula $b’; ?
$d=“Formula $b”
Quanto vale $c dopo $c.=$d ?
$c=“Formula 11 Formula $b”
BIOINFO3 - Lezione 38
2
ESERCIZIO (TIPO ESAME?)
Quante volte viene eseguito il seguente ciclo? Quali numeri stampa?
for ($i=0;$i<10;$i){
print “$i\n”;
}
Il ciclo non termina mai perché $i non viene mai modificata e non sarà mai > o
= a 10. Stampa una colonna interminabile di 0, finchè non viene ucciso con ^C
Quante volte viene eseguito il seguente ciclo? Quali numeri stampa?
for ($i=0;$i<10;$i++){
print “$i\n”;
$i++;
}
Stampa i numeri pari da 0 a 8. Il ciclo è eseguito 5 volte!
Primo ciclo $i=0 e alla fine del ciclo $i=2 …
Quinto ciclo $i=8 e alla fine $i=10;
BIOINFO3 - Lezione 38
3
ESERCIZIO
Leggere un file di sequenze EST in formato FASTA. Considerare solo le righe
che contengono il pattern “>gi|numero”. Inserire in un array questi numeri.
Copiare l’array in altri 2 array. Eseguire un ciclo su tutti gli elementi di questi
2 array incrementando di 1 tutti i numeri del primo e raddoppiando tutti i
numeri del secondo
BIOINFO3 - Lezione 38
4
Il pattern che ci interessa è descritto dall’espressione regolare
/^>gi\|(\d+)/
Ovvero tutte le stringhe che iniziano con > seguito da gi e da un | (ricordarsi di
anteporre \ davanti a |, altrimenti conserva il suo significato di alternativa).
Segue quindi un numero di almeno una cifra.
Ci interessa catturare questo numero, pertanto racchiudiamo il pattern
relativo tra parentesi. In esecuzione ritroveremo la parte di stringa che
soddisfa il pattern tra parentesi nella variabile di perl $1. N.B. Non è possibile
fare assegnamenti a $1. Se avessimo avuto altri pattern tra parentesi li
ritroveremo in $2, $3, ecc…
/^>gi\|(\d+)/
>gi|8777287|gb|AB044776.1|
$1=“8777287”
BIOINFO3 - Lezione 38
5
Notare come $1, se la riga letta ($r) contiene il pattern descritto
dall’espressione regolare, sia aggiunto alla lista @lista.
BIOINFO3 - Lezione 38
6
Il pattern è ritrovato nella stringa $r, quindi
( $r=~ /^>gi\|(\d+)/ ) è VERA e si
entra dentro all’if per eseguire la push
BIOINFO3 - Lezione 38
7
Per queste tra altre righe lette successivamente il pattern descritto dall’espressione
regolare non viene trovato (infatti non iniziano con il maggiore, non contengono gi|
ed un numero).
Quindi l’espressione ( $r=~ /^>gi\|(\d+)/ ) è FALSA e non si entra nell’if per
eseguire la push.
BIOINFO3 - Lezione 38
8
BREAKPOINT NEL DEBUGGER
Fissa un breakpoint alla riga 11
Nel debugger si possono fissare dei “breakpoint”, tipicamente quando si
devono eseguire dei cicli molto lunghi e non vogliamo premere decine o
centinaia di volte il tasto invio.
b numero-di-linea Fissa un breakpoint alla riga dal numero specificato
c
Fa procedere il programma fino al prossimo breakpoint
L’esecuzione fino al breakpoint ci posiziona alla fine del ciclo di lettura delle
righe del file. Si può notare il contenuto dell’array @lista a fine del ciclo (22
elementi)
BIOINFO3 - Lezione 38
9
L’assegnamento ai 2 array @p e @d crea 2 array identici a @lista, anch’essi di $n
elementi (con indice da 0 $n-1)
BIOINFO3 - Lezione 38
10
$p[$i] prima …
…e dopo
$d[$i] prima …
…e dopo? Perché non
viene il doppio?
Ora viene eseguito un ciclo, controllato dalla variabile $i sui $n elementi dei array
@p e @d. All’interno del ciclo viene incrementato di 1 l’i-esimo elemento di @p
($p[$i]) e raddoppiato l’i-esimo elemento di @d ($d[$i])
BIOINFO3 - Lezione 38
11
Dando un altro comando “c” al
debugger
facciamo
ripartire
l’esecuzione. Non essendoci altri
break-point settati dopo la riga 11,
l’esecuzione procede fino alla fine del
programma. Notare la stampa dei tre
array come desiderato.
BIOINFO3 - Lezione 38
12
ESERCIZIO
Passare al programma un argomento. Verificare che l’argomento sia
esattamente uno, altrimenti far morire il programma con un messaggio
d’errore.
L’argomento è il nome di un file di nomi da aprire in lettura. Se il file non
esiste far morire il programma con un messaggio d’errore.
Leggere con un ciclo while le righe del file una alla volta fino alla fine del file,
inserendo le righe in coda ad una lista.
Stampare la lista ordinata, un elemento per riga (ciclo con l’istruzione
foreach) in un file aperto in scrittura assegnandogli un nome uguale a quello
letto, con in più il suffisso “.ord”
BIOINFO3 - Lezione 38
13
Vediamo passo passo l’esecuzione del programma. L’array @ARGV contiene
gli argomenti passati da linea di comando (N.B. non i nomi dei file di
redirezione)
$ARGV[0] $ARGV[1]
@ARGV
nomi
1
@ARGV in un
contesto scalare
(!=1) vale 2, che è
diverso da 1 e
quindi si entra
nell’if che termina
il programma
BIOINFO3 - Lezione 38
14
Ora @ARGV=(“nominonesiste”)
BIOINFO3 - Lezione 38
15
BIOINFO3 - Lezione 38
16
Ora @lista=(“Marta”,”Anna”)
BIOINFO3 - Lezione 38
17
Ora @lista=(“Marta”,”Anna”,”Lucia”)
BIOINFO3 - Lezione 38
18
Alla fine della lettura delle righe del file
@lista=(“Marta”,”Anna”,”Lucia”,”Elda”,
”Irma”,”Sonia”)
La lettura successiva restituisce la stringa
vuota ($riga=<I>=“”) cioè falso e quindi si
esce dal while
BIOINFO3 - Lezione 38
19
Dopo l’istruzione di sort
@lista=(“Anna”,” Elda”,” Irma”,
”Lucia”,” Marta”,”Sonia”)
Si apre quindi il file nomi.ord in
scrittura (viene creato se non esiste)
associandolo alla handle O
O
nomi.ord
BIOINFO3 - Lezione 38
20
1
@lista=(“Anna”,” Elda”,” Irma”,
”Lucia”,” Marta”,”Sonia”)
Il foreach esegue un ciclo per ogni
elemento della lista quindi farà 6 cicli.
In ogni ciclo $a prende il valore di
uno degli elementi della lista.
Le istruzioni di print sono eseguite
sulla handle O e quindi sul file
“nomi.ord”
BIOINFO3 - Lezione 38
2
3
21
4
Terminati i 6 cicli il
foreach termina non
trovando più altri
elementi in @lista
Si può poi verificare il
contenuto del file
nomi.ord
BIOINFO3 - Lezione 38
5
6
22
ESEMPIO (Esercitazione 5 – es. 3)
Leggere un file di nomi (uno per riga) e stampare la frequenza dei nomi
(quante volte compare ogni nome). Suggerimento: usare un array associativo
avente come indici i nomi e come valori la frequenza
%freq
Marco
Matteo
Luca
3
1
6
L’array associativo è indicato come %freq nel suo complesso.
Quanto vale $freq{Matteo}?
1
Quanto vale $freq{Marco}?
3
Quanto vale $freq{Giovanni}?
Quanto vale keys (%freq) ?
0 oppure la stringa vuota (dipende dal contesto)
(“Marco”,”Matteo”,”Luca”)
Quanto vale sort ( keys (%freq)) ?
BIOINFO3 - Lezione 38
(“Luca”,” Marco”,” Matteo”)
23
BIOINFO3 - Lezione 38
24
$riga dopo il chop vale “Luca”
$freq{$riga}=$freq{Luca}=0
$freq{$riga}++ equivale a
$freq{$riga}= $freq{$riga}+1
$freq{Luca}++ equivale a
$freq{Luca}=$freq{Luca}+1=0+1=1
Luca
BIOINFO3 - Lezione 38
%freq
1
25
%freq
BIOINFO3 - Lezione 38
Luca
Marco
2
1
26
Alla fine del ciclo di lettura
%freq
BIOINFO3 - Lezione 38
Luca
Matteo
Marco
5
1
3
27
BIOINFO3 - Lezione 38
28
ESEMPIO (Esercitazione 6 – Esercizio 2)
Leggere tutti i nomi dei file della directory corrente, inserirli in una lista solo
se non contengono il carattere “.”. Ordinare e stampare la lista (un nome per
riga)
Il file . indica la directory corrente
Il file .. indica la directory padre
della corrente
L’opendir è l’equivalente della open
per le directory anziche per i file
normali
I nomi dei file contenuti nella
directory vengono letti con la
funzione readdir anziché con < >,
sempre facendo riferimento alla
handle con cui abbiamo aperto la
directory
BIOINFO3 - Lezione 38
29
Pattern matching: VERO se $f non contiene punti.
/\./ indica tutte le stringhe che contengono punti
Con !~ il pattern matching è VERO se la stringa $f
non contiene punti (si fa il not della condizione)
Il primo file letto è
“.” che però non
verifica la
condizione dell’if.
Analogamente per
il secondo: “..”
BIOINFO3 - Lezione 38
30
Poi si continua a
ciclare. Tutti i file $f
che contengono “.”
non fanno entrare
nell’if
BIOINFO3 - Lezione 38
31
Dopo molti cicli in
cui vengono letti file
contenenti un punto
finalmente si trova
un file, “sequenze”
senza il punto che
viene inserito
nell’array @lista
BIOINFO3 - Lezione 38
32
Finalmente
si
riesce
a
terminare l’elenco dei file e
quindi a far diventare falsa
l’istruzione $f=readdir(D).
A questo punto @lista contiene
7 nomi di file: (“sequenze”,
“nomi”, “numeri”, “nomi1”,
“nomi2”, “seqfasta”, “nn”)
BIOINFO3 - Lezione 38
33
1
2
Dopo l’esecuzione della sort @lista
contiene diventa: (“nn”, “nomi”,
“nomi1”, “nomi2”, “numeri”,
“seqfasta”, “sequenze” )
Si eseguono quindi 7 cicli col
foreach usando la variabile $f
come indice del ciclo. $f viene in
ogni ciclo stampata sullo STDOUT
ovvero a video
BIOINFO3 - Lezione 38
3
4
5
34
Al
termine
del
programma avremo
6
7
BIOINFO3 - Lezione 38
35
Scaricare