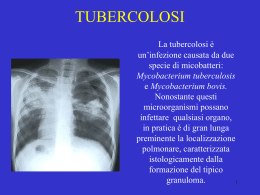
GENOMICA DEL BACILLO TUBERCOLARE Laura Rindi Dipartimento di Patologia Sperimentale, Biotecnologie Mediche Infettivologia ed Epidemiologia - Università di Pisa GENOMICA branca della biologia molecolare che si occupa dello studio del genoma degli organismi studio della struttura, contenuto, funzione ed evoluzione del genoma Genomica comparativa → approccio che consente l’identificazione di variazioni genetiche tra gli organismi che possono spiegare differenze nella fisiologia, biochimica e virulenza Nature 393, 537-544, 1998 Deciphering the biology of Mycobacterium tuberculosis from the complete genome sequence S. T. Cole, et al. …….The genome comprises 4,411,529 base pairs, contains around 4,000 genes, and has a very high guanine + cytosine content that is reflected in the biased amino-acid content of the proteins. M. tuberculosis differs radically from other bacteria in that a very large portion of its coding capacity is devoted to the production of enzymes involved in lipogenesis and lipolysis, and to two new families of glycine-rich proteins with a repetitive structure that may represent a source of antigenic variation. Circular map of the chromosome of M. tuberculosis H37Rv Positions of stable RNA genes (tRNAs are blue, others are pink) and the direct repeat region (pink cube); PPE family members (green); Scale in Mb, with 0 representing the origin of replication. coding sequence by strand (clockwise, dark green; anti-clockwise, light green); PE family members (purple, excluding PGRS); PGRS sequences (dark red) G + C content, with <65% G + C in yellow, and >65% G + C in red. repetitive DNA (insertion sequences, orange; 13E12 REP family, dark pink; prophage, blue); Nature 393, 537-544, 1998 Deciphering the biology of Mycobacterium tuberculosis from the complete genome sequence S. T. Cole, et al. + 82 geni Microbiology 148, 2967-2973, 2002 Re-annotation of the genome sequence of Mycobacterium tuberculosis H37Rv J. C. Camus, et al. ... Here the complete re-annotation of the genome sequence of Mycobacterium tuberculosis strain H37Rv is presented almost 4 years after the first submission. Eighty-two new proteincoding sequences (CDS) have been included and 22 of these have a predicted function. .. The functional classification of 643 CDS has been changed based principally on recent sequence comparisons and new experimental data from the literature. More than 300 gene names and over 1000 targeted citations have been added and the lengths of 60 genes have been modified. Presently, it is possible to assign a function to 2058 proteins (52% of the 3995 proteins predicted) and only 376 putative proteins share no homology with known proteins and thus could be unique to M. tuberculosis. CARATTERISTICHE DEL GENOMA DI M. TUBERCULOSIS H37Rv 4.411.532 bp G+C 65.6% 4.000 geni codificanti proteine 50 geni codificanti RNA stabile 52% funzione definita 48% funzione ipotetica o sconosciuta Oltre il 51% dei geni è derivato dalla duplicazione genica Il 3.4% del genoma è composto da sequenze di inserzione e profagi CLASSIFICAZIONE FUNZIONALE DEI GENI DI M. TUBERCULOSIS H37Rv CLASSE FUNZIONE NUMERO DI GENI 0 Virulence, detoxification, adaptation 99 1 Lipid metabolism 233 2 Information pathways 229 3 Cell wall and cell processes 708 4 Stable RNAs 50 5 Insertion sequences and phages 149 6 PE and PPE proteins 170 7 Intermediary metabolism and respiration 894 8 Proteins of unknown function 272 9 Regulatory proteins 189 10 Conserved hypothetical proteins 1051 METABOLISMO LIPIDICO 8% del genoma è dedicato al metabolismo lipidico (oltre 200 enzimi rispetto ai 50 di E.coli) Lipid metabolism. Degradation of host-cell lipids is vital in the intracellular life of M. tuberculosis. Host-cell membranes provide precursors for many metabolic processes, as well as potential precursors of mycobacterial cellwall constituents, through the actions of a broad family of b-oxidative enzymes encoded by multiple copies in the genome. These enzymes produce acetyl CoA, which can be converted into many different metabolites and fuel for the bacteria through the actions of the enzymes of the citric acid cycle and the glyoxylate shunt of this cycle. PROTEINE PE E PPE 7% dei geni codifica per due nuove famiglie di proteine ricche in glicina Ruolo immunologico SEQUENZE DI DNA RIPETUTE Duplicazioni di geni/famiglie di geni Sequenze di inserzione Sequenze non codificanti disperse Sequenze di DNA ripetute: SEQUENZE DI INSERZIONE Le sequenze di inserzione (IS) sono piccoli segmenti di DNA (<2.5kb) in grado di inserirsi in siti multipli del genoma. transposasi Il genoma di M. tuberculosis H37Rv contiene 56 copie di elementi IS appartenenti ad almeno 9 famiglie. FAMIGLIA IS MEMBRI IN M. TUBERCULOSIS IS3 IS6110 (16), IS1540, IS1604 IS5 IS1560, IS1560’, IS-like (2) IS21 IS1532, IS1533, IS1534 IS30 IS1603 IS110 IS1547 (2), IS1558, IS1558’, IS1607, IS1608’ (2) IS256 IS1081 (6), IS1552’, IS1553, IS1554 IS1535 IS1535, IS1536, IS1537, IS1538, IS1539, IS1602, IS1605’’ ISL3 IS1555, IS1557 (2), IS1557’, IS1561’, IS1606’’ ignota IS1556 Ad eccezione di IS6110, che traspone “frequentemente”, gli elementi IS sono stabili in H37Rv e in altri isolati. Sequenze di DNA ripetute: SEQUENZE NON CODIFICANTI DISPERSE Locus DR : direct repeat. Sequenze ripetute di 36 bp separate da sequenze non ripetute (spacers) di 36-41 bp MIRU : mycobacterial interspersed repetitive unit. 41 loci dimensioni 40-100 bp http://genolist.pasteur.fr/TubercuList ORGANISM SIZE GC CONTENT PUBLICATION Mycobacterium tuberculosis H37Rv (lab strain) 4411 Kb 4060 orfs 65.6 Nature 393,537-544 1998-06-11 Mycobacterium leprae TN 3268 Kb 2749 orfs 57.8 Nature 409, 1007-1011 2001-02-22 Mycobacterium tuberculosis CDC1551 (Oshkosh) 4403 Kb 4346 orfs 65.6 J Bacteriol 184, 5479-90 2001-10-02 Mycobacterium bovis AF2122/97(spoligotype 9) 4345 Kb 4012 orfs 65.6 PNAS 100, 7877-7882 2003-06-24 Mycobacterium avium paratuberculosis K-10 4829 Kb 4415 orfs 69.3 PNAS 102, 12344-9 2004-01-30 Mycobacterium sp MCS 5705 Kb 5752 orfs 68 Unpublished 2006-06-09 Mycobacterium smegmatis MC2 155 6988 Kb 6978 orfs 67.4 Unpublished 2006-11-20 Mycobacterium avium 104 5475 Kb 5339 orfs 69 Unpublished 2006-11-20 Mycobacterium ulcerans Agy99 5631 Kb 4291 orfs 65.5 Genome Res 17, 192-200 2006-12-01 Mycobacterium sp KMS 5737 Kb 6133 orfs 68.4 Unpublished 2006-12-20 Mycobacterium vanbaalenii PYR-1 6491 Kb 6092 orfs 67.8 Unpublished 2006-12-27 Mycobacterium bovis BCG Pasteur 1173P2 4374 Kb 4033 orfs 65.6 Unpublished 2007-01-08 Mycobacterium sp JLS 6048 Kb 5899 orfs 68 Unpublished 2007-02-27 Mycobacterium flavenscens (gilvum) PYR-GCK 5619 Kb 5723 orfs 67 Unpublished 2007-04-12 Mycobacterium tuberculosis H37Ra 4419 Kb 4132 orfs 65.6 PLoS ONE 3, e2375 2007-06-01 Mycobacterium tuberculosis F11 (ExPEC) 4424 Kb 4050 orfs 65.6 Unpublished 2007-06-07 Mycobacterium abscessus CIP 104536 5067 Kb 5041 orfs 64 Unpublished 2008-03-01 Mycobacterium marinum M, ATCC BAA-535 6636 Kb 5550 orfs 65 Genome Res. Epub 2008-04-15 http://www.genomesonline.org/gold.cgi 34 ceppi micobatterici completamente sequenziati 47 ceppi del complesso tubercolare in corso di sequenziamento M. tuberculosis complex M. africanum M. canettii M. microti M. tuberculosis H37Rv CDC1551 H37Ra in corso in corso in corso M. bovis AF2122/97 4.32 Mb M. bovis BCG BCG-Pasteur 4.31Mb 4.41 Mb Tutti i micobatteri appartenenti al complesso tubercolare condividono il 99.9% di identità a livello nucleotidico, ma differiscono ampiamente in termini di spettro d’ospite e di patogenicità PLASTICITA’ DEL GENOMA polimorfismi di singoli nucleotidi eventi di inserzione e delezione J. Bacteriol, 184, 5479-90, 2002 PNAS, 100, 7877-82, 2003 Whole-Genome Comparison of Mycobacterium tuberculosis Clinical and Laboratory Strains The complete genome sequence of Mycobacterium bovis R. D. Fleischmann et al. T. Garnier et al. Caratteristiche M. tuberculosis H37Rv M. tuberculosis CDC1551 M. bovis AF2122/97 4.411.532 4.403.836 4.345.492 65.6 65.6 65.6 3.995 4.249 3.951 polimorfismi singoli - 1135 2348 delezioni - 72 117 inserzioni - 63 108 Dimensioni del genoma, bp G+C, % Geni codificanti per proteine Rispetto a Mtb H37Rv: Il genoma di M. bovis AF2122/97 (identico per oltre il 99.5% a quello di M tuberculosis H37Rv) rispetto a quelli dei due ceppi tubercolari è più piccolo di 70 kb e contiene circa 60 geni in meno. Il 55% delle inserzioni e delezioni tra i due ceppi tubercolari riguardano geni, soprattutto quelli codificanti per le proteine PE e PPE. La variabilità tra M. bovis e Mtb riguarda prevalentemente componenti della parete cellulare e proteine di secrezione. Regioni genomiche che differiscono tra M. tuberculosis H37Rv e M. bovis BCG Pasteur (Esat-6, CFP-10) (phiRv2) (fosfolipasi C) (phiRv1) (invasina) Brosch et al. 2002 PNAS 99:3684-9. Scheme of the proposed evolutionary pathway of the tubercle bacilli illustrating successive loss of DNA in certain lineages (gray boxes). The scheme is based on the presence or absence of conserved deleted regions and on sequence polymorphisms in five selected genes. Note that the distances between certain branches may not correspond to actual phylogenetic differences calculated by other methods. Blue arrows indicate that strains are characterized by katG463. CTG (Leu), gyrA95 ACC (Thr), typical for group 1 organisms. Green arrows indicate that strains belong to group 2 characterized by katG463 CGG (Arg), gyrA95 ACC (Thr). The red arrow indicates that strains belong to group 3, characterized by katG463 CGG (Arg), gyrA95 AGC (Ser), as defined by Sreevatsan et al. Evoluzione del complesso tubercolare M. bovis X M. tuberculosis Evoluzione del complesso tubercolare M. bovis M. tuberculosis bacillo progenitore Identificazione rapida del bacillo tubercolare RDcan M. canettii “ancestral” RD 9 TbD 1 katG 463 CTGCGG RD9 + gyrA 95AGCACC eg. Beijing cluster “modern” eg. Haarlem cluster M. tub. eg. H37Rv RD 7 TbD1 - RD 8 RD 10 M. africanum mmpL6 551AACAAG RDmic M. microti RDseal seal oxyR n285 GA RD 12 oryx RD 13 pncAc57CACGAC goat RD 4 RD 1 RD 2 “classical” BCG Tokyo RD 14 BCG Pasteur M. bovis Identificazione rapida del bacillo tubercolare RDcan M. canettii RD 9 TbD 1 katG 463 CTGCGG RD9 - gyrA 95AGCACC “modern” “ancestral” M. tub. RD 7 RD 8 RD 10 M. africanum mmpL6 551AACAAG RDmic M. microti RDseal seal-isolates oxyR n285 GA RD 12 oryx-isolates RD 13 pncAc57CACGAC goat-isolates RD 4 RD 1 RD 2 “classical” BCG Tokyo RD 14 BCG Pasteur M. bovis Identificazione rapida del bacillo tubercolare RDcan M. canettii RD 9 TbD 1 katG 463 CTGCGG RD9 - mmpL6 551 gyrA 95AGCACC AAG “modern” “ancestral” M. tub. RD 7 RD 8 RD 10 M. africanum mmpL6 551AACAAG RDmic M. microti RDseal seal-isolates oxyR n285 GA RD 12 oryx-isolates RD 13 goat-isolates pncA 57CACGAC RD 4 RD 1 RD 2 “classical” BCG Tokyo RD 14 BCG Pasteur M. bovis Identificazione rapida del bacillo tubercolare RDcan M. canettii RD 9 TbD 1 katG 463 CTGCGG RD9 - gyrA 95AGCACC “modern” “ancestral” M. tub. RD 7 RD4 - RD 8 RD 10 M. africanum mmpL6 551AACAAG RDmic M. microti RDseal seal oxyR n285 GA RD 12 oryx RD 13 goat pncA 57CACGAC RD 4 “classical” M. RD 1 RD 2 BCG Tokyo RD 14 BCG Pasteur bovis Identificazione rapida del bacillo tubercolare RDcan M. canettii RD 9 TbD 1 katG 463 CTGCGG RD9 - gyrA 95AGCACC “modern” “ancestral” M. tub. RD 7 RD1 - RD 8 RD 10 M. africanum mmpL6 551AACAAG RDmic M. microti RDseal seal oxyR n285 GA RD 12 oryx RD 13 goat pncA 57CACGAC RD 4 “classical” M. RD 1 RD 2 RD 14 BCG bovis J. Clin. Microbiol. 41, 1637-1650, 2003 PCR-Based Method To Differentiate the Subspecies of the Mycobacterium tuberculosis Complex on the Basis of Genomic Deletions R. C. Huard, et al. The composite MtbC PCR typing panel. Illustrated are the typical MtbC PCR panel typing results for a single representative of each MtbC subspecies as well as MOTT (M. avium subsp. avium is shown). Lanes: 1, 16S rRNA; 2, Rv0577; 3, IS1561'; 4, Rv1510 (RD4); 5, Rv1970 (RD7); 6, Rv3877/8 (RD1); 7, Rv3120 (RD12). EVOLUZIONE DI MYCOBACTERIUM TUBERCULOSIS Gagneux S. et.al. PNAS 2006;103:2869-2873 ERA POST-GENOMICA CARATTERIZZAZIONE GENOMICA GENOMICA COMPARATIVA Identificazione di fattori di virulenza (confronto genoma Mtb/BCG) e di antigeni (proteine PE/PPE) →comprensione dei meccanismi di patogenicità e sviluppo di nuovi vaccini Allestimento di test diagnostici rapidi (identificazione delle specie del complesso tubercolare; diagnosi immunologica di infezione latente) Identificazione di molecole essenziali, potenziali bersagli per nuovi farmaci Studi evoluzionistici e di epidemiologia molecolare Sviluppo di migliori strategie di controllo dell’infezione tubercolare
Scaricare