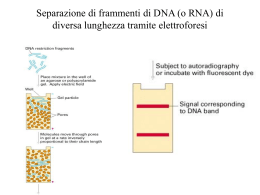
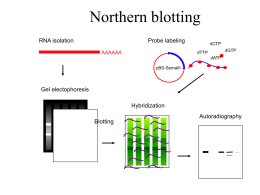
Metodi per studiare l’espressione dei geni • Northern blot • Trascrittasi Inversa -PCR (RT-PCR) • Ibridazione In situ • Trascrizione In vitro • DNA Microarray Purificazione dell’RNA • Procedura – Lisi delle cellule con un reagente che dissocia le nucleoproteine e inibisce la RNAsi – Rimozione delle proteine – Precipitazione specifica dell’RNA Northern blot • Per determinare la dimensione e la quantita’ di specifici mRNA • Studi sull’espressione genica Northern blot – Elettroforesi dell’RNA in gel contenente formaldeide per mantenere l’RNA in forma completamente lineare – Trasferimento dell’RNA su una membrana – Ibridazione con una sonda marcata tampone elettroforetico es. TAE (4mM Tris-acetato, 1mM EDTA, pH 8.0) generatore di corrente gel di agarosio 100V - + 0,2A scatola elettroforetica Gel ElettroforesiSepara le molecole in base alla dimensione Trasferimento su supporto solido Trasferimento dal gel alla membrana Preparare la sonda per il Northern Blot Ibridazione • La sonda marcata e’ aggiunta ad una soluzione contenente il supporto solido che lega l’RNA da analizzare Lavaggio • Rimozione della sonda non legata al supporto solido Rivelazione degli ibridi • Esposizione di un film se la sonda e’ marcata radioattivamente • Se la sonda e’ marcata con un enzima si procede alla reazione enzimatica che produce colore direttamente sul supporto solido Dopo il trasferimento gel membrana Northern blot • La rivelazione avviene usando: – DNA marcato con radioattivo(32P) – DNA marcato con un enzima che catalizza una reazione che produce luce o colore Northern blot Fig. 5. Northern blot analysis of E. lagascae total RNA from leaves (L), germinating seeds (Se), roots (Ro) and stems (St). The blot was hybridized with probes for ElLTP1 (top panel) and ElLTP2 (middle panel). The bottom panel shows ethidium bromide staining of the gel before blotting. The numbers to the left indicate approximate transcript sizes in kb Svantaggi del Northern Blot • Richiede grandi quantita’ di RNA • E’ un processo lungo e laborioso Trascrittasi Inversa- PCR • Rivelazione di mRNA molto rari • Analisi di RNA con pochissime quantita’ Procedura • Purificazione dell’RNA • Aggiungere i primer • mescolare RNA con la trascrittasi inversa per effettuare la sintesi del primo filamento di cDNA • Aggiungere la DNA polimerasi termostabile per effettuare la sintesi del secondo filamento di cDNA e per amplificarlo con i cicli della PCR Trascrittasi inversa - PCR (RT-PCR) mRNA 5’ Primer 1 3’ AAAAA 3’ TTTTT 5’ reverse transcriptase (RNA-dependent DNA polymerase) AAAAA 3’ TTTTT 5’ mRNA 5’ cDNA 3’ Remove RNA (RNase A) cDNA 3’ TTTTT 5´ Add PCR primers Primer 2 5’ 3’ TTTTT 3’ 5’ Primer 3 Add Taq polymerase. Run PCR Reverse transcriptase-PCR (= RT-PCR) Epoxide hydrolase + + + + - - - l 2027 1904 1584 947 831 564 l se r st l se r st pl RT Ibridazione dell’RNA in situ Rivelazione di mRNA direttamente su tessuti messi su vetrini da microscopio Preparation of RNA probe with in vitro transcription T7/T3 or SP6 promoter RNA in situ hybridization Colour substrate: nitro blue tetrazolium (NBT) + 5-bromo-4-chloro-3indolyl phosphate (BCIP) Fig. 7. RNA in situ hybridization with antisense (a-c) and sense (d) RNA probes for ElLTP1. E. lagascae seedlings were collected 7 days after sowing. co Cotyledon, en endosperm, hy hypocotyl, am apical meristem, ro root Northern blotting RNA isolation Probe labeling AAAAAA dCTP dGTP dTTP dATP pBS-SemaIII Gel electophoresis Hybridization Blotting Autoradiography reverse Northern blotting Northern reverse Northern specific probes * * labeled target cDNA arrays (continued) • macro-arrays – low-density – filter-supported – radioactive probes (duplicates) – low sensitivity – only cDNA clones spotted – cheap cDNA arrays (continued) • micro-arrays – high-density – glass-supported – fluorescent probes (single slide) – high sensitivity – ability to spot oligonucleotides – very expensive Tessuto della prostata, normale Tessuto della prostata, tumorale In questo esempio i cDNA sono stati posizionati su di un vetrino, simile ai normali vetrini usati per l’istologia. I microarrays Un microarray è un supporto solido sul quale sono stati posizionati diverse migliaia di cDNA in spot separati. Ciascuno spot rappresenta un gene, in quanto contiene numerose copie di un cDNA corrispondente a tale gene. Microarray technology Probes Microarray synthetized by photolithography or EST/oligo “linking” http://www.ym.edu.tw/excellence/HBP/HBP_CP4/procedure.htm Tessuto della prostata, normale Tessuto della prostata, tumorale utilizzo dei microarrays si confrontano i profili di espressione genica di due campioni differenti. E’ necessario estrarre le molecole di mRNA dai due campioni. Estrazione dell’mRNA dai 2 campioni di cellule che si vogliono confrontare Conversione in cDNA Marcatura con 2 fluorocromi diversi Confronto dei profili di espressione genica in due campioni cellulari diversi (1) (2) (3) (6) Immagine a colori raffigurante il microarray Eccitazione della fluorescenza tramite laser Riconoscimento (4) tra i cDNA provenienti dai 2 campioni e quelli già presenti sul microarray (5) Confronto dei profili di espressione genica in due campioni cellulari diversi I puntini gialli corrispondono a geni che sono espressi in uguale quantità nei due campioni. I puntini verdi corrispondono a geni maggiormente espressi nel campione marcato col fluorocromo che emette fluorescenza verde (ad esempio nelle cellule normali). I puntini rossi corrispondono a geni che sono maggiormente espressi nel campione marcato col fluorocromo che emette fluorescenza rossa (ad esempio nelle cellule tumorali).
Scaricare