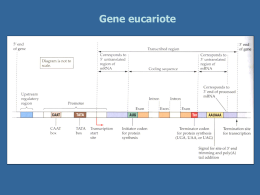
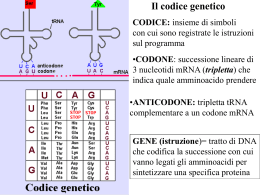
Trascrizione e maturazione degli RNA Trascrizione e traduzione: espressione dell’informazione genica L’RNA veicola l’informazione genica contenuta nel DNA (nucleo) in modo che possa esprimersi •per dare proteine (mRNA) •funzionare direttamente in attività correlate all’espressione genica/sintesi proteica (tRNA e rRNA) •svolgere funzioni varie (ribozimi, small RNA, micro RNA, …) L’RNA è sintetizzato a partire dal DNA nel processo della trascrizione Enzima coinvolto: RNA polimerasi DNA-dipendente; un tratto di filamento di DNA (“gene”) funziona da stampo Vengono incorporati nella catena di RNA nascente dei ribonucleosidi attivati in forma trifosfato (ATP, CTP, GTP, UTP) direzione 5’3’ prima di niziare la trascrizione l’RNA polimerasi riconosce e lega un sito del DNA: promotore RNA polimerasi avanzando sul DNA inserisce ribonucleotidi complementari allo stampo (DNA ss) sulla catena nascente di RNA (direzione 5’3’) La doppia elica DNA si riforma a valle della RNA polimerasi (regione ibrida RNADNA limitata a pochi nucleotidi) L’RNA resta associato all’enzima durante tutta la trascrizione Nei batteri la RNA polimerasi è di un solo tipo ed è formata da varie subunità proteiche Il riconoscimento del promotore è mediato dalla subunità σ Il promotore presenta sequenze conservate (consenso) a – 35 e -10 bp dal sito di inizio di trascrizione Fasi della trascrizione procariotica •Riconoscimento del promotore •Inizio trascrizione ed allungamento •Terminazione determinata da fattore ρ dissociazione dell’enzima dallo stampo e dall’mRNA •Nei batteri gli mRNA possono essere policistronici: portano informazione per più polipeptidi (o rRNA o tRNA) sullo stesso trascritto La terminazione può essere indotta da strutture stem and loop che rallentano la RNA pol In alcuni casi terminazione indipendente da fattore rho A volte l’RNA pol ha bisogno di un attivatore per potersi legare al promotore Maturazione dei trascritti procariotici mRNA maturazione quasi assente traduzione contemporanea a trascrizione (solo in Archea fenomeni di splicing) tRNA e rRNA maturazione mediante taglio di precursori Negli eucarioti l’apparato di trascrizione è + complesso 3 enzimi che riconoscono promotori diversi e sintetizzano classi diverse di RNA RNA pol II è formata da un “core” (con attività polimerasica ) e da varie subunità che agiscono di concerto per riconoscere il promotore, legare il DNA e srotolarlo. Sul promotore viene riconosciuta in particolare la seq TATA box Altre sequenze a monte (anche 200 nt al 5’) possono attivare o inibire la RNA pol mediante interazione con coattivatori o corepressori Altre sequenze ancora più a monte possono attivare/inibire trascrizone (enhancer) Es: il coattivatore CBP induce la trascrizione di tutti i geni con seq attivatrice CREB ( cAMP responsive element) Trascrizione di geni controllata da livelli intracellulari di cAMP Terminazione di trascrizione RNA pol II ed altri fattori tagliano l’mRNA a valle del sito di poliadenialzione rilascio dell’mRNA, degradazione del segmento di RNA 3’ e dissociazione di RNA pol II dallo stampo Maturazione dei trascritti eucariotici trascritto primario o pre-mRNA (hnRNA) •Capping •Metilazione •Poliadenilazione •Splicing •Editing mRNA maturo NUCLEO La maturazione degli RNA è cotrascrizionale Capping L’aggiunta del CAP di metil guanosina (GMP) al 5’P dell’mRNA ne protegge l’estremità evitando degradazione da parte di ribonucleasi e lo posiziona correttamente sul ribosoma durante traduzione Poliadenilazione aggiunta di 30-200 adenosine (AMP) al 3’OH dell’mRNA Stabilizza l’RNAm La poliAdenilazione avviene a valle di sequenza specifica riconosciuta da vari fattori, subito dopo il taglio che termina trascrizione Splicing Rimozione di sequenze prive di significato (non codificanti) o «introni» e risaldatura degli esoni •I componenti dello splicesoma riconoscono siti di giunzione esone/introne/esone •Tagliano al 5’ per dare struttura a cappio (lariat) •Taglio al 3’ dell’introne eliminazione e risaldatura tra i 2 esoni Splicesoma= complesso di proteine e snRNA snRNP: small nuclear ribonucleoprotein particle probabilmente ribozimi NB in alcuni casi si ha self-splicing introne è ribozima RNA Editing Cambiamento nella seq dell’mRNA a livello post-trascrizionale: aggiunta-delezione di nt o cambiamento di basi azotate (UC) •Tipico di geni mitocondriali e di cloroplasti •Richiede stampi di RNA guida (gRNA) originati da introni Es editing tessuto specifico del mRNA per apoliporoteinaB (ApoB) Sunto dei vari step di maturazione di mRNA eucariotico Durante maturazione da hnmRNA ad mRNA accorciamento Solo gli mRNA maturi lasciano il nucleo: selezione a livello del poro nucleare Maturazione degli rRNA eucariotici •rRNA 28S, 18S, 5,8S derivano da singolo precursore trascritto da RNA pol I gene ripetuto in tandem centinaia di volte nella zona NOR zona fibrillare del nucleolo , attivamente trascritta in alcune fasi della vita cellulare •rRNA 5S invece trascritto da RNA pol III – cluster di geni ripetuti fuori dal nucleolo e diverso precursore Maturazione del precursore di rRNA 28S, 18S, 5,8S •Modificazione di nucleotidi (metilazione e sostituzione di U con Ψ) mediata da snoRNA •Associazione con proteine ribosomali •eliminazione degli spaziatori intragenici Maturazione dei tRNA eucariotici Trascritti da RNA pol III che riconosce promotore interno al gene (su tratto codificante) •geni organizzati in cluster •Precursori con geni singoli sono accorciati al 5’ e 3’, subiscono modificazione di basi ed eventuale splicing •La maturazione dei pretRNA coinvolge il ribozima RNasiP CONCETTO DI GENE •anni ’40: esperimenti di Beadle & Tatum Si determina la relazione tra geni ed enzimi grazie a ricerche su alcuni ceppi della muffa del pane (NEUROSPORA ) definiti «mutanti nutrizionali». ALTERAZIONE DNA DIFETTO PROTEINA DIFETTO DI CRESCITA Un gene un enzima •Un gene un polipeptide Sono possibili varie definizione di gene!! 1) Il gene è una sequenza di DNA contenente l’istruzione per la produzione di un polipeptide (fornisce cioè le istruzioni per la sintesi proteica) (questa definizione non tiene conto dei geni per tRNA, rRNA , ribozimi e small RNA) 2) Il gene è un’unità trascrizionale (questa definizione esclude le seq regolatrici e la possibilità che gene dia più prodotti mediante splicing e/o inizi/fine di trascrizione alternativi) 3) Gene: segmento di DNA contenente un’informazione, non sempre univoca 4) Gene: unità ereditaria Elementi fondamentali di un gene: •promotore e regioni regolative, •tratto codificante, •terminatore di trascrizione Negli eucarioti il tratto codificante è “interrotto” da introni!! genomi +ampi Nei procarioti + geni sono trascritti insieme ( mRNA policistronici) The sequence of a prokaryotic proteincoding gene is colinear with the translated mRNA; that is, the transcript of the gene is the molecule that is translated into the polypeptide. The sequence of a eukaryotic protein-coding gene is typically not colinear with the translated mRNA; that is, the transcript of the gene is a molecule that must be processed to remove extra sequences (introns) before it is translated into the polypeptide. Il codice genetico stabilisce la corrispondenza tra seq nucleotidica (DNA RNA) e seq. aminoacidica (proteine) Il codice si basa triplette di basi: CODONI le istruzioni che specificano la sequenza di amminoacidi di un polipeptide sono scritte nel DNA (e sull’mRNA) come codoni 64 combinazioni possibili vs 20aa Seconda base azotata Prima base azotata Quasi tutti gli organismi (dai batteri alle piante agli animali) condividono lo stesso codice genetico. A G Cys U C UUU UUC Phe UCU UCC UUA UCA UAA Stop UGA Stop A UUG UCG UAG Stop UGG Trp G CUU C CUC CUA CUG CCU CCC CAU CAC His CGU CGC U CCA CCG CAA CAG CGA Gln CGG ACU ACC AAU AAC Asn AGU AGC Ser ACA ACC AAA AAG Lys AGA AGG Arg A G GCU GCC GAU GAC Asp GGU GGC U Il codice genetico è universale! C AUU A AUC AUA Piccole differenze solo per geni mitocondriali e per alcuni protozoi, funghi Leu Leu Ile o AUG Met inizio G GUU GUC GUA GUG Val Ser Pro Thr Ala UAU UAC GCA GAA GCG GAG Tyr UGU UGC Arg C A G Glu GGA GGG U C U Gly C A G Terza base azotata U Gli esperimenti di Brenner e Crick su fagi mutanti hanno definito le proprietà principali del codice genetico Basato su triplette di basi (perché inserzioni/delezioni multiple di 3 generalm. non alterano/compromettono significato genico) Non sovrapposto (perché mutazioni di singoli nt cambiano significato di un solo aa) Continuo senza virgole (perché mutazioni per inserzione/delezione di basi provocano slittamento della fase di lettura a valle del sito mutato) Degenerato: con più codoni codificanti per lo stesso aa, ma non ambiguo in q. ad ogni codone corrisponde un solo aa (altrimenti nel tratto mutato i codoni di stop sarebbero molto frequenti) degenerazione evidente sulla terza base Il codice è stato decifrato grazie ad esperimenti di sintesi proteica in vitro usando come stampo RNA sintetici a sequenza nota •Il messaggio sull’mRNA è letto in direzione 5’3’ •il polipeptide è sintetizzato in direzione NH2 COOH (esp di Dintzis) Il codice genetico mette in relazione seq nucleotidica con seq amminoacidica Questo codice molecolare consente di decifrare/tradurre in sequenza aminoacidica l’informazione genetica del DNA Filamento da trascrivere T A C T T C A A A A T C A T G A A G T T T T A G U A G DNA Trascrizione 5’ A U G A A G U U U mRNA Codone di inizio Codone di arresto Traduzione Polipeptide NH2 Met Lys Phe COOH 3’ La traduzione: trasferimento dell’informazione genetica dall’RNA alle proteine – Il messaggio sull’mRNA è tradotto in sequenza amminoacidica – avviene nel citoplasma sui ribosomi (gli organuli che coordinano le operazioni necessarie per passare dalle sequenze nucleotidiche alle catene polipeptidiche). L’apparato di traduzione comprende ribosomi e tRNA che interagiscono con l’mRNA per tradurlo in polipeptide I ribosomi sono complessi formati da rRNA e proteine: rRNA componente strutturale e catalitica più importante Ricerche recenti indicano che gli rRNA hanno attività catalitica (ribozimi) 70S 80S Le subunità ribosomali si pre-assemblano nella regione granulare del nucleolo Le subunità magg e min si associano solo durante sintesi proteica Nobel 2009 Chimica a scienziati che hanno studiato la struttura 3D dei ribosomi applicazioni quali disegno di nuovi antibiotici Nobel 2009 Chimica a scienziati che hanno studiato la struttura 3D dei ribosomi applicazioni quali disegno di nuovi antibiotici Il ribosoma ha 4 siti di legame per RNA : 3 per tRNA (E,P, A) e 1 per mRNA Le due subunità associandosi formano un tunnel nel quale scorre l’mRNA in traduzione (direzione 5’-3’) La subunità maggiore ha attività peptidil-trasferasica (forma legami peptidici tra aa portati dai tRNA) dovuta ad rRNA23S Subunità minore offre piattaforma di appaiamento tra tRNA e mRNA tRNA È un «adattatore»: lega da un lato l’aa (3’OH) dall’altro il codone sull’mRNA (anticodone) 2 regioni cruciali Si ripiega su se stesso e ha 4 regioni di appaiamento interno (doppia elica) Struttura simbolica a trifoglio Struttura 3D ad “L” Il tRNA interagisce con il messaggero a livello del suo anticodone che si appaia con il codone complementare sul mRNA La terza base dell’anticodone è spesso vacillante (wobble) in q puo’ non essere determinante/stringente per l’appaiamento sul codone Vacillamento: la terza base dell’anticodone (al 5’) si può appaiare con più di un nucleotide 2 codoni (mRNA) diversi solo per terza base e specificanti stesso aminoacido possono usare/essere riconosciuti dallo stesso tRNA stesso aa inserito Enzima aminoacil-tRNA sintetasi lega specifico aa al 3’OH del sito accettore sul tRNA corrispondente (la reazione consuma ATP) Di regola esistono almeno 20 enzimi (1 per aa), ma numero varia tra le specie Ridondanza del codice wooble: stesso tRNA (aa) riconosce vari codoni più tRNA (anticodoni) per stesso aa L’enzima aatRNA sintetasi riconosce il tRNA grazie a seq nt specifiche sul tRNA Le 3 fasi della traduzione Inizio: riconoscimento del codone di inizio ed assemblaggio componenti (ribosoma, tRNA e mRNA) Elongazione: sintesi della catena polipeptidica e scorrimento del ribosoma sull’ mRNA Terminazione: codone di stop e dissociazione tra mRNA, ribosoma e polipeptide Inizio traduzione nei procarioti •IF-1-2-3 si legano a subunità minore •Legame di tRNA iniziatore (formil-Met) •mRNA si posiziona su subunità minore grazia appaiamento tra seq Shine-Dalgarno e rRNA 16S •Idrolisi di GTP e associazione di subunità maggiore in eucarioti altri IF legano subunità minore; riconoscimento del 5’ su mRNA grazie a CAP e scorrimento verso 3’ fino a primo AUG Un aatRNA entra in A e si forma il primo legame peptidico (attività peptidil transferasi di subunità maggiore) Elongazione in 4 step: 1) un nuovo aatRNA (scortato da Eftu) si posiziona in A grazie a riconoscimento codone-anticodone;2) la catena polipeptidica in P (C terminale) si lega all’ aa del tRNA in A; 3) subunità maggiore scivola ; 3) subunità minore scivola avanti (vs 3’) di un codone e si libera A il tRNA scarico passa in E si dissocia Il ciclo di elongazione si ripete più volte per inserire altrettanti aa Terminazione •Il codone nonsenso (stop) è riconosciuto da fattori di rilascio •La catena polipeptidica si stacca dal tRNA liberando stremità C; subunità ribosomali si dissociano •Consumo di GTP Lo stesso mRNA viene “letto “ contemporaneamente da più ribosomi che scorrono verso il 3’ poliribosoma Le cellule hanno sistema di sorveglianza che degrada mRNA prodotti da geni con mutazioni non senso premature Il complesso EJC resta associato all’mRNA fino a suo I° ciclo di traduzione Se questa si interrompe prematuramente (mutazione nonsenso:STOP) EJC resta attaccato a mRNA segnale di degradazione dell’mRNA Nei procarioti trascrizione e traduzione sono contemporanee e avvengono in contiguità spaziale In eucarioti trascrizione e traduzione sono fasi distinte temporalmente e spazialmente Differenze nella traduzione eucariotica Nella fase di inizio il posizionamento sul mRNA si basa sul cap e meccanismo di “scansione” dell’mRNA fino a trovare il primo AUG (all’interno della seq Kozak) Molti + fattori di inizio coinvolti mRNA monocistronici vs mRNA policistronici tipici di batteri Le differenze nel processo di traduzione eucariotica e procariotica sono alla base dell’uso di farmaci antibatterici selettivi Molti antibiotici funzionano come inibitori della sintesi proteica batterica (di origine fungina, perché i funghi occupano stesse nicchie ecologiche dei batteri e competono con questi) Ripiegamento, maturazione e traffico delle proteine Prima di svolgere la sua funzione biologica il polipeptide deve ripiegarsi correttamente e, a volte, subire modificazioni chimiche specifiche (legame cofattori, acetilazione, metilazione, glicosilazione, fosforilazione, aggiunta di lipidi, associazione subunità). Il ripiegamento/folding proteico può essere assistito da altre proteine chaperon Molti chaperon altamente conservati tra procarioti ed eucarioti si attivano in condizioni di shock termico (HSP) Alcune patologie neurodegenerative ripiegamento proteico difettoso derivano da Es Alzheimer, Parkinson, derivano da accumulo di fibrille amiloidi (depositi di proteine insolubili malripiegate) morte neuroni Hungtington disease: aggregati proteici di proteina misfolded a causa di mutazione genica Malattia da prioni: neurodegenerazione dovuta ad accumulo di proteina PrP malripiegata Nella encefalopatia spongiforme le proteine PrP malripiegate assunte con l’alimentazione inducono alterazione strutturale delle PrP endogene in una reazione a catena Unico caso noto di agente”infettivo” di natura proteica
Scaricare