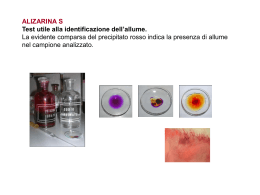
Denaturata per aggiunta del riducente ditiotreitolo Riducente dializzato Aggiunto glutatione ossidato Processo fermato per aggiunta di iodoacetato Che reagisce con cisteina formando Carbossimetilcisteina Proteina eluita e separati i diversi intermedi Ponti 30-51 5-55 14-38 Scambio ionico I tre intermedi con due disolfuri non formano mai il terzo ma si convertono lentamente nel 5-55 30-51 , sono intermedi non-nativ1 Che definiscono il cammino di ripiegamento In loro assenza ( 14 38) la velocità di ripiegamento è fortemente ridotta Il ripiegamento è cooperativo Esistono vie preferenziali al ripiegamento Presenza di intermedi che non sono forme parziali di proteina ripiegata, ma necessitano di riarrangiamento L’intermedio 30-51 è una via obbligata si forma per primo benchè non sia il disolfuro più stabile nella proteina nativa I tre intermedi 30-51,5-14 30-51,5-38 30-51,14-38 non nativi Definiscono una via obbligata alla formazione del native-like 30-51,5-55 lo step lento della reazione Dove C’è un alta barriera energetica k1 kex Folded < = > open < = > H ex k-1 kex>k-1 la rate di scambio è k1 kex<k-1 kexk1/k-1 Il dicroismo circolare (CD) viene osservato quando molecole otticamente attive assorbono diversamente la luce circolarmente polarizzata destra e sinistra. Il dicroismo circolare (CD) viene osservato quando molecole otticamente attive assorbono diversamente la luce circolarmente polarizzata destra e sinistra. a) NMR exchange b) H exchange ESMS c) far UV CD ( 225 nm) d) near UV CD ( 289 nm ) e) intrinsic fluorescence f) quenching by iodide g) binding of ANS h) binding fluorescent Inhibitor andamento bifasico. La fase rapida ha una costante di tempo dell’ordine di 5/10 millisecondi ed un ampiezza media del 40% e del 25% per il dominio α e β rispettivamente. in questa proteina, il ripiegamento del dominio β non può avvenire in assenza di quello del dominio α, mentre non è vero il contrario dicroismo circolare nel lontano ultravioletto diagnostico di percentuale struttura secondaria CD vicino ultravioletto processo monofasico, indicativo Riarrangiamento aromatici Cinetica di ripiegamento monitorata attraverso la fluorescenza Intrinseca andamento bifasico Cinetica di ripiegamento monitorata attraverso il quenching di fluorescenza. Cinetica di legame dell’ANS anilinonaphthalene sulfonate Cinetica di legame di un inibitore fluorescente. Collasso idrofobico iniziale più vie di ripiegamento, in quanto l’ordine di assemblaggio delle diverse strutture secondarie non È identico Processo rapido i IDENTIFICAZIONE DEGLI STATI DI TRANSIZIONE E DEGLI INTERMEDI NEL PROCESSO DEL FOLDING A. Fersht JMB 1992 IPOTIZZARE CATENE IMPORTANTI PER LA STABILITà O LA CINETICA DI RIPIEGAMENTO MODIFICARE LA CATENA PER MUTAGENESI DETERMINARE DA MISURE DI EQUILIBRIO E DI CINETICA IL PROFILO DI ENERGIA PER LA PROTEINA NATIVA E MUTATA VALUTARE IL PARAMETRO Φ DAI VALORI DI ENERGIA MISURATI Effettuare singola mutazione e verificare la differenza di energia libera sia da un punto di vista termodinamico che cinetico FEBS Lett. 1993 325 5-16 J.M.B. 1992 224 771 ΔΔGU-F = ( GU- GF ) – (GU’ - GF’ ) equilibrio ΔΔG#-F= ( G#- GF ) – (G#’ - GF’ ) velocità Φunf = (ΔG#- ΔGF ) / (ΔGu- ΔGF ) ΔΔG#-F / ΔΔGU-F Φ unf= 1 ΔG# = ΔGu regione mutazione in transizione come in denaturata Φunf = 0 ΔG# = ΔGF regione mutazionein transizione come in ripiegata PNAS 1994 91 10422-10425 Inibitore chimotripsina Non ci sono siti con Φfol = 1 in cui regione mut. Ha stato trans. Come in nativa Ovvero siti di nucleazione Ripiegamento con collasso globale assenza regioni preferenziali formazione di alcuni elementi di struttura secondariache in seguito si assemblano tra di loro Ci sono siti di nucleazione Φfol = 1 Un modello di ripiegamento • La topologia è l’elemento determinante il folding infatti: • Grandi cambi nella sequenza sia sperimentali che evoluzionistici, che non alterano la topologia della proteina hanno un basso effetto sulla velocità di folding • Proteine con struttura 3D simile e sequenze differenti hanno stati di transizione di ripiegamento simili • Proteine con una grossa frazione di contatti tra residui vicini in sequenza ( basso ordine di contatto) si ripiegano più rapidamente che proteine con molti contatti non-locali ( alto ordine di contatto) • Baker Nature 2000 405 39-42 ORDINE DI CONTATTO è LA DISTANZA MEDIA IN SEQUENZA TRA TUTTE LE COPPIE DI RESIDUI IN CONTATTO NORMALIZZATA ALLA LUNGHEZZA TOTALE DELLA SEQUENZA CO = Σ Δ SIJ LN Δ S = SEPARAZIONE IN SEQUENZA L = NUMERO TOTALE RESIDUI N = NUMERO TOTALE CONTATTI • Il costo entropico nel formare contatti non locale è alto • la stabilita di una proteina e il meccanismo di ripiegamento sono determinati dalla sequenza aminoacidica, tuttavia la stabilità è sensibile ai dettagli delle interazioni inter-atomiche, mentre il meccanismo di ripiegamento è più sensibile alla topologia • Modelli semplici basati su rappresentazioni binario del polipeptide predicono abbastanza bene le vie del ripiegamento • La proteina precursore della betaamiloide è una grande proteina di membrana che normalmente gioca un ruolo importante nella crescita e nella riparazione dei neuroni. . La APP può essere tagliata in una serie di frammenti funzionali da alcune proteasi specializzate, chiamate secretasi. Il piccolo peptide è chiamato peptide beta-amiloide. Nella proteina intera il peptide si estende attraverso la membrana ancorando la proteina. Ma quando, in seguito all'idrolisi, il peptide diventa libero, lascia la membrana, cambia forma e si aggrega in lunghe fibrille.
Scaricare