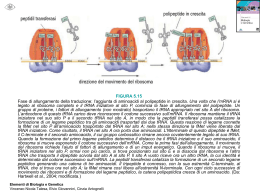
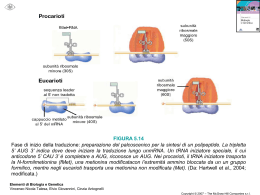
Corso di Laurea in Farmacia Insegnamento di BIOCHIMICA Angela Chambery Lezione 23 Il codice genetico Concetti chiave: • Il codice genetico è basato su insiemi di codoni formati da tre nucleotidi che sono letti in modo sequenziale. • Ciascun codone rappresenta un amminoacido o un segnale di Stop. • Il codice genetico è degenerato, non casuale e pressoché universale. Il codice genetico Poiché i nucleotidi sono solo quattro e gli amminoacidi 20 non è possibile che ci sia una semplice corrispondenza “uno a uno” tra nucleotidi e amminoacidi. Per specificare un singolo amminoacido è necessario quindi un gruppo di più basi detto “codone”. E’ in tal modo possibile avere 43=64 possibili triplette di basi. Il codice genetico Il codice genetico è degenerato ma non sovrapposto Il codice genetico Mediante eleganti esperimenti di Crick e Brenner sul batteriofago T4 si scoprì che una mutazione per delezione di un nucleotide poteva eliminare la funzione di un gene specifico. Tuttavia, una seconda mutazione per inserzione in una posizione diversa era in grado di ripristinare la funzione del gene. Tali mutazioni alterano lo schema di lettura (frameshift mutations). THE BIG RED FOX ATE THE EGG THE IGR EDF OXA TET HEE GG I tre possibili schemi di lettura Ciascuno schema di lettura può determinare la produzione di un polipeptide diverso. Il codice genetico Il codice genetico standard Il codice genetico è degenerato. Solo Met e Trp hanno un solo codone. Arg, Ser e Leu sono specificati da 6 diversi codoni. I codici differiscono soltanto a livello del terzo nucleotide. Le triplette UAG, UAA e UGA sono codoni di stop AUG è il codone di inizio della catena polipeptidica. Il codice genetico standard, anche se ampiamente diffuso, non è universale Il codice genetico standard L’appaiamento di codoni anticodoni è di tipo antiparallelo. e Alcuni tRNA contengono in prima posizione il nucleotide inositato (I), che contiene l’inconsueta base ipoxantina. L’inositato può formare legami idrogeno con tre diversi nucleotidi U, C e A. Lo stesso tRNA può riconoscere vari codoni; il numero di tRNA per ciascun amminoacido non è pertanto necessariamente uguale al numero dei suoi codoni. L’RNA transfer e la sua amminoacilazione Concetti chiave: • Le molecole di tRNA hanno una struttura caratteristica. • Una amminoacil‐tRNA sintetasi carica ogni tRNA con l’appropriato amminoacido. • L’ipotesi di oscillazione spiega la capacità del tRNA di riconoscere più di un codone. Struttura secondaria a forma di trifoglio del tRNA La molecola di tRNA serve come un adattatore che si lega a ciascun codon e che trasporta con sé l’amminoacido destinato a essere inserito nella catena proteica. Sono costituiti da una catena singola e contengono da 73 a 93 ribonucleotidi Contengono molte basi insolite o rare, in genere da 7 a 15 per molecola. Alcune sono derivati metilati o demetilati di A, U, C e G, formati da modificazioni enzimatiche di un tRNA precursore Circa la metà dei ribonucleotidi sono appaiati tra loro, formando tratti a doppia elica. Cinque gruppi di basi non sono appaiati formando anse I tRNA hanno un gruppo fosforico 5’ terminale; il residuo terminale 5’ è di solito pG Struttura secondaria a forma di trifoglio del tRNA Le regioni non appaiate includono: la regione CCA all’estremità 3’, che fa parte dello stelo accettore l’ansa TΨC, che prende il nome dalla sequenza ribotimina‐ pseudouracilecitosina il braccio extra o variabile, che contiene un numero variabile di residui l’ansa diidrouridina (DHU) o braccio D, che contiene numerosi residui di diidrouracile l’ansa dell’anticodone Nucleosidi modificati presenti nei tRNA Nucleosidi modificati presenti nei tRNA Nucleosidi modificati presenti nei tRNA Nucleosidi modificati presenti nei tRNA Sequenza del tRNAPhe di lievito Struttura ai raggi X del tRNAPhe di lievito I tRNA hanno una struttura terziaria complessa. Nel 1974 è stata determinata la prima struttura tridimensionale di un tRNA (Phe‐tRNA) (Rich e Klug). La molecola ha una forma a L in cui lo stelo accettore e lo stelo T formano un segmento della lettera e lo stelo D e quello dell’anticodone formano l’altro. La maggior parte delle basi delle non elicoidali formano regioni egualmente legami idrogeno con appaiamenti diversi da quelli del modello di Watson e Crick. Struttura ai raggi X del tRNAPhe di lievito Ansa TC La struttura dei tRNA è perfettamente adatta al suo ruolo: Estremità CCA l’anticodone è disponibile per interagire con il codone appropriato sull’mRNA l’estremità legata all’amminoacido attivato è in posizione adatta per formare il legame peptidico Ansa DHU Ansa anticodone Amminoacil‐tRNA sintetasi Il legame di un amminoacido e un tRNA è fondamentale: L’attacco di un dato amminoacido a uno specifico tRNA determina il codice genetico La formazione di un legame peptidico tra amminoacidi liberi non è una reazione termodinamicamente favorita. E’ necessario che l’amminoacido sia attivato prima di essere legato in una catena polipeptidica. Nella sintesi proteica gli intermedi attivati sono esteri degli amminoacidi, nei quali il loro gruppo carbossilico è legato al gruppo ‐OH 2’ o 3’ dell’unità di ribosio che si trova all’estremità 3’ del tRNA (amminoacil‐tRNA) Nella prima fase della sintesi proteica i 20 amminoacidi vengono legati mediante legame estere al loro tRNA ad opera di amminoacil‐tRNA sintetasi Generalmente esiste un’amminoacil‐tRNA sintetasi per ciascun amminoacido. Per amminoacidi che hanno due o più tRNA corrispondenti, di solito la stessa amminoacil‐ tRNA sintetasi provvede alla amminoacilazione di tutti. Formazione dell’amminoacil‐adenilato La prima tappa nella formazione di un amminoacil‐tRNA è la formazione di un amminoacil adenilato a partire da un amminoacido e ATP. L’intermedio amminoacido‐AMP non si dissocia dall’enzima. Un amminoacil‐tRNA La tappa successiva è il trasferimento dell’amminoacido a una molecola di tRNA: Amminoacil‐AMP+tRNAamminoacil‐tRNA+AMP La reazione complessiva catalizzata un’amminoacil‐tRNA sintetasi è la seguente: da Mg2+ Aa + tRNA + ATP amminoacil‐tRNA + AMP + PPi 2Pi Amminoacil‐tRNA sintetasi Le amminoacil‐tRNA sintetasi hanno siti di attivazione degli amminoacidi altamente specifici. Una sintetasi aggiunge a un tRNA un amminoacido sbagliato una volta su 104 o 105 reazioni. Ogni sintetasi riconosce specificamente le proprietà dell’amminoacido che funge da suo substrato. La treonil‐tRNA sintetasi ha uno ione zinco che si coordina alla treonina tramite il suo gruppo amminico e il suo gruppo ossidrilico Amminoacil‐tRNA sintetasi La correzione di bozze da parte delle amminoacil‐ tRNA sintetasi aumenta la fedeltà della sintesi proteica Studi strutturali e di mutagenesi hanno dimostrato che molte sintetasi hanno un sito di correzione di bozze situato ad una distanza di circa 20 Å dal sito di attivazione In genere il sito del legame acilico respinge gli amminoacidi più grandi di quelli corretti (non ha spazio sufficiente), il sito di idrolisi scinde le specie attivate più piccole di quelle corrette. Amminoacil‐tRNA sintetasi Il braccio flessibile CCA di un amminoacil tRNA può spostare l’amminoacido tra il sito di attivazione e il sito di correzione di bozze Amminoacil‐tRNA sintetasi In che modo le sintetasi riconoscono e scelgono i tRNA? Questo riconoscimento è responsabile effettivamente della “traduzione”, cioè della correlazione tra amminoacidi e acidi nucleici. Poiché ciascun tRNA ha un anticodone diverso, molte sintetasi riconoscono i rispettivi tRNA soprattutto in base all’anticodone, anche se possono riconoscere altri aspetti della struttura del tRNA. Complesso della treonil‐tRNA sintetasi La sintetasi è legata sia allo stelo accettore sia all’ansa dell’anticodone Amminoacil‐tRNA sintetasi Le amminoacil‐tRNA sintetasi sono state divise in due classi in base alle differenze nella struttura primaria e terziaria e nel meccanismo di azione. • Gli enzimi di classe I formano un legame acilico con il gruppo ossidrilico in posizione 2’ dell’adenosina terminale del tRNA • Gli enzimi di classe II (eccetto quello per la Phe‐tRNA) formano un legame acilico con il gruppo ossidrilico in posizione 3’ Le due classi legano l’ATP in conformazioni diverse. Quasi tutti gli enzimi di classe I sono monomerici, mentre quasi tutti quelli di classe II sono dimerici o multimerici. Classe I 2’ OH Classe II 3’ OH Amminoacil‐tRNA sintetasi Classe I Classe II Classificazione delle amminoacil‐tRNA sintetasi di E. coli GlnRS•tRNAGln•ATP di E. coli GlnRS‐tRNAGln‐ATP di E. coli PDBids 1GTR AspRS•tRNAAsp di lievito AspRS – tRNAAsp di lievito PDBid 1ASY Struttura generale degli amminoacil‐tRNA I ribosomi Concetti chiave: • Il ribosoma consiste di una subunità maggiore e di una minore, entrambe composte da RNA e da un gran numero di piccole proteine. • La complessa struttura del ribosoma gli permette di legare l’mRNA e tre molecole di tRNA e di portare avanti la sintesi proteica. I ribosomi I componenti del ribosoma di E. coli Il ribosoma degli eucarioti è più grande e più complesso Struttura secondaria dell’RNA ribosomiale 16S Ciascuno degli RNA ribosomiali a singola elica possiede una specifica conformazione tridimensionale conferita da appaiamenti delle basi. Struttura secondaria dell’RNA ribosomiale 23S Gli RNA ribosomiali hanno un ruolo centrale nella sintesi proteica Gli RNA ribosomiali sono ripiegati in strutture precise, con numerosi brevi tratti a doppia elica. Per molti anni si è ritenuto che la sintesi proteica fosse organizzata e diretta dalle proteine. L’opinione attuale è invece opposta. I siti attivi del ribosoma sono composti quasi interamente da RNA Il ribosoma ad alta risoluzione Fotografia in criomicroscopia elettronica di un ribosoma Distribuzione delle proteine e dell’RNA nelle subunità ribosomiali RNA ribosomiali PDBids 1J5E e 1JJ2 Ribosoma 80S di lievito La traduzione Concetti chiave: • I fattori d’inizio aiutano ad assemblare le subunità ribosomiali, posizionano il tRNA iniziatore e, negli eucarioti, identificano il codone d’inizio. • Il ribosoma seleziona il corretto amminoacil‐tRNA, catalizza la reazione di transpeptidazione e poi trasloca lungo l’mRNA durante la fase di allungamento della sintesi proteica. • Un fattore di rilascio e il fattore di riciclo del ribosoma partecipano alla terminazione della sintesi del polipeptide. La traduzione Monitorando l’incorporazione di amminoacidi marcati è stato osservato che la sintesi di un polipeptide procede dall’estremità N‐terminale a quella C‐terminale L’iniziazione della catena richiede un tRNA iniziatore Il codone di inizio AUG specifica un residuo di metionina amminoterminale Nei batteri il residuo amminoacidico di avvio è la N‐Formilmetionina, che penetra nel ribosoma associata ad uno specifico tRNA (N‐Formilmetionina ‐ tRNAfMet). Negli eucarioti tutti i polipeptidi iniziano con un residuo di Metionina (invece che fMet), ma anche in questo caso viene usato un tRNA specializzato diverso da quello usato per l’inserimento della metionina nelle posizioni interne. Formilazione del metionil‐tRNA Il tRNA di inizio è prima caricato con la metionina, poi viene trasferito un gruppo formile sul metionil‐tRNA Sequenza Shine‐Dalgarno La sequenza Shine‐Dalgarno viene riconosciuta da una sequenza complementare ricca di pirimidine vicino all’estremità 3’ del’rRNA 16S della subunità 30S. Questa interazione mRNA‐ rRNA immobilizza l’mRNA in modo che l’AUG sia posizionato sulla subunità 30S per l’inizio della traduzione. Un mRNA procariotico può avere più sequenze Shine‐Dalgarno e quindi stiti multipli di inizio. Gli eucarioti non hanno la sequenza Shine‐Dalgarno e il codone AUG più vicino al 5’ dell’mRNA è selezionato come sito d’inizio. Siti di legame del ribosoma per il tRNA I ribosomi hanno 3 siti di legame per il tRNA, localizzati tra le subunità 30S e 50S. Sito A (amminoacidico); Sito P (peptidico) e Sito E (sito di uscita). Siti di legame del ribosoma per il tRNA Meccanismo della sintesi proteica La reazione della peptidil transferasi ribosomiale Il gruppo amminico dell’amminoacil‐tRNA produce un attacco nucleofilo sul gruppo carbonilico del legame estere del peptidil‐tRNA, formando un intermedio tetraedrico. Il centro della peptidil transferasi contiene basi che favoriscono la reazione aumentando la reattività del gruppo NH2 sull’aminoacil‐tRNA nel sito A e stabilizzando l’intermedio tetraedico che si produce L’intermedio si dissocia, generando il legame peptidico e rilasciando il tRNA privo dell’amminoacido La reazione della peptidil transferasi ribosomiale Un peptidil‐tRNA “Via di fuga” del polipeptide La subunità 50S è attraversata da un tunnel che inizia nel sito di formazione del legame peptidico. La catena polipeptidica nascente passa attraverso il tunnel. Un possibile ruolo della formilazione Se il gruppo amminoterminale è libero, il dipeptidil‐tRNA può formare un legame interno, ciclizzandosi e separandosi dal tRNA. La formilazione dell’estremità amminoterminale blocca questa reazione. Fattori proteici solubili necessari per la traduzione Sintesi proteica: inizio L’inizio della sintesi proteica nei batteri richiede: • la subunità ribosomale 30S • l’mRNA • l’fMet‐tRNAfMet • i fattori d’inizio IF‐1, IF‐2 e IF‐3 • GTP • la subunità ribosomale 50S • Mg2+ Il legame di IF‐1 e IF‐3 alla subunità 30S impedisce che quest’ultima si leghi prematuramente alla subunità 50S. Il fattore IF‐2 lega il formil‐ metionil‐tRNAf e contribuisce al suo corretto posizionamento Sintesi proteica: inizio Complesso d’inizio negli eucarioti Gli eucarioti utilizzano molti più fattori di inizio e le loro interazioni sono più complesse. eIF4E lega il 5’cap, PAB lega la coda di poli(A) a formare un mRNA che assume una forma circolare che facilita il legame ai ribosomi. Il fattore di allungamento Tu La molecola di tRNA da inserire nel sito A viene trasportata da una proteina detta fattore di allungamento Tu (EF‐Tu), un membro della famiglia delle proteine G, lega l’amminoacil‐tRNA solo quando ha legato a sé il GTP. EF‐Tu protegge dall’idrolisi il labile legame estere dell’amminoacil‐tRNA. Il GTP legato all’EF‐Tu è idrolizzato solo quando si forma una corretta associazione tra il complesso EFTu e il ribosoma (accuratezza della sintesi proteica). Il fattore di allungamento Ts si unisce al complesso EF‐Tu/GDP e favorisce la dissociazione del GDP. I fattori EF1α e EF1 sono la controparte dei fattori EFTu e EFTs procariotici. Sintesi proteica: allungamento Meccanismo peptidil trasferasico del ribosoma Il gruppo α‐amminico dell’amminoiacido nel sito A agisce da nucleofilo, spostando il tRNA nel sito P per formare un legame peptidico. L’attività enzimatica che catalizza la formazione del legame peptidico viene chiamata peptidil transferasi ed è probabilmente il ribozima dell’rRNA 23S. Traslocazione Il ribosoma si muove di una lunghezza pari a un codone verso l’estremità 3’ dell’mRNA. Il dipeptidil‐tRNA si sposta dal sito A al sito P e il tRNA deacilato viene spostato dall’iniziale sito P al sito E. Il tRNA verrà poi rilasciato dal sito E nel citosol. Lo spostamento del ribosoma richiede EF‐G ed energia fornita dall’idrolisi del GTP. Traslocazione Ef‐G si lega al sito di legame di EF‐Tu sulla subunità 50S L’idrolisi del GTP induce un cambiamento conformazionale in EF‐G che inserisce lo stelo nel sito A della subunità 30S I tRNA e l’mRNA si spostano di una distanza corrispondente a un codone Analogo non idrolizzabile del GTP Terminazione L’allungamento prosegue finché il ribosoma aggiunge l’ultimo amminoacido. La terminazione è segnalata da uno dei tre codoni di terminazione dell’ mRNA (UAA, UAG, UGA), che segue il codone dell’ultimo amminoacido. Terminazione Quando un codone di terminazione occupa il sito A sul ribosoma, tre fattori di terminazione, RF1, RF2 e RF3 provvedono ad effettuare: •Idrolisi del legame terminale del peptidiltRNA •Rilascio del polipeptide libero e dell’ultimo tRNA; •Dissociazione del ribosoma 70S nelle sue subunità 30S e 50S Terminazione RF1 riconosce i codoni UAG e UAA mentre RF2 riconosce i codoni UGA e UAA RF1 o RF2 (a seconda di quale codone è presente) si lega al codone di terminazione e induce la peptidil transferasi a trasferire la catena polipeptidica a una molecola d’acqua invece che a un altro amminoacido La funzione di RF3 è quella di regolare le interazioni di RF1 e RF2 con il ribosoma Negli eucarioti, un unico fattore di rilascio, eRF riconosce tutti e tre i codoni di terminazione. Terminazione RF3 scambia il GDP con il GTP rilasciando RF1 Terminazione Per il rilascio del tRNA e dell’mRNA dal ribosoma 70S è necessario un ulteriore fattore proteico detto fattore di rilascio del ribosoma (RRF) e l’EF‐G che richiedono GTP EF‐G idrolizza il GTP espellendo il tRNA, l’mRNA e gli altri fattori, portando alla dissociazione delle subunità del ribosoma Terminazione Un fattore di rilascio eucariotico ha una struttura simile a quella del tRNA. In corrispondenza dell’estremità della struttura che ricorda lo stelo accettore è presente la sequenza Gly‐Gly‐Gln che lega una molecola di acqua. La molecola di acqua è così trasportata in corrispondenza del centro della peptidil transferasi. Polisoma La traduzione simultanea di un singolo mRNA da parte di numerosi ribosomi permette di utilizzare l’mRNA in modo molto efficiente. Polisoma Accoppiamento della trascrizione e della traduzione nei batteri Ribosomi associati al RE Negli eucarioti i ribosomi sono associati al lume del reticolo endoplasmatico (RER) Ribosomi associati al RE Tutte le proteine integrali di membrana ad eccezione di quelle localizzate sulle membrane di mitocondri e cloroplasti sono sintetizzate sui ribosomi associati all’ER Sorting delle proteine Secrezione proteine extracellulari Le proteine di secrezione vengono trasportate fuori la cellula mediante vescicole di secrezione Diversi antibiotici bloccano la sintesi proteica Il cloramfenicolo blocca la peptidil transferasi batterica, ma non quella eucariotica Le tetracicline bloccano il sito A del ribosoma Modificazioni post‐traduzionali Le catene polipeptidiche neosintetizzate vanno poi incontro a ripiegamenti e modificazioni post‐ traduzionali che includono fosforilazioni, modificazioni ammino‐terminali e carbossi‐terminali, perdita della sequenza segnale, glicosilazioni, aggiunta di gruppi prostetici, modificazioni proteolitiche, formazione di legami disolfuro. Degradazione delle proteine Le proteine vengono degradate mediante il sistema ubiquitina‐proteasoma
Scaricare