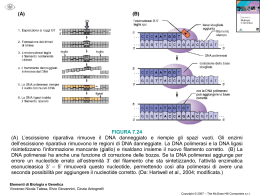
Analisi molecolare dei geni Denaturazione e rinaturazione di una molecola di DNA Si rompono i legami idrogeno 100°C Denaturazione del DNA Rinaturazione per riassociazione delle sequenze complementari Ogni frammento di DNA ha una caratteristica temperatura di fusione (Tm-> melting temperature) … Maggiore e’ il contenuto delle G C, maggiore e’ la Tm Tm-> temperatura alla quale meta’ delle molecole di DNA sono denaturate in singoli filamenti Effetto ipercromico delle basi nucleotidiche, all’aumentare della separazione dei due filamenti aumenta la OD a 260nm Applicazioni sperimentali della complementarietà delle basi Le cinetiche di riassociazione suggeriscono che il DNA umano e’ formato da: fast Cinetica di rinaturazione del genoma umano intermediate slow Fast: DNA altamente ripetuto, presente in numerose copie (trasposoni inattivi, copie geniche inattive, ripetizione di sequenze semplici, sequenze ripetute in tandem, duplicazioni segmentali) Intermediate: DNA mediamente ripetuto Slow: DNA presente in sequenze uniche Composizione del genoma umano PCR (Polymerase Chain Reaction o reazione a catena della polimerasi) 1 Gene a sequenza nota La PCR permette l’amplificazione di un milione di volte o piu’ di una sequenza specifica di DNA che e’ gia’ nota o di cui sono note delle piccole sequenze all’estremita’ del gene 2 La possibilita’ di conoscere i geni deriva dalla capacita’di manipolarli: -isolare un gene (enzimi di restrizione) -clonaggio (amplificazione) vettori -sequenziamento -funzione VETTORE PLASMIDICO -si replica autonomamente rispetto al cromosoma batterico -possiede un polylinker -possiede un marcatore di selezione Vettore di espressione Oligonucleotidi o primers Oligonucleotidi o primers PCR (Polymerase Chain Reaction) DNA polimerasi dNTP Mg2+ Reazione 2n dove n=numero di cicli Si possono ottenere informazioni sulla sequenza a partire da ridottissime quantita’ di DNA come sangue, capelli, etc Southern/Northern Blot Ibridazione con sonda di DNA - PER INDIVIDUARE LA PRESENZA DEL GENE CHE SI STA CERCANDO - PER OSSERVARE LA PRESENZA DI UN TRANSGENE INSERITO NEI CROMOSOMI (PRESENZA O ASSENZA) - PER OSSERVARE TRASLOCAZIONI CROMOSOMICHE - PER OSSERVARE LE DIMENSIONI DEL TARGET (NON OSSERVABILI CON ALTRE TECNICHE) - PER OSSERVARE LA COLLOCAZIONE SUBCROMOSOMICA O NEL TESSUTO O NELLA CELLULA DEL TARGET DETERMINAZIONE DI RFLP PATOGENI Anemia falciforme RFLP (Restriction fragment lenght polymorfism) polimorfismo dovuto a differenze di dimensione di frammenti di restrizione allelici causati dalla presenza o assenza di un sito di restrizione Applicazione del DNA ricombinante: Diagnosi dell’anemia falciforme tramite Southern Blot La mutazione abolisce il sito MstII Ibridazioni in situ Il sequenziamento del DNA (Sanger) Determinazione della sequenza nucleotidica del DNA il metodo di Sanger 2’-3’ DIDEOSSINUCLEOTIDI DNA stampo DNA polimerasi dNTP primer marcato Il contenuto di ciascuna provetta viene caricato in 4 pozzetti separati di un gel di poliacrilammide * Indica il primer (lungo 15 nucleotidi) ** I dNTP sono ad una concentrazione 100 volte superiore di quella dei ddNTP in ciascuna provetta Sequenziamento del DNA: il metodo di Sanger pozzetti autoradiogramma Si legge la sequenza mano mano sintetizzata dal basso verso l’ alto. Il nucleotide G è il più vicino al primer (* indica il primer (lungo 15 nucleotidi)) all’estremità 5’ mentre il nucleotide T è all’estremità 3’. 5’GCTCCACGTAACGT3’ Questa sequenza è complementare al filamento stampo. ddNTP marcati con fluorofori cromatogramma Mappe fisiche di molecole di DNA basate su taglio di enzimi di restrizione (Mappe di Restrizione) Un Sito di restrizione o sequenza consenso viene definito come una particolare sequenza di DNA riconosciuta da un enzima di restrizione o endonucleasi come il punto in cui tagliare la molecola di DNA. Questi siti sono generalmente palindromici e con una sequenza ben precisa. Gli enzimi di restrizione sono degli enzimi che i batteri utilizzano per difendersi dagli attacchi di un DNA estraneo Frammenti di restrizione La mobilita’ di ciascun fammento e’ inversamente proporzionale al logaritmo della sua lunghezza Mappe di restrizione Rappresentano delle mappe fisiche del DNA in cui i siti di restrizione fungono da marcatori. Forniscono le reali distanze in coppie di basi tra 2 geni elettroforesi A COSA SERVE L’ANIMALE TRANSGENICO? • RIPRODUZIONE DI MODELLI DI MALATTIE UMANE IN TOPI • ANALISI IN VIVO DELLA FUNZIONE O MANCANZA DI UN GENE (analisi di ridondanza, fenotipica, sviluppo etc.) • DETERMINAZIONE DI APPROCCI FARMACOLOGICI IDONEI SPERIMENTABILI SU ANIAMALI CHE RIPRODUCONO LA PATOLOGIA UMANA • ANIMALE TRANSGENICO COME “BIOREATTORE” O PRODUTTORE DI PROTEINE UMANE DA POTER PURIFICARE E USARE COME FARMACO Animali transgenici fondatori fondatori progenie MICROINIEZIONE NEI PRONUCLEI ANIMALI CHIMERICI e le CELLULE STAMINALI EMBRIONALI blastocisti Cellule embrionali PROPRIETA’ DELLE CELLULE STAMINALI EMBRIONALI (ES) • Possono dividersi illimitatamente in modo simmetrico senza differenziare • Mantengono un normale corredo cromosomale • Producono cellule di tipo ecto-meso ed endodermico • Si integrano in tutti i tessuti fetali durante lo sviluppo • Colonizzano la linea germinale per originale le cellule delle gonadi • Sono clonogeniche, ovvero da una singola cellula si origina una colonia di cellule geneticamente identiche • Esprimono il fattore di trascrizione Oct-4, che attiva una serie di geni che mantengono la cellula in uno stato proliferativo e indifferenziato • Posono essere indotte a proliferare o differenziare • Spendono la maggior parte del loro ciclo cellulare nella fase S e non necessitano di stimoli esterni per proliferare • Non mostrano inattivazione dell’X Vantaggio nell’uso delle ES - Possono essere sottoposte a diverse manipolazioni genetiche prima di reiniettarle nella blastocisti ricevente - Il transgene puo’ essere coespresso insieme ad un gene marcatore (neo) che consente di selezionare solo le cellule contenenti il transgene (importante soprattutto nei casi di mutagenesi mirata in cui deve avvenire ricombinazione omologa) a b Progenie con cellule germinali portatrici del transgene Eredita’ mendeliana del transgene Cellule germinali aploidi 50% + 50% - Come introdurre il transgene nella cellula zigote o nelle cellule staminali embrionali? •INIEZIONE NEL PRONUCLEO DI PLASMIDI INEFFICIENTE E COSTOSO • ONCORETROVETTORI MoMLV SILENZIAMENTO DEL VETTORE DURANTE LO SVILUPPO • LENTIVETTORI NON SOGGETTI A SILENZIAMENTO
Scaricare