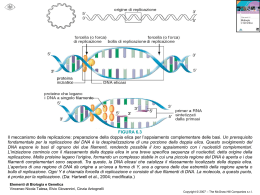
Nucleotidi e acidi nucleici I nucleotidi rappresentano la forma di energia corrente che viene utilizzata nelle attività metaboliche. I nucleotidi sono segnali chimici. I nucleotidi sono i componenti strutturali di un certo numero di cofattori enzimatici. I nucleotidi sono i costituenti degli acidi nucleici. Struttura dei nucleotidi Purina o Pirimidina Fosfato Pentosio Nei deossiribonucleotidi il gruppo ossidrilico -OH nella posizione 2’ (-OH) viene sostituito da un atomo di idrogeno. L’atomo di carbonio anomerico è legato all’atomo di azoto di un’amina mediante un legame N-glicosidico. Legame N-glicosidico I legami N-glicosidici hanno una configurazione β in quasi tutte le biomolecole naturali Pirimidina Purina Adenina Guanina Purine Citosina Timina (DNA) Pirimidine Uracile (RNA) Conformazioni del ribosio Aldeide β-Furanosio L’anello ribofuranosico può esistere in quattro diverse conformazioni Quattro dei cinque atomi sono coplanari; il quinto (il C-2’ o il C-3’) può essere sullo stesso lato del piano in cui si trova l’atomo C-5’ (conformazione endo); oppure sul lato opposto (conformazione eso). Deossiribonucleotidi Deossiadenilato (deossiadenosina 5’-monofosfato) Deossiguanilato (deossiguanosina 5’-monofosfato) Deossitimidilato (deossitimidina 5’-monofosfato) Deossicitidilato (deossicitidina 5’-monofosfato) Deossiguanosina Deossitimidina (o timidina) Deossicitidina Simboli: Deossiadenosina Ribonucleotidi Adenilato (adenosina 5’-monofosfato) Guanilato (guanosina 5’-monofosfato) Uridilato (uridina 5’-monofosfato) Deossicitidilato (citidina 5’-monofosfato) Simboli: Adenosina Guanosina Uridina Citidina Il DNA e l’RNA possono contenere piccole quantità di altre basi meno comuni. Basi minori del DNA deossiribosio deossiribosio 5-metilcitidina N6-Metiladenosina deossiribosio N2-Metilguanosina deossiribosio 5-Idrossimetilcitidina Basi minori del’RNA Ribosio Ribosio Inosina Pseudouridina Ribosio 7-Metilguanosina Ribosio 4-Tiouridina Le cellule contengono anche nucleotidi con gruppi fosforici in posizioni diverse dalla 5’. Adenina Adenina Adenosina 5’-monofosfato Adenina Adenosina 3’-monofosfato Adenosina 2’-monofosfato Adenina Adenosina 2’, 3’-monofosfato ciclico Negli acidi nucleici i nucleotidi sono uniti da legami fosfodiesterei Ponte fosfodiestere L’estremità 5’ manca di un nucleotide nella posizione 5’. I nucleotidi del DNA e dell’RNA sono uniti tra loro in successione mediante “ponti” covalenti tra gruppi fosforici, in cui il gruppo ossidrilico 5’ di un’unità necleotidica è unito al gruppo ossidrilico 3’ di quella successiva, formando un legame fosfodiestere. Lo scheletro covalente degli acidi nucleici è costituito da un’alternanza di gruppi fosforici e di residui di pentosio, mentre le basi azotate possono essere considerate come gruppi laterali uniti allo scheletro. L’estremità 3’ manca di un nucleotide nella posizione 3’. Lo scheletro covalente del DNA e dell’RNA è idrofilico. Lo scheletro covalente del’RNA, ma non quello del DNA, viene idrolizzato rapidamente in ambiente alcalino. Il gruppo ossidrilico in posizione 2’ partecipa al processo idrolitico. Derivato 2’, 3’-monofosfato ciclico Miscela di derivati 2’- e 3’-monofosfato L’assenza del gruppo ossidrilico sull’atomo di carbonio 2’ nel DNA aumenta la sua resistenza all’idrolisi. RNA accorciato Rappresentazione schematica di una sequenza nucleotidica Le basi azotate Le purine e le pirimidine sono composti moderatamente basici e per questo motivo vengono chiamate basi. Le risonanze che coinvolgono numerosi atomi degli anelli delle basi azotate conferiscono alla maggior parte dei legami un carattere di parziale doppio legame. Le pirimidine sono pertanto molecole planari e le purine presentano solo un leggero ripiegamento. Le basi puriniche e pirimidiniche libere possono esistere in due o più forme tautomeriche a seconda del pH della soluzione. Lattame Lattime Doppio lattime Forme tautomeriche dll’uracile; la forma predominante a pH 7 è la forma lattamica. Strutture delle basi azotate costituenti degli acidi nucleici nelle loro forme tautomeriche predominanti a pH 7: Adenina Guanina Purine Citosina Timina (DNA) Pirimidine Uracile (RNA) Sempre per effetto della risonanza, tutte le basi assorbono la luce UV, e gli acidi nucleici sono caratterizzati proprio da un elevato assorbimento della luce a una lunghezza d’onda intorno a 260 nm. Le basi azotate (2) Le purine e le pirimidine sono molecole idrofobiche e relativamente poco solubili in acqua ad un pH neutro. Le interazioni di impilamento idrofobico, in cui i piani degli anelli di due o più basi si vengono a trovare in posizioni parallele, l’una sopra all’altra, rappresentano uno dei due principali sistemi di interazione tra le basi. I gruppi funzionali più importanti delle basi puriniche sono gli atomi di azoto degli anelli, i gruppi carbonilici e i gruppi amminici esociclici. I legami idrogeno nelle coppie di basi definite da Watson e Crick. Timina Adenina Citosina Guanina Il DNA conserva l’informazione genetica L’esperimento di Griffith Batterio utilizzato: Ceppo S (capsulato, virulento) di Streptococcus pneumoniae Batterio utilizzato: Ceppo R (non capsulato, e non virulento) di Streptococcus pneumoniae L’esperimento di Avery-MacLeod-McCarty (1944) Il fattore trasformante di Griffith era DNA Il trattamento del DNA con enzimi proteolitici non distruggeva la sua capacità trasformante, il trattamento con deossiribonucleasi sì. L’esperimento di Hershey-Chase (1952) L’esperimento di Hershey-Chase (1952) La conferma dei risultati venne quando altri campioni di batteri e di particelle fagiche marcate furono incubati insieme per periodi di tempo più lunghi, in modo da ottenere una generazione di particelle virali. La progenie di particelle T2 non conteneva 35S, mentre conteneva ancora il 32P. Il DNA, e non le proteine, era trasmesso da una generazione virale all’altra. L’informazione ereditaria dei virus era contenuta nel DNA. “Regole di Chargaff” (Erwin Chargaff, fine degli anni ‘40) 1) La composizione in basi varia da una specie all’altra. 2) Le molecole di DNA isolate da tessuti diversi della stessa specie hanno la stessa composizione in basi. 3) La composizione in basi del DNA di una data specie non si modifica con l’età dell’organismo, con lo stato nutrizionale o in seguito a variazioni dell’ambiente 4) In tutte le molecole di DNA, indipendentemente dalla specie cui appartiene: Rosalind Franklin e Maurice Wilkins analizzarono all’inizio degli anni ‘50 i cristalli di DNA con il metodo della diffrazione dei raggi X. Risultati: I polimeri di DNA erano elicoidali con 2 periodicità lungo il loro asse: una primaria di 3,4 Å e una secondaria di 34 Å. La struttura primaria di un acido nucleico è la sua struttura covalente Qualsiasi struttura stabile e regolare nucleotidi di un acido nucleico può secondaria assunta essere da una parte o da tutti i considerata come struttura Il complesso ripiegamento di un cromosoma all’interno del nucleoide batterico o della cromatina degli eucarioti viene in genere considerato come struttura terziaria Nel 1953 James D. Watson e Francis Crick proposero un modello tridimensionale della struttura del DNA che poteva spiegare tutte le proprietà sino ad allora note. Il DNA è costruito da due catene elicoidali avvolte intorno allo stesso asse per formare una doppia elica destrorsa. Lo scheletro covalente idrofilico, composto da un’alternanza di deossiribosio e gruppi fosforici, è all’esterno della doppia elica, in contatto con l’ambiente circostante. L’anello furanosico di ogni residuo di deossiribosio è nella conformazione C-2’ endo. Le basi puriniche e pirimidiniche sono impilate all’interno della doppia elica con le loro strutture planari ad anello poste in posizione praticamente perpendicolare al lungo asse longitudinale della molecola. La relazione spaziale che si crea tra le catene genera una scanalatura maggiore (o solco maggiore) e una scanalatura minore (o solco minore) sulla superficie della doppia elica. Ogni base nucleotidica di una catena è appaiata sullo stesso piano con una base dell’altra catena (G≡C; A=T). Le due catene dell’elica sono antiparallele. Modello di Watson e Crick della struttura del DNA Solco minore Solco maggiore Il modello originale proponeva che vi fossero 10 coppie di basi oppure 34 Å per ogni giro dell’elica. Misure successive hanno dimostrato che in ogni giro d’elica vi sono 10,5 coppie di basi e 36 Å. Modello a bastoncini e modello spaziale. La doppia elica del DNA viene tenuta insieme da due gruppi di forze: I legami idrogeno tra coppie di basi complementari Le interazioni tra le basi impilate. A causa del fatto che i legami glicosidici non sono diametralmente opposti l’uno all’altro, ciascuna coppia di basi ha un lato più esteso che delimita la scanalatura maggiore e un lato meno esteso che delimita la scanalatura minore I solchi maggiore e minore sono caratterizzate da potenziali donatori (in blu) e accettori (in rosso) di legami idrogeno. Le molecole degli acidi nucleici a doppia elica contengono due infossature, o scanalature, dette rispettivamente scanalatura (o solco) maggiore e scanalatura (o solco) minore. Il solco maggiore presenta più caratteristiche che differenziano una coppia di basi dall’altra rispetto al solco minore. Le dimensioni più ampie della scanalatura maggiore nel DNA-B rendono questa struttura maggiormente accessibile alle interazioni con proteine che riconoscono sequenze specifiche del DNA Vista assiale del DNA Nella doppia elica le basi sono impilate una sull’altra Replicazione del DNA suggerita da Watson e Crick Catena nuova Catena nuova Catena preesistente (progenitrice) Catena figlie Catena preesistente (progenitrice) Sono state descritte numerose deviazioni dalla struttura del DNA descritta da Watson e Crick (forma B del DNA). Le variazioni strutturali dipendono da tre fattori: 1) Una conformazione diversa del deossiribosio. 2) Rotazioni intorno ai legami contigui che fanno parte dello scheletro i fosfodeossiribosio. 3) Rotazione libera intorno al legame N-glicosidico sul C-1’. sin-Adenosina anti-Adenosina anti-Citidina A causa di costrizioni steriche, i nucleotidi purinici e pirimidinici possono assumere solo 2 conformazioni stabili rispetto al deossiribosio, chiamate sin e anti. Le pirimidine sono in genere nella conformazione anti a causa di interferenze steriche tra lo zucchero e l’ossigeno carbonilico sul C-2. La struttura proposta da Watson e Crick (forma B del DNA) è quella più stabile per una molecola di DNA con sequenza casuale e in condizioni fisiologiche. Confronto tra le tre varianti strutturali del DNA La forma A del DNA (a) La forma A è favorita in condizioni povere di acqua. •Il DNA è sempre organizzato in una doppia elica destrorsa. • L’elica è un po’ più larga e più corta dell’elica B. •Le coppie di basi sono maggiormente ruotate rispetto all’asse dell’elica in confronto a quelle della forma B, che sono quasi perpendicolari. •Il numero di coppie di basi per giro d’elica è 11 invece di 10,5. La forma A del DNA (b) Molte delle differenze strutturali esistenti tra il DNA-B e il DNA-A derivano da una diversa compattazione delle loro unità di ribosio. Nel DNA-A l’atomo di carbonio in posizione 3’ si trova fuori dal piano della molecola formato dagli altri quattro atomi dell’anello furanosico (conformazione endo-C-3’). Nel DNA-B è invece il carbonio in posizione 2’ a trovarsi fuori dal piano (conformazione endo-C-2’). La maggiore vicinanza delle strutture endo-C-3’ nel DNA-A determina una inclinazione di 19o delle coppie di basi. I gruppi fosforici e altri gruppi dell’elica di tipo A legano meno molecole di acqua rispetto a quelli del DNA-B. La disidratazione quindi favorisce la forma A. La modificazione strutturale presente nel DNA A rende più profondo il solco maggiore e meno profondo il solco minore. L’elica di tipo A non è presente solo nel DNA disidratato. Le regioni a doppia catena di RNA e alcuni ibridi RNA-DNA adottano una forma a doppia elica simila a quella del DNA-A. La posizione del gruppo ossidrilico lagato all’atomo di carbonio 2’ del ribosio impedisce all’RNA di formare un’elica classica B del tipo di Watson e Crick a causa dell’ingombro sterico. L’atomo di ossigeno in posizione 2’ si avvicinerebbe troppo ai tre atomi del gruppo fosforico adiacente e a uno degli atomi della base successiva. In un’elica di tipo A l’atomo di ossigeno in posizione 2’ è rivolto verso l’esterno, lontano dagli altri atomi. Il DNA-Z è una doppia elica sinistrorsa nella quale lo scheletro zucchero-fosfato è disposto a zigzag La forma Z del DNA è radicalmente diversa dalla forma B La differenza più evidente è la rotazione dell’elica in senso sinistrorso. Vi sono 12 coppie di basi per giro d’elica e la struttura appare più sottile ed allungata. La forma Z del DNA Lo scheletro covalente azzume un andamento a zig -zag. Alcune sequenze nucleotidiche si ripiegano in eliche Z sinistrorse molto più facilmente di altre (es.: purine alternate a pirimidine). Sono necessarie concentrazioni elevate di sali per ridurre al minimo le repulsioni elettrostatiche tra i gruppi fosforici dello scheletro, che nella ztruttura Z sono più vicini gli uni agli altri. I residui di purina devono “saltare” alla conformazione sin, alternandosi a residui di pirimidina nella conformazione anti. Il solco maggiore è appena accennato e il solco minore è stretto e profondo. Confronto tra le forme A, B e Z del DNA Forma A Forma B Forma Z Destrorsa Destrorsa Sinistrorsa ≈ 26 Å ≈ 20 Å ≈ 18 Å Coppie di basi per ogni giro d’elica 11 10,5 12 Distanza tra base e base 2,6 Å 3,4 Å 3,7 Å Piegamento delle basi normale all’asse dell’elica 200 60 70 Conformazione dello zucchero C-3’-endo C-2’-endo Anti Anti Senso dell’elica Diametro Conformazione del legame glicosidico C-2’-endo per le pirimidine; C-3’-endo per le purine Anti per le pirimidine; sin per le purine Alcune sequenze di DNA adottano strutture insolite. Un palindromo è una parola che risulta la stessa letta in una direzione o in quella inversa (es.: OSSESSO). Il termine viene applicato alle regioni di DNA di in cui vi sono ripetuti invertiti di una sequenza di basi con una doppia simmetria presente nelle due catene del DNA Quando la sequenza è presente in ciascuna catena, la sequenza viene detta ripetuto speculare. Ripetuto speculare Un palindromo può formare strutture a forcina e a forma di croce. Struttura a forcina (hairpin) Struttura a croce I nucleotidi che partecipano all’appaiamento tipo Watson e Crick possono formare altri legami idrogeno con gruppi disposti nel solco maggiore. Un residuo di citidina (se protonato) può appaiarsi a una guanosina di una coppia C≡G, e la timina può appaiarsi a un’adenosina di una coppia A=T Appaiamento tipo Hoogsteen L’appaiamento di Hoogsteen consente di formare DNA triplex. Le strutture triplex si possono formare in lunghe sequenze contenenti in una data catena solo pirimidine o solo purine. Sequenze ricche in residui guanosinici possono formare tetraplex o o DNA H Tripla elica Un’organizzazione insolita del DNA, nota con il nome di DNA H, si può formare in tratti di polipirimidine/polipurine che contengono anche all’interno della sequenza ripetuti speculari. Questa struttura produce un brusco ripiegamento del DNA. mRNA dei procarioti mRNA monocistronico mRNA policistronico Molti RNA hanno strutture tridimensionali complesse Per l’RNA non esiste una semplice struttura secondaria che rappresenti un punto di riferimento per la sua struttura, come accade per la doppia elica del DNA Le interazioni deboli, in particolare le interazioni idrofobiche tra le basi impilate, sono determinanti per la stabilità della struttura. Quando sono presenti sequenze complementari, la struttura predominante è una doppia elica destrorsa nella forma A. Struttura elicoidale destrorsa di una catena singola di RNA dettata dalle interazioni create dall’impilamento delle basi Possibile struttura secondaria dell’RNA M1, un componente dell’enzima Rnasi P1 di E. coli. L’RNA si può appaiare a catene complementari di DNA o di RNA. Le regole sono le stesse del DNA: La G si appaia con la C La A con la U (o con la T, occasionalmente presente nell’RNA). Un’eccezione è l’appaiamento della G con la U, facilmente riscontrabile tra due catene di RNA ma assente nel DNA. Le catene appaiate di RNA o di RNADNA duplex sono antiparallele, come nel DNA. Forcina Catena singola Ansa interna Rigonfiamento Struttura secondaria degli RNA Doppia elica nella struttura a forcina Nelle eliche regolari destrorse di tipo A sono comuni interruzioni della struttura a causa di errori di appaiamento delle basi che portano alla formazione di protuberanze o ad organizzazioni di anse (loop) interni alla molecola. Alcune brevi sequenze di basi (es.: UUCG) sono spesso presenti alle estremità delle strutture a forcina e sono in grado di formare anse particolarmente stabili. Struttura tridimensionale del tRNA della fenilalanina del lievito Contributi strutturali aggiuntivi alla stabilizzazione della struttura secondaria di alcuni RNA possono arrivare da legami idrogeno che non fanno parte dell’appaiamento standard di Watson e Crick. Il gruppo ossidrilico 2’ del ribosio può formare un legame idrogeno con altri gruppi. Ribozima a testa di martello (virus delle piante) Struttura di un introne del protozoo ciliato Tetrahymena thermophila, in grado di catalizzare la propria escissione (ribozima), separandosi dagli esoni. Il DNA e l’RNA a doppia elica possono essere denaturati La denaturazione degli acidi nucleici Il calore e pH estremi, così come determinano la denaturazione delle proteine globulari, causano anche la denaturazione (o fusione) del DNA a doppia elica. Il processo avviene mediante la rottura dei legami idrogeno tra le basi appaiate e delle interazioni idrofobiche tra le basi impilate. La doppia elica si apre liberando le due catene singole. I legami covalenti del DNA non vengono rotti. Quando la temperatura o il pH vengono riportati a valori vicini a quelli fisiologici, i segmenti srotolati delle due catene si riavvolgono (riassociazione o annealing) Il DNA e l’RNA a doppia elica possono essere denaturati DNA a doppia elica Denaturazione Rinaturazione (annealing) DNA parzialmente denaturato Separazione delle catene Associazione delle catene mediante appaiamento delle basi Catene separate di DNA Con una struttura ad avvolgimento casuale (random coiled) Denaturazione al calore del DNA Ogni specie di DNA ha una temperatura di denaturazione caratteristica, detta anche punto di fusione (tm). Relazione tra il tm e il contenuto di GC di un DNA Il punto di fusione è tanto più alto quanto più alto è il contenuto in coppie di basi GC. Effetto ipercromico L’interazione tra le basi impilate ha l’effetto di ridurre la quantità di luce UV assorbita e, pertanto, un filamento singolo di DNA assorbe la luce in maniera più efficace rispetto ad una molecola di DNA a doppio filamento. L’assorbimento della luce a 260 nm di una soluzione di DNA aumenta quando la doppia elica fonde e le catene si separano. La transizione da DNA a doppia elica a DNA denaturato a catena singola può essere monitorata misurando l’assorbimento della luce UV. E’ possibile visualizzare il DNA parzialmente denaturato Possono essere denaturati anche gli acidi nucleici a doppia catena composti da due catene di RNA o da una catena di RNA e una di DNA. I duplex di RNA sono molto più stabili di quelli di DNA. A pH neutro, un RNA a doppia elica spesso si denatura a una temperatura almeno 20 oC più alta di quella richiesta da una molecola di DNA con una sequenza comparabile. La stabilità dell’ibrido RNA-DNA è in genere intermedia tra quella dell’RNA e quella del DNA. La capacità di appaiamento di due catene di DNA (o di RNA o di DNA-RNA) complementari può essere utilizzata per identificare sequenze di DNA simili in specie diverse oppure nel genoma della stessa specie. Gli acidi nucleici di specie diverse possono formare ibridi I nucleotidi e gli acidi nucleici vanno incontro a trasformazioni non enzimatiche Reazioni di deamminazione Depurinazione Timine adiacenti Dimero di timina ciclobutanico Fotoprodotto 6-4 Piega Sodio nitrito Sodio nitrato Nitrosammina Precursori dell’acido nitroso che promuove reazioni di deamminazione Agenti alchilanti metionina adenosina S-Adenosilmetionina Dimetilnitrosammina Dimetilsolfato Mostarda azotata Tautomeri della guanina O6-Metilguanina E’ possibile determinare la sequenza di lunghi tratti di DNA Catena primer Catena stampo Analogo 2’, 3’ - dideossi Stampo della sequenza da identificare DNA polimerasi, 4 dNTP 4 ddNTP Denaturazione Segmenti di DNA marcati con un colore, copiati dallo stampo con la sequenza da identificare Segmenti di DNA marcati con un colore, copiati dallo stampo con la sequenza da identificare Migrazione del DNA I segmenti marcati con il colore sono caricati su un capillare contenente un gel di supporto e sottoposti ad elettroforesi. Raggio laser Rivelatore Laser I risultati che si ottengono dal computer dopo che le bande colorate sono passate davanti al rivelatore Genoma di Hemophilus influenzae, il primo genoma di un organismo vivente che sia stato sequenziato Per vedere questa immagine occorre QuickTime™ e un decompressore Animation. E’ possibile sintetizzare oligonucleotidi I nucleotidi trasportano energia chimica Estere Adenina Anidride Anidride Anidride acetica, un’anidride acida carbossilica Metilacetato, un estere acido carbossilico I nucleotidi adeninici fanno marte di molti cofattori enzimatici Coenzima A Nicotinamide adenin dinucleotide (NAD+) a Alcuni nucleotidi sono molecole regolatrici Adenina Guanina Adenosina 3’, 5’ -monofosfato ciclico (AMP ciclico, cAMP) Guanina Guanosina 3’-difosfato, 5’ -difosfato (Guanosina tetrafosfato, ppGpp) Guanosina 3’, 5’ -monofosfato ciclico (GMP ciclico, cGMP) FINE
Scarica