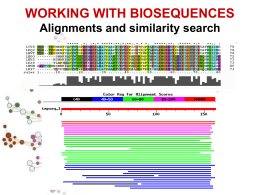
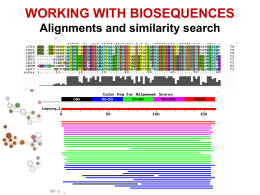
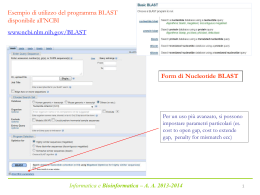
Ricerca di similarità di sequenza (FASTA e BLAST) Allineamento di due sequenze Allineamento multiplo di sequenze RICERCA DI SIMILARITA’ E ALLINEAMENTO DI SEQUENZE BLAST e PSI-BLAST http://www.ncbi.nlm.nih.gov/blast/ FASTA http://fasta.bioch.virginia.edu/ oppure http://www.ebi.ac.uk/fasta33/ Alcune caratteristiche dei tools più usati: BLAST (Basic Local Alignment Search Tool), sviluppato dal National Center for Biotechnology Information, NCBI): - allineamento locale - estremamente veloce - parte cercando brevi frammenti della sequenza, che poi prova ad estendere - usa una matrice di sostituzione in entrambe le fasi del processo di allineamento (scansione del database e estensione della subsequenza): più preciso ha quattro opzioni fondamentali: BLASTP: confronta sequenze proteiche contro un database proteico BLASTN: confronta sequenze nuclotidiche contro un database nucleotidico TBLASTN: confronta una sequenza proteica contro un database nucleotidico, traducendo ciascuna sequenza del database nucleotidico nei suoi 6 frames di lettura BLASTX: confronta una sequenza nucleotidica contro un database proteico, dopo averla tradotta nei suoi 6 frames di lettura. BLAST: BLASTP Seconda parte della pagina di BLAST: Numero atteso di HSP (Highscoring Segment Pair) valutato su base statistica Dimensione delle parole Scelta della matrice di sostituzione I valori di default usati da BLAST sono W=3, T=13, Matrice=BLOSUM 62 Penalità assegnata ai gap Terza parte della pagina di BLAST: FASTA: http://www.ebi.ac.uk/fasta33/ Ktup: lunghezza delle parole Align: numero di allineamenti finali Open e residue: Penalità per i gap Vari database Sequenza in formato FASTA Allineamento di due sequenze: BLAST: bl2seq LALIGN: http://www.ch.embnet.org/software/LALIGN_form.html EMBOSS: http://www.ebi.ac.uk/emboss/align/ LALIGN: ALLINEAMENTO MULTIPLO DI SEQUENZE Informazione biologica maggiore rispetto a quella riportata l’allineamento di due sole sequenze: i residui più importanti dal punto di vista strutturale o funzionale saranno estremamente conservati tra tutte le sequenze dell’allineamento. “Una sequenza amminoacidica fa la timida; un paio di sequenze omologhe sussurrano; molte sequenze allineate gridano”. Per essere informativo un allineamento multiplo dovrebbe contenere una distribuzione di sequenze sia strettamente sia lontanamente correlate: Svantaggi: •tutte strettamente correlate => ridondanza •tutte lontanamente correlate => allineamento inaccurato => inutilità ALLINEAMENTO MULTIPLO DI SEQUENZE Programmi per l’allineamento multiplo globale: CLUSTALW: http://www.ebi.ac.uk/clustalw/ o scaricare il programma eseguibile KALIGN Multalin TCOFFEE http://msa.cgb.ki.se/cgi-bin/msa.cgi http://bioinfo.genopole-toulouse.prd.fr/multalin/multalin.html http://www.ch.embnet.org/software/TCoffee.html CLUSTAL W: -il tool più comune utilizzato per l’allineamento multiplo di sequenza: - potenziato per allineamenti di sequenze proteiche divergenti favorisce l’apertura di gaps in regioni in cui è potenzialmente presente un loop piuttosto che una struttura secondaria ordinata (in base a una penalità residuo-specifica e a una penalità ridotta in regioni idrofiliche) favorisce l’apertura di gaps nelle stesse posizioni.
Scaricare