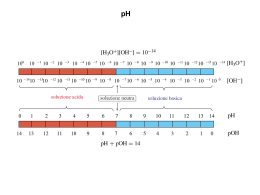
3. Materiali e Metodi 3. MATERIALI E METODI 46 3. Materiali e Metodi 3.1 Campionamento Sono stati campionati complessivamente 29 formaggi, di cui 8 caprini, 12 pecorini e 9 caciotte, di cui 2 da latte bovino e 7 da latte misto (bovino ed ovino). Di questi, le produzioni ottenute con latte crudo locale (19 campioni) sono state prelevate presso caseifici artigianali della regione Marche, mentre i formaggi a latte pastorizzato aggiunto di starter commerciali (10 campioni) sono stati acquistati presso supermercati della provincia di Ancona. I campioni sono stati trasportati in laboratorio in condizioni refrigerate e sottoposti ad analisi microbiologiche, chimico-fisiche e molecolari. Parallelamente al campionamento, per ciascuna produzione oggetto di studio sono state raccolte informazioni relative agli ingredienti utilizzati, la tecnologia di lavorazione e le condizioni di stagionatura. 3.2 Analisi microbiologiche Dieci g di ciascun campione sono stati addizionati di 90 mL di acqua peptonata (0,1 %, p/v) sterile ed omogenati con un omogenizzatore peristaltico (Stomacher 400 Circulator, International PBI), per 2 minuti a 260 rpm, in sacchetti in polietilene ad alta resistenza meccanica, dotati di filtro (SEWARD, UK). Ciascun omogenato (10-1) è stato sottoposto a diluizioni seriali (10-2-10-7); aliquote di 0,1 mL dell’omogenato e di ciascuna diluizione sono state distribuite per spatolamento su mezzi agarizzati selettivi per la conta di (i) lattobacilli (MRS agar Oxoid, addizionato di 170 mg/L di cycloeximide (Hatzikamari et al., 1999) ed incubato a 37°C per 48 h in anaerobiosi); (ii) lattococchi mesofili (M17 agar Oxoid, incubato a 22°C in aerobiosi per 48 h); (iii) cocchi termofili (M17 agar Oxoid incubato a 45°C in aerobiosi per 24 h). Tutti i campioni sono stati testati per Salmonella spp, Listeria monocytogenes ed enterotossina stafilococcica utilizzando l’apparecchio miniVIDAS (Biomerieux ®) con metodo ELFA (Enzyme Linked Fluorescent Assay), come descritto nel protocollo validato ANFOR (Vitek Immuno Diagnostic Assay System; BIO 12/10 09/02; BIO 12/11 03/04). 3.3 Analisi chimico-fisiche Per ciascuna analisi, sono state effettuate tre misurazioni indipendenti. I risultati sono stati espressi come valore medio ± deviazione standard. 47 3. Materiali e Metodi 3.3.1 Determinazione del pH Il pH è stato misurato al cuore del prodotto e nella sottocrosta, utilizzando un pHmetro dotato di un elettrodo da solidi (HI2031, HANNA INSTRUMENTS). 3.3.2 Attività dell’acqua (aw) L’attività dell’acqua (aw) è stata misurata con un indicatore idrometrico elettrico (AW Win, PBI INTERNATIONAL). 3.3.3 Determinazione del contenuto in cloruro di sodio Il contenuto in cloruro di sodio dei campioni è stato determinato utilizzando il metodo VOLHARD (Haouet et al., 2006), basato sulla precipitazione dei cloruri, in seguito all’aggiunta di nitrato d’argento in eccesso, come argento cloruro, che può essere titolato con una soluzione di ammonio solfocianuro, fino alla comparsa di una colorazione rosa persistente. La quantità di cloruro di sodio è calcolata in relazione al volume di titolante utilizzato, secondo la seguente procedura: si pongono 1,5 g di campione in un becher da 50-100 mL; si addizionano 100 mL di acqua distillata, 10 mL di acido nitrico e 25 mL di nitrato d’argento 0,1 N; si porta la soluzione ad una leggera ebollizione per circa 10 min, fino a precipitazione dell’argento cloruro, favorita dalla agitazione frequente; si chiude il becher con carta stagnola e si lascia raffreddare al buio; si aggiungono 5 mL di soluzione satura di allume ferrico e si lavano le pareti del becher con alcuni mL di acqua distillata; si titola con ammonio solfocianuro 0,1 N fino alla comparsa di un colore rosa persistente. In relazione al volume di ammonio solfocianuro utilizzato, viene calcolato il contenuto in cloruro di sodio, espresso in g/100 g di campione, utilizzando la seguente formula: [(V1 x M1) - (V2 x M2)] x 58.45 x 100 P x 1000 V1 = volume in mL di nitrato d’argento M1 = molarità del nitrato d’argento V2 = volume in mL di ammonio solfocianuro M2 = molarità dell’ammonio solfocianuro P = peso in grammi del campione 58.45 = PM del cloruro di sodio 48 3. Materiali e Metodi 3.3.4 Determinazione del contenuto in azoto e proteine Il contenuto in azoto totale (azoto organico più azoto ammoniacale) dei campioni è stato determinato secondo il metodo Kjeldahl (Suppl. ordinario G.U. 2-10-86, Serie generale n. 229, 13). Più in dettaglio, la procedura prevede una fase iniziale di mineralizzazione dell’azoto totale presente nel campione, che porta alla formazione di ammoniaca, successivamente distillata e titolata con acido cloridrico. In relazione al volume di acido cloridrico utilizzato, viene calcolato il contenuto in azoto. Tale valore viene poi moltiplicato per una costante specifica per il latte ed i prodotti lattiero-caseari, al fine ottenere il contenuto in proteine dell’alimento. Questa procedura prevede l’utilizzo di un’unità di mineralizzazione ed un analizzatore automatico Kjeldahl, utilizzato nelle fasi di distillazione e titolazione. Fase di mineralizzazione: si pesa 1 g di campione in un contenitore di vetro compatibile con l’unità di mineralizzazione e con l’analizzatore automatico Kjeldahl; si aggiungono due pasticche di catalizzatore (pasticche Kjeltab) e 15 mL di acido solforico concentrato, agitando delicatamente; si accende l’abbattitore di fumi acidi e si pone l’apposito contenitore in vetro nell’unità di mineralizzazione che provvede al riscaldamento del campione, prima moderatamente fino a scomparsa della schiuma, quindi più intensamente fino ad ebollizione regolare della soluzione; si mantiene in ebollizione fino a quando la soluzione non diventa limpida e di colore verde pallido; si spegne l’unità di mineralizzazione e si lascia raffreddare il campione per almeno mezz’ora, mantenendo l’abbattitore dei fumi acceso; si toglie il campione dall’unità di mineralizzazione e si lascia ulteriormente raffreddare a temperatura ambiente. Fase di distillazione e titolazione: si imposta, nell’analizzatore automatico Kjeldahl, il programma necessario per la distillazione e la titolazione dell’azoto totale; si esegue la taratura dell’elettrodo usando i tamponi di riferimento a pH 4,0 e 7,0; si eseguono almeno tre prove con acqua distillata, di cui la prima è di lavaggio, le altre sono bianchi (prove in bianco): 49 3. Materiali e Metodi - si inserisce un opportuno contenitore contenente acqua distillata nel condensatore dell’analizzatore automatico Kjeldahl: lo strumento erogherà in automatico circa 90 mL di NaOH al 40 % (p/v); - nel recipiente di raccolta defluiranno 60 mL di acido borico al 4% (v/v) a pH 4,65 che rappresenta il punto finale della reazione; conclusa la distillazione (circa 5 min) inizierà la titolazione con acido cloridrico 0,2 N; si pone, quindi, il campione nell’analizzatore automatico Kjeldahl e si ripetono le operazioni descritte al punto precedente (prove in bianco), rilevando il volume in mL di acido cloridrico 0,2 N usato per la titolazione. Si sottrae dal volume di titolazione del campione il volume medio ottenuto dalle prove in bianco (senza considerare il valore relativo al lavaggio con acqua distillata). [(V x B) x N x 14 x 100] P x 100 V = volume di acido cloridrico usato per la titolazione B = media di volumi di acido cloridrico usati per la titolazione dei bianchi N = normalità dell’acido cloridrico 14 = PM dell’azoto P = peso del campione usato Il contenuto in proteine espresso in g/100 g di campione è dato dalla seguente formula: Azoto (g / 100 g) x C C = fattore di conversione dell’azoto in proteine (specifico per ogni alimento): 6.38 latte e prodotti lattiero caseari 3.3.5 Determinazione del numero di perossidi della sostanza grassa Il numero di perossidi della sostanza (espresso in milliequivalenti di ossigeno attivo per Kg di formaggio) è stato valutato utilizzando il metodo proposto da Martillotti e collaboratori (1987), e di seguito brevemente illustrato. Estrazione delle sostanze grasse si pesano almeno 50 g di campione e si introducono in una beuta a collo smerigliato da 300-400 mL; si aggiunge una soluzione di cloroformio-metanolo (2:1) in un volume pari al doppio del peso del campione, agitando delicatamente; si lascia in estrazione per almeno 2-3 h a riposo, oppure per circa 30 min in agitazione continua; si filtra su filtro a pieghe in un imbuto separatore da 300-500 mL; si aggiungono 80 mL di cloruro di sodio al 2 % (p/v); 50 3. Materiali e Metodi dopo alcuni secondi di agitazione, si lasciano riposare le fasi; si raccoglie il cloroformio in un pallone da 250-300 mL; si porta a secco con un evaporatore rotante, applicando una temperatura massima di circa 40°C ± 5°C e una bassa velocità di rotazione; si lascia il pallone a temperatura ambiente per circa 30 min. Il numero di perossidi è determinato utilizzando il seguente protocollo: si pesa 1 g di materia grassa, in una beuta da 250-400 mL; si aggiungono 25 mL della miscela acido acetico glaciale-cloroformio e si agita fino a completa dissoluzione; si aggiungono 0,5 mL di una soluzione satura di ioduro di potassio avendo cura di chiudere subito la beuta. si agita con un movimento rotatorio per circa 1 min; si lascia riposare a temperatura ambiente al buio per 5 min esatti; si diluisce la soluzione con 75 mL di acqua distillata e si aggiungono 0,5 mL di salda d’amido come indicatore (la soluzione acquista un colore grigioviola di intensità variabile in funzione del numero di perossidi); si titola con una soluzione di tiosolfato sodico 0,01 N fino a un viraggio della soluzione ad un colore bianco latte; si effettua in parallelo una prova in bianco, effettuando una titolazione con reagenti, senza campione. Il numero di perossidi, espresso in milliequivalenti di ossigeno attivo per Kg di sostanza grassa, è calcolato utilizzando l’espressione: (V x N x 1000) m V = volume di tiosolfato di sodio N = normalità del tiosolfato di sodio m = quantità di sostanza grassa in grammi 3.3.6 Determinazione del contenuto di lipidi Il contenuto lipidico dei campioni è stato determinato attraverso l’estrazione delle sostanze grasse con etere di petrolio, previa idrolisi acida (G.U. n. 88 del 2 ottobre 1986). Dopo evaporazione del solvente, il tenore lipidico è stato determinato per via gravimetrica, come di seguito brevemente descritto: si pone un pallone da 100-250 mL a condizionare a 105°C ± 5°C in stufa, per almeno 1 h; 51 3. Materiali e Metodi si lascia raffreddare in essiccatore per almeno 30 min, e si pesa il pallone su bilancia analitica; 3 g di campione sono addizionati di 8-10 mL di una soluzione di acido cloridrico al 25% (v/v) in un cilindro da 100 mL dotato di tappo; si pone il cilindro in acqua bollente, agitando leggermente, per circa 30 min; si raffredda a temperatura ambiente; si aggiungono 10 mL di alcool etilico e si mescola; si aggiungono 25 mL di etere etilico, si chiude il cilindro e si agita vigorosamente per circa 1 min; si apre il cilindro delicatamente e si aggiungono 25 mL di etere di petrolio; si chiude ancora il cilindro, e lo si agita per inversione, per circa 30 sec; si lascia riposare fino a separazione completa delle fasi; si raccoglie accuratamente, con una pipetta, la fase limpida superiore, che viene posta nel pallone precedentemente condizionato e pesato; si procede ad ulteriori due passaggi di estrazione con 15 mL di etere etilico e 15 mL di etere di petrolio (prelevando sempre la fase limpida superiore); si porta a secco in un evaporatore rotante ad una temperatura non superiore a 40-45°C; si pone il pallone in stufa a 105°C ± 5°C per almeno 2 h; si lascia raffreddare in essiccatore, per circa 30 min e si pesa il campione utilizzando una bilancia analitica. Il contenuto in lipidi (espresso in percentuale/100 g di campione) è calcolato sulla base della seguente formula: (C2 – C1) x 100 M C2 = peso del pallone contenente grasso C1 = peso del pallone vuoto dopo condizionamento M = massa in grammi del campione 3.4 Analisi PCR-DGGE (Denaturing Gradient Gel Electrophoresis) 3.4.1 Estrazione di DNA batterico da formaggio L’estrazione di DNA batterico da formaggio è stata eseguita seguendo la procedura proposta da Rantsiou e collaboratori (2008) ed in parte modificata come di seguito descritto: 52 3. Materiali e Metodi si aggiungono 2 g di campione finemente tritato a 20 mL di una soluzione di sodio citrato (2% trisodio citrato di-idrato) e si incuba a 45°C per 30 min; si vortexa per 5 min e si centrifuga a 8.000 rpm per 10 min; si rimuove il sovranatante e si risospende il pellet in 300 µL di tampone TE (10mM TrisHCl; 1mM EDTA, pH 8); si centrifuga alla massima velocità per 10 min e si elimina il sovranatante; si risospende il pellet in 120 µL di proteinasi K buffer ( 50mM Tris-HCl; 10mM EDTA; 0.5% SDS, pH 7,5); si trasferisce la sospensione in un nuovo tubo contenente 0,3 g di sfere di vetro con diametro di 0,5 mm; si aggiungono 20 µL di una soluzione di proteinasi K (25 mg/mL) e lisozima (140 mg/mL) e si incuba a 50°C per 1 h; si aggiungono 150 µL di 2X breaking buffer (4% Triton X-100; 2% SDS; 200mM NaCl; 20mM Tris; 2mM EDTA, pH 8); si aggiungono 300 µL di fenolo/cloroformio/alcol isoamilico 25:24:1; si vortexa 3 volte per 30 sec intervallati ogni volta da 10 sec; si centrifuga a 4°C a 13.000 rpm per 10 min; si trasferisce la fase acquosa in un nuovo tubo; si aggiunge 1 mL di etanolo assoluto e si mescola delicatamente; si centrifuga a 4°C a 14.000 rpm per 10 min; si lascia asciugare il pellet all’aria; si dissolve il DNA in 50 µL di acqua sterile; si aggiunge 1 µL di RNasi DNasi-free (10 mg/mL) e si incuba a 37°C per 1 h. 3.4.2 Preparazione dei bulk di colonie di batteri lattici Le piastre di MRS e M17, utilizzate per la conta di lattobacilli, lattococchi mesofili e cocchi termofili (paragrafo 3.2), sono state utilizzate per la preparazione dei bulk di colonie come suggerito da Ercolini e collaboratori (2001). La patina batterica sviluppatasi sulle piastre corrispondenti a due diluizioni seriali (2 ed una piastra contabile, ovvero con un numero di colonie compreso fra 30 e 300) è stata risospesa in 53 3. Materiali e Metodi 1,5 mL di acqua peptonata (0,1 %, p/v) sterile; 300 µL della sospensione cellulare così ottenuta sono stati trasferiti in tubi sterili ed utilizzati per l’estrazione del DNA batterico. 3.4.3 Estrazione di DNA dai bulk di colonie L’estrazione di DNA batterico dai bulk di colonie è stata effettuata secondo la procedura descritta da Hynes e collaboratori (1991) ed in parte modificata come di seguito descritto: si centrifuga a 13.000 rpm per 5 min; si rimuove il sovranatante e si risospende il pellet cellulare in 1 mL di tampone STE (10mM TrisHCl, pH 8; 100 mM NaCl; 1 mM EDTA, pH 8) contenente saccarosio (20%, p/v) e lisozima (25mg/mL); si incuba a 37°C per 3 h; si centrifuga a 13.000 rpm per 3 min; si risospende il pellet in 1 mL di tampone di lisi (50 mM KCl; 10 mM Tris-HCl, pH 8.3; 0,1 mg/mL gelatina; 0,45% Triton X-100; 0,45% Tween 20) contenente proteinasi K (100 µg/mL); si incuba a 60°C per 3 h; si tratta il campione a 95°C per 10 min allo scopo di inattivare la proteinasi K contenuta nel tampone. 3.4.4 Quantizzazione spettrofotometrica Concentrazione e purezza del DNA estratto sono state determinate spettrofotometricamente (Spectrophotometer Cary 1E UV-visible, Varian, Australia, Pty. Ltd), misurando l’assorbanza a 260 nm, 280 nm e 234 nm, come descritto da Sambrook e collaboratori (1989). 3.4.5 Amplificazione della porzione del gene per il 16S rRNA compresa tra le regioni variabili V6 e V8 L’amplificazione delle regioni variabili V6-V8 del gene codificante il 16S rRNA è stata effettuata utilizzando i primer U968-f [5’-AAC GCG AAG AAC CTT AC -3’] e L1401-r [5’-GCG TGT GTA CAA GAC CC-3’] (Randazzo et al., 2002). Il primer U968-f è stato disegnato inserendo all’estremità 5’ la CGclamp descritta da Muyzer e collaboratori (1993). Centoventi ng di DNA templato sono stati amplificati in un volume finale di 25 µL, contenente: tampone di reazione 1X; MgCl2 3 mM; dNTPs 0,2 mM; primer 0,1 µM; Taq polimerasi (EuroTaq, CelBio) 0,625 U, utilizzando le condizioni illustrate in Figura 3.1. Le amplificazioni sono state condotte nel ciclatore termico My cycler Thermal cycler (Biorad, Hercules, Canada). 54 3. Materiali e Metodi 94 °C 94 °C 5‘ 30” 56 °C 72 °C 72 °C 40” 30’ 30 “ 15 °C 35 cicli Figura 3.1. Ciclo termico utilizzato per l’amplificazione via PCR delle regioni variabili V6-V8 del gene codificante il 16S rRNA. 3.4.6 Elettroforesi su gel di agarosio I prodotti di PCR sono stati preliminarmente visualizzati mediante elettroforesi su gel di agarosio 1,5% (p/v), contenente 2,5% (v/v) di etidio bromuro (10 mg/mL), in tampone TBE (Tris Borato-EDTA) 0,5X (Sambrook et al., 1989). La migrazione elettroforetica è stata condotta ad un voltaggio di 4 V/cm per 45 min utilizzando il marcatore molecolare DNA Ladder Mix (Fermentas M-Medical, Milano, Italia). Gli ampliconi sono stati visualizzati agli UV (λ 320 nm) utilizzando l’acquisitore Gel Doc 1000 (BioRad) ed il programma Multi-Analyst (BioRad). 3.4.7 Reconditioning-PCR Al fine di ridurre il numero eventuali molecole eterologhe (Thompson et al., 2002), 5 µL del prodotto di PCR sono stati re-amplificati (reconditioning-PCR) in una miscela di reazione di 50 µL contenente: tampone di reazione 1X; MgCl2 3 mM; dNTPs 0,2 mM; primer 0,1 µM; Taq polimerasi (EuroTaq, CelBio) 0,625 U. La reazione è stata effettuata utilizzando il ciclo termico illustrato in Figura 3.2. 94 °C 94 °C 5‘ 30” 56 °C 72 °C 72 °C 40” 30’ 30 “ 15 °C ∞ 3 cicli Figura 3.2: Reconditioning - PCR 55 3. Materiali e Metodi 3.4.8 Elettroforesi su gel di poliacrilamide in gradiente denaturante Gli ampliconi sono stati separati come descritto da Cocolin e collaboratori (2001), utilizzando l’apparato Dcode Universal Mutation System (Biorad) (Figura 3.3). Aliquote di 20 µL dei prodotti di PCR sono state caricate su gel di poliacrilamide (acrilamide-bisacrilamide 37.5:1) contenente un gradiente denaturante lineare (50-70,6%) [una soluzione 100% denaturante contiene urea 7 M e formamide 40 % (p/v)]. Figura 3.3: Apparato Dcode Universal Mutation System. Preparazione della cella elettroforetica Preliminarmente alla migrazione elettroforetica, vetri, pettini e spacer sono stati lavati con una soluzione detergente di metanolo e KOH 5 % (p/v) (Sambrook et al.,1989), risciacquando accuratamente prima con acqua e poi con etanolo. Preparazione della soluzione denaturante La soluzione denaturante (Vf= 20 mL) è stata preparata aggiungendo: Urea (7 M) 8,4 g Formamide (40%) 8 mL H2O distillata sterile 12 mL Preparazione del gel di poliacrilamide Il gel di poliacrilamide è stato realizzato utilizzando due soluzioni contenenti differenti concentrazioni di agente denaturante. Low solution (50 % agente denaturante, Vf =15 mL): agente denaturante 7,5 mL acrilamide 4 mL APS 10% (Ammonio Persolfato) 0,105 mL TAE 50 X 0,3 mL [trizma base; 1M acido acetico glaciale; 50 mM EDTA; pH 8,0] H2O distillata sterile 3,09 mL TEMED 5,25 µL High solution (70,6 % agente denaturante, Vf =15 mL): agente denaturante 10,6 mL acrilamide 4 mL APS 10% 0,105 mL TAE 50X 0,3 mL TEMED 5,25 µL Le due soluzioni sono state trasferite in due siringhe opportunamente montate su di un generatore di gradiente e versate, per mezzo di tale strumento, nell’intercapedine tra i due vetri (Figura 3.4). 56 3. Materiali e Metodi Dopo aver versato il gel, è stato inserito immediatamente il pettine, evitando quanto possibile la formazione di bolle. Al termine della polimerizzazione (dopo circa 60 min a 25°C), il pettine è stato rimosso ed i residui di acrilamide non polimerizzata nei pozzetti rimossa con un puntale contenente TAE 1X. Figura 3.4. Allestimento del gel di poliacrilamide mediante un generatore di gradiente. 3.4.9 Costruzione del ladder Al fine di identificare preliminarmente le specie microbiche presenti nei campioni di formaggio in esame, tre colture pure di batteri lattici, i cui ampliconi ottenuti con i primer U968-f e L1401r hanno prodotto su gel profili costituiti da una singola banda, sono stati selezionati per la preparazione di un ladder di identificazione, ottenuto miscelando uguali quantità di ampliconi (10 µL). I tre ceppi, Streptococcus thermophilus DSMZ 20617, Lactococcus lactis subsp. lactis DSMZ 20729 e Lactobacillus brevis NCIMB 8664, sono stati ottenuti dalla “Deutsche Sammlung von Mikrorganismen und Zellkulturen” (http://www.dsmz.de/) (Braunsweig, Germany) e dalla “National Collection of Industrial Marine and Food Bacteria” (NCIMB, Aberdeen, Scotland, UK) (http://www.ncimb.com/culture.html). 3.4.10 Estrazione di DNA da banda e purificazione Le bande visualizzate su gel sono state excise con opportuni puntali monouso; il DNA è stato eluito ponendo ciascuna banda excisa in 50 µL di tampone TE ed incubata a 4°C overnight. Sei µL di eluito sono stati sottoposti ad amplificazione utilizzando il primer U968-f privo di GC-clamp, come descritto al paragrafo 3.4.5. Gli ampliconi sono stati, quindi, purificati utilizzando microcolonne cromatografiche (il GFXTM PCR DNA and Gel Band Purification Kit, GE Healthcare, Buckingamshire, UK), seguendo la procedura suggerita dal fornitore. 3.4.11 Sequenziamento Il DNA eluito dalle bande excise dal gel è stato quantificato mediante migrazione elettroforetica su gel di agarosio all’1,5 % (p/v) utilizzando il marcatore di peso molecolare MassRuler TM DNA Ladder Mix (Amersham Bioscience, Piscataway, USA) caricato nel gel in due aliquote di 5 µL e 10 µL. Le reazioni di sequenza sono state effettuate dalla MWG Biotech (Ebersberg, Germany) a partire da aliquote di 20 ng/100-bp di DNA essiccato. 57 3. Materiali e Metodi 3.4.12 Allineamento delle sequenze di 16S rDNA Le sequenze in formato FASTA ottenute dalla MWG Biotech sono state allineate alle sequenze depositate nella banca dati GenBank, utilizzando il software BLAST disponibile liberamente in rete al sito http://ncbi.nlm.nih.gov (Altschul et al., 1997). 3.5 Analisi SPME-GC (Solid Phase Microextraction – Gas Chromatography) L’analisi SPME-GC è stata effettuata seguendo la procedura proposta da Pinho et al. (2001) e di seguito descritta: si pongono 3 g di campione finemente grattugiato in una fiala di vetro da 15 mL dotata di setto in PTFE (politetrafluoroetilene) (SUPELCO); si aggiunge 1 mL di acqua deionizzata, miscelando delicatamente; si incuba a 60°C per 40 min, ponendo la fiala in un bagnetto termostatico (fase di equilibratura); Figura 3.5. Rappresentazione schematica della fase di adsorbimento. si inserisce attraverso il setto in PTFE della fiala un supporto manuale (SUPELCO) per SPME equipaggiato con una fibra in poliacrilato di lunghezza 2 cm e spessore 85 µm (SUPELCO); si espone la fibra allo spazio di testa del campione, incubando a 60°C per 30 min (fase di adsorbimento); si inserisce la fibra nella porta dell’iniettore (posto in modalità splitless) del gascromatografo SHIMADZU GC-2014. Le analisi cromatografiche sono state effettuate secondo le condizioni riportate da Pinho et al (2001) e di seguito descritte: colonna DB-WAX (J&W Scientific): 30 m di lunghezza, 0,25 mm di diametro interno e 0,25 mm di spessore della fase stazionaria; temperatura della colonna: 40°C per 2 min seguiti da un incremento di 5°C/min fino ad una temperatura finale di 230°C mantenuta per altri 10 min (durata totale del programma 50 min); 58 3. Materiali e Metodi caratteristiche e temperatura dell’iniettore: modalità splitless, 230°C; gas carrier: azoto con un flusso di 1,47 mL/min; caratteristiche e temperatura del rivelatore: FID, 230°C. Al fine di determinare i tempi di ritenzione dei principali composti volatili che generalmente caratterizzano le produzioni casearie (Pinho et al, 2001; Chiofalo et al, 2004; Guillén et al, 2004), sono stati selezionati ed analizzati, sia singolarmente sia in miscela, 13 composti standard (SIGMA ALDRICH) (Tabella 3.1). Etilacetato n° atomi di Carbonio 4 n° di doppi legami - Peso Molecolare 88,11 Acetone 3 - 58,8 Etanolo 2 - 46,07 Acido acetico 2 0 60,5 Nome IUPAC Nome COMUNE Acido butanoico Acido butirrico 4 0 88,11 Acido esanoico Acido caproico 6 0 116,16 Acido eptanoico Acido enantico 7 0 130,18 Acido ottanoico Acido caprilico 8 0 144,21 Acido nonanoico Acido pelargonico 9 0 158,24 Acido decanoico Acido caprico 10 0 172,26 Acido undecanoico 11 0 186,29 Acido undecenoico 11 1 184,28 12 0 200,32 Acido dodecanoico Acido laurico Tabella 3.1. Composti standard selezionati per l’identificazione dei composti aromatici nei campioni di formaggio. A partire dalle soluzioni pure, sono state realizzate soluzioni in N-esano (concentrazione 150 mg/mL): 1 mL di ciascuna soluzione è stato, quindi, iniettato direttamente in colonna. 3.6 Elaborazione statistica I dati ottenuti mediante analisi chimico-fisiche, microbiologiche, molecolari e gas-cromatografiche sono stati sottoposti ad analisi della varianza (ANOVA) ad una via in associazione al calcolo del coefficiente di Tukey Kramer (P > 0,05) utilizzando il software JMP v 8 (Cary, NC, USA). Gli stessi dati sono stati sottoposti anche ad analisi PCA (Principal Component Analysis) e PLS-DA (Partial Least Squares-Discriminant Analysis) utilizzando il software SIMCA-P v 11.5 (UMETRICS). 59 3. Materiali e Metodi 3.7 Consumer test Otto dei 29 formaggi oggetto di studio sono stati selezionati e sottoposti ad un Consumer test. In totale sono stati reclutati 144 giudici non addestrati, che ci hanno consentito di ottenere, per ciascuna tipologia di formaggio, un numero di responsi (72) tale da garantire l’attendibilità dei dati (Resurreccion AVA, 1998). La selezione dei giudici è stata basata su sesso ed età, con la finalità di definire un gruppo di potenziali consumatori rappresentativo del profilo demografico nazionale (ISTAT 2005). I formaggi testati sono stati acquistati in forme intere, conservati secondo le modalità indicate dai produttori e successivamente porzionati in aliquote delle dimensioni di 1,5 x 1,5 x 5 cm, come proposto da Lavanchy et al., 1999. Gli otto formaggi selezionati per il presente Consumer test sono stati suddivisi in 4 differenti tipologie, quali Caprino, Pecorino, Caciotta e Caserotto; quest’ultimo, che risulta incluso nella categoria delle caciotte per le analisi chimico-fisiche, microbiologiche e relative al profilo aromatico, consiste in realtà in una produzione casearia innovativa ottenuta con latte crudo vaccino addizionato, in fase di coagulazione, di olio di oliva di varietà Raggia, tipica della provincia di Ancona. Ad ogni consumatore sono state servite due coppie di formaggi, ognuna comprendente due formaggi appartenenti alla medesima tipologia, di cui uno a latte crudo ed uno a latte pastorizzato aggiunto di starter commerciali; il Caserotto in questo caso è stato confrontato con un prodotto che ovviamente non presenta le medesime caratteristiche peculiari ma viene ottenuto comunque con latte vaccino, analogamente al Caserotto stesso. La somministrazione dei formaggi, serviti a temperatura ambiente, è stata effettuata seguendo uno schema monadico sequenziale (Resurreccion AVA, 1998). I quesiti sottoposti ai giudici sono stati formulati evitando risposte aperte e prevedendo una scala di valutazione in 9 punti (Peryam e Pilgrim, 1957). Al termine del questionario è stato chiesto di esprimere, per ciascuna tipologia di formaggio, la preferenza verso la produzione a latte crudo o pastorizzato. La necessità di presentare i campioni in forma anonima ha portato alla scelta di una procedura di codifica degli stessi, utilizzando un codice randomizzato a 3 cifre. Il Consumer test è stato condotto in un laboratorio di analisi sensoriale per garantire: i) la standardizzazione delle condizioni ambientali; ii) la riduzione delle distrazioni psicologiche e delle interazioni tra i soggetti chiamati a svolgere il test; iii) il raggiungimento di un livello di comfort adeguato. L’elaborazione statistica dei dati è stata condotta utilizzando il software Statistica (versione 5.1, StatSoft Inc., Tulsa) attraverso l’analisi della varianza (ANOVA) ad una via in associazione al calcolo del coefficiente di Tukey Kramer (P > 0,05). 60
Scarica