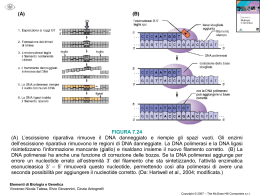
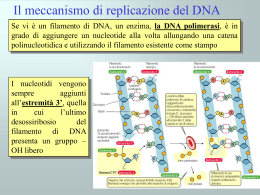
La Struttura del DNA La struttura laterale del DNA è composta da unità ripetute ed alternate di gruppi fosfato e di 2deossiribosio, uno zucchero pentoso (a cinque atomi di carbonio) che si lega ai fosfati adiacenti attraverso legami fosfodiesterici presso il terzo ed il quinto carbonio. Conseguenza di questi legami asimmetrici è il fatto che ogni filamento di DNA ha un senso. In una doppia elica, il senso di un filamento è opposto a quello del filamento complementare. Per tale motivo, i due filamenti che costituiscono una doppia elica sono detti antiparalleli. Le estremità asimmetriche di un filamento di DNA sono definite estremità 5′ (cinque primo) ed estremità 3′ (tre primo). Figura riportata nella pubblicazione originale di Watson e Crick Nature, Aprile 1953 La larghezza è sempre di 22-26 Å ed è costante. Infatti i “pioli” che formano questa scala sono divisi in 2 gruppi: pirimidinici (Guanina e Adenina) che sono più grandi rispetto alle purine (Citosina e Timina). L’ Appaiamento delle Basi Ogni tipo di base presente su un filamento forma un legame con la base posta sul filamento opposto. Tale evento è noto come appaiamento complementare. Le basi puriniche formano legami idrogeno con le basi pirimidiniche: Adenina può legare solo Timina e Guanina può legare solo Citosina. L'associazione di due basi viene comunemente chiamata paio di basi ed è l'unità di misura maggiormente utilizzata per definire la lunghezza di una molecola di DNA. Dal momento che i legami idrogeno non sono covalenti, essi possono esser rotti e riuniti in modo relativamente semplice. I due filamenti possono essere allontanati tra loro come avviene per una cerniera sia dalle alte temperature che da un'azione meccanica (come avviene durante la replicazione del DNA). Conseguenza di questa complementarietà è il fatto che tutte le informazioni contenute nella doppia elica possono essere duplicate a partire da entrambi i filamenti, evento fondamentale per una corretta replicazione del DNA. L’appaiamento delle basi implica la formazione di legami idrogeno H citosina N H N O N sugar H O H CH3 timina H sugar N O N N H O N N N N H H N N N N sugar adenina N sugar guanina I Legami fra le Basi Azotate I due tipi di paia di basi formano un numero differente di legami idrogeno: A e T ne formano due, G e C tre. Per tale motivo, la stabilità del legame GC è decisamente maggiore di quello AT. Di conseguenza, la stabilità complessiva di una molecola di DNA è direttamente correlata alla frequenza di GC presenti nella molecola stessa, nonché alla lunghezza dell'elica stessa: una molecola di DNA è dunque tanto più stabile quanto più contiene GC ed è lunga. Un'altra conseguenza di tale evento è il fatto che le regioni di DNA che devono essere separate facilmente contengono un'elevata concentrazione di A e T. In laboratorio, la stabilità dell'interazione tra filamenti è misurata attraverso la temperatura necessaria a rompere tutti i legami idrogeno, chiamata temperatura di melting (o Tm). Quando tutti i legami idrogeno sono rotti, i singoli filamenti si separano e possono assumere strutture molto variegate. La doppia elica del DNA ha 2 scanalature: una principale e una secondaria. La doppia elica è una spirale destrorsa (si avvolge in senso orario). Con l'avvitarsi su sé stessi dei due filamenti, restano esposti dei solchi tra i diversi gruppi fosfato. Il solco maggiore è largo 22 Å, mentre il solco minore è largo 12 Å. La differente ampiezza dei due solchi si traduce concretamente in una differente accessibilità delle basi, a seconda che si trovino nel solco maggiore o minore. Proteine come i fattori di trascrizione, dunque, solitamente prendono contatto con le basi presenti nel solco maggiore. 20 Senso e Antisenso Una sequenza di DNA è definita senso se la sua sequenza è la stessa del relativo mRNA. La sequenza posta sul filamento opposto è invece detta antisenso. Dal momento che le RNA polimerasi lavorano producendo una copia complementare, il filamento necessario per la trascrizione è l'antisenso. Sia nei procarioti che negli eucarioti vengono prodotte numerose molecole di RNA antisenso a partire dalle sequenze senso. La funzione di questi RNA non codificanti non è stata ancora completamente chiarita. Si ritiene che gli RNA antisenso possano giocare un ruolo nella regolazione dell'espressione genica. Esistono alcune sequenze di DNA, sia in procarioti che in eucarioti (ma soprattutto nei plasmidi e nei virus) in cui la differenza tra sequenze senso ed antisenso è meno chiara, dal momento che le sequenze di alcuni geni si sovrappongono tra loro. In questi casi, dunque, alcune sequenze rivestono un doppio compito: codificare una proteina se lette in direzione 5'→3′ su un filamento; codificarne un'altra se lette sull'altro (sempre in direzione 5'→3′). Nei batteri, questa sovrapposizione genica è spesso coinvolta nella regolazione della trascrizione, mentre nei virus il fenomeno è dovuto alla necessità di contenere in un piccolo genoma un'elevata quantità di informazioni. Un altro modo di ridurre le dimensioni genomiche è quello individuato da altri virus, che contengono molecole di DNA lineare o circolare a singolo filamento. DANNI AL DNA Il DNA può essere danneggiato dall'azione di numerosi agenti, genericamente definiti mutageni. Tra di essi figurano ad esempio agenti ossidanti, agenti alchilanti ed anche radiazioni ad alta energia, come i raggi X e gli UV. Il tipo di danno causato al DNA dipende dal tipo di agente: gli UV, ad esempio, danneggiano il DNA generando la formazione di dimeri di timina, costituiti da ponti aberranti che si instaurano tra basi pirimidiniche adiacenti. Agenti ossidanti come i radicali liberi o il perossido di idrogeno, invece, producono danni di tipo più eterogeneo, come modificazioni di basi (in particolare di guanine) o rotture del DNA a doppio filamento. Gli acidi nucleici possono formare vari tipi di doppia elica Il DNA esiste in diversi tipi di conformazione. Esse sono denominate A-DNA, B-DNA, CDNA, D-DNA, E-DNA, H-DNA L-DNA,, P-DNA e Z-DNA. In ogni caso, solo A-DNA, B-DNA e Z-DNA sono stati osservati in sistemi biologici naturali. La conformazione del DNA può dipendere dalla sequenza, dal superavvolgimento, dalla presenza di modificazioni chimiche delle basi o dalle condizioni del solvente, come la concentrazione di ioni metallici. Di tali conformazioni, la conformazione B è la più frequente nelle condizioni standard delle cellule Le due conformazioni alternative sono differenti dal punto di vista della geometria e delle dimensioni. La forma A è un'ampia spirale destrorsa (il solco minore è largo ma poco profondo, quello maggiore è più stretto e profondo), con un passo di 2,9 nm (circa 11bp) ed un diametro di 2,5 nm. Tale conformazione è presente in condizioni non fisiologiche, quando il DNA viene disidratato. In condizioni fisiologiche, è la conformazione che caratterizza gli eteroduplex di DNA e RNA e che può formarsi nelle associazioni DNA-proteina. La conformazione Z è tipica invece delle sequenze che presentano modificazioni chimiche come la metilazione, in tratti ricchi di basi C e G. Essa assume da un andamento sinistrorso, opposto rispetto alla conformazione B Ha un passo di 4,6 nm ed un diametro di 1,8 nm, il solco maggiore più superficiale ed il solco minore stretto; deve il suo nome all'andamento a zig-zag che la caratterizza. Queste strutture inusuali possono essere riconosciute da specifiche Z-DNA-binding proteins, con conseguenze notevoli nella regolazione della trascrizione. destrorsa Fonti •Wikipedia.org •Molecularlab.it •Parcogeneticasalute.it
Scaricare