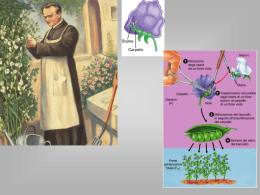
Lezione V-VI martedì 18-X-2011 corso di genomica laurea magistrale Biotecnologia Industriale aula 8 orario : Martedì ore 14.00 - 16.00 Giovedì ore 13.00 - 15.00 non ci sarà lezione martedì 1 e giovedì 3 Novembre D. Frezza se vi indignate Non prendetevela con me ! Certo si potrebbe protestare di più e meglio sullo scarso investimento sulla cultura, ma è un ventennio che nessuno ci investe ed i risultati si vedono Poca tecnologia nei nuovi investimenti a tutti i livelli Questo per esempio è un settore in grande sviluppo ma per operarci in Italia non ci sono molti spazi perché le riflessioni su evoluzione È come dover parlare di architettura senza sapere con quali materiali si costruisce Nell’800 è stato iserito l’uso dell’acciaio in edilizia(tour Eiffel 1889) Anche i romani conoscevano il calcestruzzo ed il ferro però ne producevano di meno Quic kTime™ e un dec ompres sore TIFF (Non c ompres so) s ono nec es sari per visualiz zare ques t'immagine. Poi il cemento armato ha permesso strutture molto più leggere Senza sapere come sono venuti fuori i genomi la descrizione sarebbe piatta e di difficile comprensione polarismi scoperta con i sequenziamenti procarioti ed eucarioti : variazione e conservazione mettiamo insieme queste due evenienze formamlmente in opposizione. abbiamo visto che esiste una polarità una discrepanza logica che appare spesso in natura, la polarità è compatibile senza dover eliminare una delle due forme polari ? come possiamo rendere compatibile una contrapposizione così forte? la risposta la sapete già: è possibile i meccanismi che rendono possibile abbiamo detto che giocano sulla variabile tempo gestire il polarismo salviamo il polarismo utilizzando questa contraddizione, possono coesistere entrambe: variabilità e conservazione il primo nei tempi lunghi, il secondo nel tempo breve Abbiamo già detto: era implicita in Mendel la conservazione era implicita in Darwin la variabilità delle specie una nell’altra Sembrava difficile trovare la soluzione quando è stata scoperta la mutagenesi si è aperta una nuova finestra per interpretare questi due fenomeni! la variabilità come mutazione In the 1920s, Hermann Muller discovered that x-rays caused mutations in fruit flies. He went on to use x-rays to create Drosophila mutants that he used in his studies of genetics. He also discovered that x-rays not only mutate genes in fruit flies but also have effects on the genetic makeup of humans.[1] The first mutagens to be identified were carcinogens, or cancer-causing substances. Early physicians detected tumors in patients more than 2,000 years before the discovery of chromosomes and DNA. In 500 B.C., the Greek Hippocrates named crab-shaped tumors cancer, meaning crab. 1 Campbell, Neil A. and Jane B. Reece. Biology. 7th ed. San Francisco, CA: Pearson Education, Inc, 2005. scoperta legata ad un evento tragico chi scoprì il fenomeno legato alla variabilità genetica se ne accorse associando l’uso di sostanze tossiche belliche ad eventi di trasformazione genetica: le mutazioni prima Muller con Drosofila e raggi X, poi UV e poi la chimica: mostarda azotata, azoiprite ed altre in seguito, scoperte dalla Charlotte Auerbach mutagenesi chimica The discovery of Mustard Gas by Charlotte Auerbach and Robson Science. 1947 Mar 7;105(2723):243-7. The Chemical Production of Mutations. Auerbach C, Robson JM, Carr JG. Nature. 1946 Mar 9;157:302. Chemical production of mutations. AUERBACH C, ROBSON JM. Perspectives on genetics: anecdotal, historical, and critical ... - Google Books Result James Franklin Crow, William F. Dove - 2000 - Medical - 723 pages June 1993 The Discovery of Mustard Gas Mutagenesis by Auerbach and Robson in ... had been obtained from experiments on chemical mutagenesis prior to 1940. ... books.google.it/books?isbn=029916604X... bilanciato il polarismo? possono coesistere variabilità e conservazione studio dei genomi tramite sequenziamento completo slancio nella ricerca genomica e sviluppo di nuove tecniche nuove applicazioni di tecniche già note per sveltire i metodi i genomi a disposizione le nuove evidenze: procarioti compatti ma senza meiosi (aploidi) eucarioti con nuove strutture per la diploidia la complicazione a partire dagli eucarioti unicellulari lieviti, funghi, (Saccharomyces, Neurospora) strutture di base invariate già dai procarioti: RNA, ribosomi, tRNA, polimerasi, membrane, la conoscenza negli eucarioti strutture paraloghe che assumono nuove funzioni (omologie analogie) ma le nuove strutture ancora non si conoscono si parte dai geni, ma sono il 5% TUTTO IL RESTO é nuovo e questa è GENOMICA si sapeva che c’era il DNA ripetuto: trasposoni, regioni GC rich, mini e microsatelliti e poi? Si consideravano regioni ricche di AT poco informative i meccanismi dell’evoluzione elementare i processi evolutivi sono lontani da essere compresi nella complessità alcuni sono evidenti dalle analisi genomiche più semplici come abbiamo detto i confronti “evolutivi” ci aiutano i confronti ci parlano del rapporto tra funzione e strutture i geni divergono e si duplicano Geni ortologhi e geni paraloghi Geni ortologhi: geni simili riscontrabili in organismi correlati tra loro. Il fenomeno della speciazione porta alla divergenza dei geni e quindi delle proteine che essi codificano. es. la -globina di uomo e di topo hanno iniziato a divergere circa 80 milioni di anni fa, quando avvenne la divisione che dette vita ai primati e ai roditori. I due geni sono da considerarsi ortologhi. Geni paraloghi: geni originati dalla duplicazione di un unico gene nello stesso organismo. es. -globina e -globina umana hanno iniziato a divergere in seguito alla duplicazione di un gene globinico ancestrale. I due geni sono da considerarsi paraloghi. variabilità per divergenza Gene ancestrale duplicazione genica Gene A Gene B speciazione Gene A1 ortologhi Gene A2 paraloghi Gene B1 Specie 1 ortologhi Gene B2 Specie 2 alla scoperta del genoma: allontanandosi dai geni (cod. geni < del 5% del genoma) ricerca delle funzioni: analogia con il periodo precedente alla scoperta del codice genetico interpretazioni: studi attraverso i confronti su modelli i modelli sono validi perchè è esistita l’evoluzione conclusione: il genoma è dinamico a breve e lungo termine come ha fatto ad attrezzarsi per poter evolvere? come mai si usano organismi modello: ribadiamolo è possibile perchè ci sono: - i geni ortologhi (stessa origine nell’ancestrale e divergenza nelle specie successive), hanno la stessa funzione nelle specie diverse - i geni paraloghi derivano da duplicazioni e possono mantenere funzioni simili (DNA binding, kinasi ecc.) (l’evoluzione rimane la premessa inevitabile) l’unica spiegazione sembra autocelebrativa, tautologica il genoma non poteva esistere senza interazione (per definizione) se la vita è nata nel mondo ad RNA e poi è venuto il DNA ed ha cominciato ad interagire con proteine diventandone dipendente, ma anche il responsabile, l’informazione genetica non poteva non essere dinamica. come sarebbe il genoma non interattivo? da dove parte l’interattività del genoma non c’è funzione che non preveda interazione con altre strutture ed enzimi, l’informazione passa attraverso interazioni - potrebbe risultare passivo quando deve essere replicato/duplicato; la struttura rende possibile ogni altra funzione che già conosciamo o che è ancora da individuare - sembra passivo ma non lo è nemmeno per la sintesi dell’mRNA, manda segnali e ne riceve continuamente cambia strutturalmente : per il differenziamento: epigenetica meiosi: gameti: zigote: embrione: adulto interazione del genoma nelle varie fasi sotto il nome della regolazione la struttura (anatomia) informazioni per le funzioni anche solo per la trascrizione la cromatina si altera la trasformazione inizia molto prima della trascrizione trascrizione = una delle funzioni finali non c’è soluzione di continuità l’interazione non è saltuaria tutto il ciclo vitale di ogni organismo prevede interazione dinamica ad ogni passaggio il DNA-cromatina subisce trasformazione, alterazioni dallo zigote alle mitosi al differenziamento, ogni fase prevede quella successiva,il genoma riceve messaggi continuamente per sapere se fermarsi, procedere e cambiare funzioni ovvio ma fondamentale l’interazione genoma “ ambiente “ è continuo gli stimoli devono permettere la reattività ci deve essere esecuzione del programma ci deve essere flessibilità il programma può andare avanti se c’è “feedback” bidirezionalità della trasformazione funzionale genoma (cromatina) ambiente: nucleo, citoplasma, extracellulare.....ecc. quando il genoma si attiva è perchè c’è l’ambiente giusto per far partire determinate funzioni come si inserisce la variabilità le differenze esistono, si sono accumulate, ad ogni meiosi si rimescolano ogni organismo che nasce ha un genoma “ricombinante” cosa comporta questa diversificazione ? microvariabilità i polimorfismi : genetica quantitativa Interattoma modulabile (non solo tra proteine) modulazione dovuta ad effetti dei polimorfismi (osservati col confronto tra fenotipi/genotipi) turn-over (rapidità-lentezza) effetti a cascata (attivazioni tramite trasformazioni posttraduzionali: fosforilazioni, metilazioni, glicosilazioni ecc.) trasduzione del segnale tipica dell’interazione intra-extra cellulare e citoplasmatica - nucleare dal nucleo al genoma e viceversa (ciclo continuo) andata e ritorno l’informazione arriva all’informatore e riparte (difficile separare i ruoli nel ciclo continuo) è un artificio porre un inizio e scegliere lo zigote come start-point, si potrebbero prendere i gameti o le gonadi o l’individuo adulto la trasduzione del segnale mette in comunicazione le varie parti della cellula e quindi dell’organismo ogni punto di un ciclo può essere scelto come inizio o fine adesso prendiamo il genoma come nostro riferimento come possiamo approcciare il genoma in maniera globale la post genomica (di già?) dopo il sequenziamento dei genomi interi ancora una volta si scopre di non sapere è l’inizio del salto verso la genomica regolativa / o dello studio del genoma non tradotto è riduttivo credere che si conosca il genoma conoscendo solo la porzione codificante conseguenza: una parte del trascrittoma non è noto ignorando una bella parte delle funzioni del genoma post genomica non significa che la genomica sia superata, va capito che non basta conoscere le sequenze genomiche approcci globali e olistici nuove metodologie trascrittoma proteoma - interattoma ma il restante 95%? 95% ? wide genome screening screening con tecniche diverse studio nuovo cosa si può analizzare in che maniera genomica obbligatoriamente porta al confronto tra i vari possibili organismi modello o intraspecie centralità del modello umano oppure riferimento al modello umano (ricchezza di polimorfismi) ricerca di modelli per trovare semplificazioni e generalizzazioni cosa si vuole vedere ? come sempre: strutture e variabilità micro-arrays per ibridazione resequencing shot-gun parziali o globali SNPs e VNR ogni 500 bp del genoma i vari approcci i diversi modelli permettono di produrre i nuovi approcci e le diverse tecniche dalle domande generali a quelle più specifiche verso i meccanismi sottostanti allargare l’interattoma anche alla interazione col genoma pensare in maniera globale cercare di essere onnicomprensivi (presuntuoso ma necessario) il problema globale rischio di genericità tutto o niente lo studio con le libraies è stato l’inizio i metodi globali: osservazioni col grand’angolo necessità di non perdere anche i particolari guardare dal satellite ma con una risoluzione altissima osservazione diretta e indiretta in quasi tutte le scienze si passa alla oservazione indiretta il DNA o le proteine non le vediamo, abbiamo prove indirette ci fidiamo delle osservazioni indirette ed il metodo passa attraverso l’uso dei controlli ossia della contraffabilità del risultato ancora di più vale per bioinformatica in cui si lavora sotto simulazione convergenza delle osservazioni e riprove l’uso di organismi modello come riprova della universalità osservazioni riproposte anche su organismi di specie diverse difficoltà di riscontrare patologie in organismi molto diversi possibilità di osservare fenotipi simili in organismi anche molto diversi generalità di un fenomeno riscontrabile su altre specie (quanto più è generale) troppi esempi a riprova dell’universalità dei promotori eucariotici e procariotici : tutti i costrutti inducibili o costitutivi transfettati cosa possiamo fare per capire il genoma nell’era post-genomica mantenere la capacità di analisi nelle interazioni / variazioni nel contesto e fuori contesto in vitro veritas fenotipi mutanti e polimorfismi come può essere l’approccio sperimentale? dove si guarda: nei possibili target (regioni predette) o con un sistema non “bias” onnicomprensivo senza selezione cercando le funzioni genomiche creazione di esche per trovare con cosa interagiscono riprova in vivo con l’organismo transgenico per avere una conferma approccio simmetrico da un fenotipo ricerca del fenomeno da riprodurre in vitro verificare se lo stesso costrutto interagisce con le stesse proteine Esempio: tentativi non sempre di successo con two-hybrid systems, le tecniche hanno delle approssimazioni esca e oggetto interagente (isolamento e conferma in vitro) transcription factories Passando alla struttura del genoma, nuove tecniche ed ipotesi Simposio sulla struttura e funzioni del genoma Cold Spring Harbor Symposia on Quantitative Biology CSH 17 maggio 2011, vol. LXXV CH Eskiw, NF Cope, I Clay, S Schoenfelder, T Nagano and P Fraser. Laboratory of Chromatin and Gene Expression, The Babraham Institute, Cambridge CB22 3AT, United Kingdom Correspondence: [email protected] abstract transcript fact.CSHQ The dynamic compartmental organization of the transcriptional machinery in mammalian nuclei places particular constraints on the spatial organization of the genome. The clustering of active RNA polymerase I transcription units from several chromosomes at nucleoli is probably the best-characterized and universally accepted example. RNA polymerase II localization in mam- malian nuclei occurs in distinct concentrated foci that are several-fold fewer in number compared to the number of active genes and transcription units. Individual transcribed genes cluster at these shared transcription factories in a nonrandom manner, pref- erentially associating with heterologous, coregulated genes. We suggest that the three-dimensional (3D) conformation and rel- ative arrangement of chromosomes in the nucleus has a major role in delivering tissue-specific geneexpression programs. riassunto art. Eskiw- Fraser Il sistema dinamico della compartimentazione della macchina della trascrizione nel nucleo dei mammiferi pone dei limiti alla organizzazione spaziale del genoma. La localizzazione delle unità attive di trascrizione della RNA pol I di parecchi cromosomi nei nucleoli è l’esempio meglio caratterizzato ed accettato universalmente. I foci della RNA pol II concentrati nei nuclei di mamm. sono di meno rispetto al numero di geni ed alle unità di trascrizione. Unità di trascrizione di cluster di geni sono associate in maniera non random a “transcription factories” condivise in preferenza con geni eterologhi coregolati. Suggeriamo che la conformazione 3D e arrangiamenti relativi dei cromosomi nel nucleo abbiano un ruolo importante per attivare programmi di espressione genica tessuto specifica. introduzione dell’articolo Nella introduzione si parla di molti argomenti di questo corso di genomica. E’ grande interesse però è una storia che vi dovete leggere voi e di cui in gran parte vi ho già parlato nelle lezioni introduttive sulla genomica, per definirla ed identificarla. Differente conoscenza Tra nucleo e citoplasma QuickTime™ e un decompressore TIFF (Non compresso) sono necessari per visualizzare quest'immagine. del nucleo si sa abbastanza poco: nucleolo, nucleoplasma, membrana interna/esterna, pori, cromatina (euetero), ribosomi, ~matrice nucleare QuickTime™ e un decompressore TIFF (Non compresso) sono necessari per visualizzare quest'immagine. del citoplasma: vacuoli, RE, centrioli, lisosomi, mitocondri, Golgi app., vescicole, citosol, citoscheletro, pori etc. attività trascrizionale dei geni Trascrizione dei geni più attivi discontinua e ad impulsi Geni singoli alternano brevi periodi ad alta attività trascrizionale a periodi inattivi di durata variabile. Il 90% degli alleli di e globina sono attivi allo stadio corretto con analisi FISH (fluoresc. in situ hybridization) Topi transgenici con LCR deleta mostrano un pattern on / off dinamico suggerendo che la presenza stabilizzi lo stato on. Tutte le cellule esprimono un basso livello di globina, ma solo una frazione mostra segnali di trascrizione con RNA FISH. Per la maggior parte degli altri geni con FISH con sonde introniche si vede che geni attivi sono “off” per la maggior parte del tempo. (LCR = locus control region) conseguenze regolative La conseguenza di questi stadi di inattività prolungata è che questi geni possono essere modulati variando i livelli di espreessione genica alternando dinamicamente le fasi “on” e fasi “off”. Molto diverso che non modulare una fase di trascrizione continua rallentandola o accelerandola. geni associano dinamicamente con “transcription factories” Le TF (transcription factories) sono foci nucleari discreti che contengono alte conc. di RNA pol II fosforilata e sti di produzione di RNA nascente. Analisi di geni singoli con immuno-FISH mostra invariabilmente segnali associati ai foci di RNApol II con la nascente sintesi di RNA nelle TF. Analisi con immuno-FISH mostrano che gli alleli inattivi di geni attivi sono lontani dalle TF. la cromatina dinamica nei territori occupati Ipotesi : la dinamica della cromatina porta molti geni in attività a ricollocarsi fuori dai territori cromosomici I geni perciò si muovono in TF preassemblate quando sono attivi ? Inoltre i movimenti dinamici della cromatina hanno un ruolo nella regolazione della trascrizione muovendosi dentro e fuori dalle TF? gene modello -globina Tecnica RNA-FISH cellule eritroidi in fase di maturazione associazione di quasi tutti gli alleli nelle TF Doppia marcatura di geni lontani attivi dello stesso cromosoma associano e condividono gli stessi TF dei geni della globina ad alte frequenze. Geni a megabasi di distanza colocalizzano al 40-50% di più di una localizzazione random. Colocalizzazione di geni distanti con un’altra tecnica: 3C chromosome conformational capture. altre evidenze con linfociti B murini Linfociti B murini “resting” di milza esprimono alti livelli di IgH Quasi tutti i loci delle catena IgH mostrano segnali RNAFISH associati a TF - è un sist. modello come la globina per le cellule eritroidi - induz. di geni early (Fos, Myc) in B resting - Fos e Myc non erano espressi al t.0 - dopo 5 minuti la > parte di alleli F e M erano trascritti ed associati nei foci con RNA pol II; al 30% Fos sta a ~30 Mb dal locus IgH sul chrms 12 di topo - Myc sul crms 15 risulta al 25% nelle TF del locus IgH
Scaricare