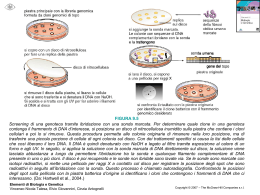
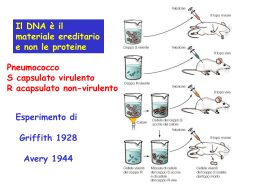
SVILUPPO DI UNA METODICA DI CGH-ARRAY FINALIZZATA ALLA REALIZZAZIONE DI UN MICROARRAY PER LA CARATTERIZZAZIONE GENETICA DI SOGGETTI AFFETTI DA RITARDO MENTALE IDIOPATICO S.Gatto1, G. Nannetti2, G. Marseglia2, A. Magi2, F. Torricelli3 1 AB ANALITICA srl., Padova ITALIA; 2 Laboratorio di Citogenetica, Ospedale Careggi , Firenze; 3CONSORZIO CEO (CENTRO DI ECCELLENZA OPTRONICA) Firenze INTRODUZIONE L'avvento della CGH-Array, descritta per la prima volta nel 1997, ha aperto la strada alla possibilità di una più alta e più ampia risoluzione nella rilevazione delle anomalie cromosomiche strutturali. La CGH-Array si basa sugli stessi principi della CGH classica ma a differenza di quest’ultima (che prevede l’utilizzo di un supporto vitreo con cromosomi in metafase), è stata sviluppata sostituendo i cromosomi delle metafasi di riferimento con un supporto vitreo chimicamente modificato su cui vengono stampate sonde di DNA (cloni BAC e PAC) complementari a loci specifici di cromosomi umani. L’utilizzo di tali cloni permette una correlazione immediata tra l’eventuale anomalia cromosomica e una precisa posizione nel genoma. Dai dati riportati in letteratura, il CGH Array è stata utilizzata con successo per tracciare il profilo genomico in una vasta gamma di tumori presentandosi come il metodo più utile per rilevare e localizzare simultaneamente perdita o guadagno di materiale genetico. Per tale motivo la sua applicazione è in rapido incremento anche allo studio di anomalie cromosomiche costituzionali. SCOPO Lo scopo del lavoro è stato quello di sviluppare la metodica di CGH-array, dalla stampa dei vetrini al protocollo di ibridazione, per la costruzione “personalizzata” di array locus specifici. Ciò significa che una volta messo a punto il metodo, è possibile stampare vetrini utilizzando sonde che siano specifiche per il caso in esame e che permettano di analizzare contemporaneamente più regioni cromosomiche di interesse. Come modello iniziale abbiamo scelto di studiare una casistica di circa 100 soggetti affetti da ritardo mentale idiopatico precedentemente caratterizzati nel nostro laboratorio mediante Hr-CGH (High Resolution CGH) e metodica FISH. MATERIALI E METODI L’approccio metodologico corrente per la costruzione degli array prevede l’utilizzo di vettori batterici (BAC , PAC e cosmidi) per il clonaggio dei frammenti di DNA di interesse. Le sonde utilizzate nel nostro lavoro, lunghe circa 150 Kb, sono in grado di ibridare a livello delle regioni subtelomeriche dei cromosomi umani e sono clonate in E. coli in vettori BAC (Bacterial Artificial Chromosome) e PAC (P1-derived Artificial Chromosome). La sensibilità e la specificità di tali sonde è stata validata mediante metodica FISH allestita su metafasi di soggetti sani. Per ridurre il peso molecolare della sonda estratta dal vettore plasmidico abbiamo utilizzato la DOP-PCR (degenerate oligonucleotide-primer polymerase chain reaction), un metodo che utilizza primer degenerati in grado di amplificare il DNA umano (sonda d'interesse) a scapito del DNA del vettore batterico. La sonda viene così ridotta in frammenti di 300-3000 bp che, una volta purificati e depositati in piastra, vengono stampati su un supporto vitreo mediante l’utilizzo di una stazione robotizzata (Calligrapher MiniArrayBioOdyssey). Il robot è infatti dotato di un braccio meccanico che termina con delle punte (pin) in grado di prelevare nanolitri della sonda presente in piastra per poi depositarne una quantità uniforme sulla superficie dell’array. Ogni sonda stampata rappresenta una regione subtelomerica di interesse ed è presente sul vetrino almeno in triplice copia per minimizzare i possibili errori. L’esperimento di ibridazione viene eseguito coibridando sullo stesso array due campioni di DNA genomico: 1 g di DNA test e 1 g un DNA controllo (reference) marcati in maniera differenziale con molecole fluorescenti (Cy3/Cy5). Una volta ibridati, i vetrini sono sottoposti a scansione (Axon 4000B) ed analisi (GenePix Pro 4.1). Lo scanner e’ in grado di rilevare i livelli di fluorescenza (Cy3 e Cy5) di ogni spot; nell’immagine risultante i segnali Cy3 e Cy5 sono sovrapposti: i colori rappresentano il logaritmo in base 2 dei rapporti tra le intensita’ della fluorescenza Cy5 e Cy3 per ciascuno spot. Per l’analisi dei dati, l’informazione ottenuta dalla scansione del vetrino viene sottoposta ad un processo di normalizzazione globale (sottrazione del background e correzione mediana) in grado di rimuovere le distorsioni sistematiche presenti. I dati così ottenuti sono stati sottoposti ad inferenza statistica attraverso il test SAM (t-test modificato) per l’identificazione delle alterazioni cromosomiche. La successiva analisi statistica dei dati è quindi in grado di evidenziare le differenze significative tra test e controllo permettendo in tal modo di caratterizzare eventuali riarrangiamenti. Preparazione dell’array PREPARAZIONE DELL’ARRAY Analisi dell’immagine Vetrino CGH Array PREPARAZIONE DELLA SONDA Reference Test Estrazione DNA Cloni selezionati DNA reference DNA test Preparazione della sonda e della soluzione di stampa Stampa del DNA sul vetrino Marcatura con Cy3 uguale intensita’ dei due fluorocromi Marcatura con Cy5 incrementata presenza del test rispetto al reference incrementata presenza del reference rispetto al test CONCLUSIONI Come noto dalla letteratura, la CGH-Array si presenta come il metodo più utile per rilevare e localizzare simultaneamente in un unico esperimento di ibridazione, perdita o guadagno di materiale genico. Le potenzialità di questa metodica permetteranno alla CGH-Array di divenire una delle metodologie dominanti nella diagnosi degli squilibri cromosomici anche se l'incapacità di evidenziare anomalie cromosomiche nei casi in cui non si abbia nè perdita nè guadagno di materiale genetico (traslocazoni reciproche, traslocazioni Robertsoniane, inversioni e inserzioni bilanciate) rappresenta una delle grosse limitazioni della tecnica. Questo progetto di ricerca ci ha consentito di verificare la fattibilità e l'applicabilità della tecnica CGH-ARRAY per la caratterizzazione genetica delle regioni subtelomeriche nei pazienti affetti da RMI. Attualmente ci si propone di consolidare questo risultato con la trasformazione del prototipo in kit diagnostico. BIBLIOGRAFIA Lapierre JM, Cacheux V, Collot N, Da Silva F, et al. Ann Genet. 1998;41(3):133-40 Kallioniemi A, Kallioniemi OP, Sudar D et al. Science. 1992 Oct 30;258(5083):818-21 Fiegler H, Carr P, Douglas EJ et al. Genes Chromosomes Cancer. 2003 Jun;37(2):223 Pinkel D, Segraves R, Sudar D et al. Nat Genet. 1998 Oct;20(2):207-11 Wilhelm M, Veltman JA, Olshen AB et al. Cancer Res. 2002;62(4):957-60 Buckley PG, Mantripragada KK, Benetkiewicz M et al. Hum Mol Genet. 2002 Dec 1;11(25):3221-9 Flint J, Thomas K, Miclen G, Raynham H et al. Nat Genet 15(1997) Veltman JA, Schoenmakers EF, Eussen BH et al. Am J Hum Genet. 2002 May;70(5):1269-76. Epub 2002 Apr 09 Gentleman RC, Carey VJ, Bates DM, Bolstad B et al.Genome Biol 2004; 5:R80. Tusher VG, Tibshirani R, Chu G. Proc Natl Acad Sci U S A. 2001 Apr 24;98(9):5116-21. Progetto finanziato dalla Regione Veneto (Azione Biotech 1) AB ANALITICA srl, Via Svizzera 16-35127 PADOVA Tel +39 049 761698 - Fax +39 049 8709510 e-mail: [email protected] www.abanalitica.it Poster CGH Az. Biotech1_Rev.041007 Coibridazione del DNA test e reference
Scaricare