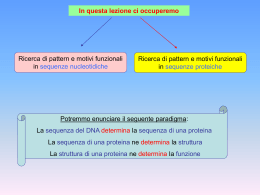
Università degli Studi di Padova C.L. Biologia Molecolare A.A. 2010/2011 Corso di Biologia Molecolare 2 Relazione di Laboratorio Studentessa: Silvia Bacco È un tool di Expasy; permette di ricercare motivi proteici nel database di Prosite; si può utilizzare nelle modalità: Quick Scan o Advanced Scan; è disponibile all’indirizzo web: http://expasy.org/tools/scanprosite/ Dopodiché si può avviare la ricerca contro i motivi di Prosite premendo Si inseriscono da 1 a un massimo di 8 sequenze per riga in formato FASTA UniprotKB oppure il CA identificativo o la carta d’identità nell’apposito spazio. 1 - Sequenza in formato FASTA - codice identificativo della sequenza AC o ID - un profilo di Prosite tramite codice identificativo dello stesso (AC o ID) - inserire sequenze multiple una sotto l’altra (fino ad un massimo di otto se confrontate con tutti i profili Prosite o fino a 16 se confrontate con più di un profilo e 1000 se confrontate con un solo profilo) Se dobbiamo inserire pattern bisogna considerare l’altro box dell’interfaccia come indicato in 2 Per inserire sequenze proteiche bisogna considerare l’apposito box dell’interfaccia del programma e attenersi alle regole indicate in 1 (per Default si escludono i motivi con alta probabilità di occurrence; si può anche scegliere di non scansionare profili) 2 -inserire il pattern (usando la convenzione di Prosite) -inserire i profili multipli uno dopo l’altro con il proprio AC/ID o pattern compilato a mano (al massimo 8 se li cerco in riferimento a un database oppure 16 se contro una proteina) Nella sezione Protein Database(s) è possibile selezionare database diversi (come UniprotKB/Swiss-Prot, UniProtKB/trEMBL, PDB) nei quali effettuare la ricerca e si possono eventualmente includere le varianti di risposta dovute a splicing alternativo (ed escludere i frammenti). Randomize database: (no per Default) nel caso in cui abbiamo inserito dei pattern da confrontare con le banche dati si può utilizzare per avere un riscontro di maggiore accuratezza tramite la modalità reverse di sequenze o tramite la modalità shuffle. Nella sezione Filter(s) si possono inserire dei filtri di ricerca sulla tassonomia o sulla descrizione. Per Default sono inseriti quelli per UniProtKB. Nella sezione Pattern si può ridurre il campo di ricerca a hits che sono stati trovati al massimo un numero X di volte (per Default è uguale a 1). Il Match mode permette di compiere delle ricerche scegliendo la combinazione di queste tre opzioni a seconda del nostro interesse: greedy che estende gli elementi alle lunghezze più variabili dei pattern; overlap che considera zone di sovrapposizione; includes che considera i match di riscontro di zone sovrapposte. Il formato può essere quello selezionato con rappresentazione grafica oppure simple HTML; plain text output; plain text FASTA output È possibile inserire un numero massimo di riscontri ottenibili e stabilire una soglia di hits per escludere quelli meno probabili che hanno punteggio basso. Mostra risultati con punteggi più bassi rispetto al valore soglia Se abilitato inserisce le informazioni complete della proteina di partenza accanto ai risultati Permette di ricevere le informazioni richieste direttamente nella propria casella di posta (soprattutto quando i riscontri sono più di quelli stabiliti per Default) La prima parte dell’output riguarda la ricerca di hits by profiles Passando col mouse sopra il dominio TDH si evidenzia in giallo la parte di sequenza associata al dominio La seconda parte dell’output riguarda la ricerca di hits by patterns. Ancora una volta passando col mouse sopra ADH_ZINC si evidenzia in giallo la parte di sequenza associatagli. In entrambi i casi cliccando sopra il dominio evidenziato dal riscontro degli hits (sia per i profiles che per i patterns) si apre una nuova schermata… … con la descrizione di struttura, funzioni, relazioni evolutive della proteina e i relativi link ad altre fonti… … la scheda tecnica in cui possiamo visualizzare ad esempio i consensus pattern, le strutture che si possono visualizzare con PDB… … con le referenze su autori che hanno studiato l’argomento e relativi link. Per ulteriori informazioni è disponibile il manuale d’uso on-line all’indirizzo: http://expasy.org/tools/scnpsit3.html
Scaricare