UNIVERSITA’ DI MILANO-BICOCCA
LAUREA MAGISTRALE IN
BIOINFORMATICA
Corso di
BIOINFORMATICA: TECNICHE DI BASE
Prof. Giancarlo Mauri
Lezione 3
Mappe genetiche
Alfabeti, parole, linguaggi
Alfabeto
=
insieme finito S di elementi detti lettere, caratteri o
simboli
Esempi
S = {0,1}
Alfabeto binario
S = {a, b, c, ... , v, z}
Alfabeto italiano
S = {A, C, G, T}
Alfabeto del DNA
S = {GLY, ALA, VAL, LEU, …}
Alfabeto delle proteine
2
Alfabeti, parole, linguaggi
Parola, stringa o sequenza su S
=
lista ordinata di simboli di S scritti consecutivamente da
sinistra a destra
Formalmente:
Una stringa w = a1a2…an è una funzione w: {1,2,…,n} S con:
w(i) = ai carattere i-esimo di w
n lunghezza di w (denotata anche con |w|)
ESEMPIO:
w = AATGCA
Parola vuota e
|w| = 6
|e| = 0
L’insieme delle parole su S
viene indicato con S* (chiusura
di S)
3
Alfabeti, parole, linguaggi
Sottosequenza di w
=
sequenza ottenuta per cancellazione di uno o più caratteri di w
Esempio
w = AATGCATTCGCT
Supersequenza di w’
w’= A TG AT CG T
Sottosequenza di w
4
Alfabeti, parole, linguaggi
Sottostringa di w
=
stringa formata da caratteri consecutivi di w
Esempio
w = AATGCATTCGCT
Superstringa di w’
w’=
Sottostringa di w
TGCATTC
Una sottostringa di w è anche
sottosequenza di w (ma non vale
il viceversa)
5
Alfabeti, parole, linguaggi
Concatenazione di w e v, wv
=
stringa formata dai caratteri di w, seguiti da quelli di v
Esempio
v = AATGC
w = ATTCGCT
vw = AATGCATTCGCT
6
Alfabeti, parole, linguaggi
Prefisso di w
=
stringa v tale che w = vt per qualche t S*
Esempio
w=AATGCATTCGCT
Suffisso di w
=
stringa t tale che w = vt per qualche vS*
Esempio
w=AATGCATTCGCT
7
Gene hunting
Ricerca del gene responsabile di un particolare evento (in genere
malattia)
Esempio
Malattia: fibrosi cistica (frequenza 1/2500)
Causa: gene alterato presente con frequenza 1/25 (se ereditato
da ambedue i genitori causa la malattia)
Scoperte:
primi anni ‘80: inizia la ricerca del gene responsabile della FC
(per diagnosi prenatale e cura)
1985: viene individuato il cromosoma 7 su cui risiede il gene
1989: il gene viene localizzato sul cromosoma 7 (la proteina
corrispondente comprende 1480 aminoacidi)
8
Mappaggio genetico
Posizionamento approssimato di un gene su un particolare
cromosoma (prima fase del gene hunting)
Idea generale:
analizzare la frequenza di diverse combinazioni di fenotipi nella
discendenza per determinare l’ordine dei geni
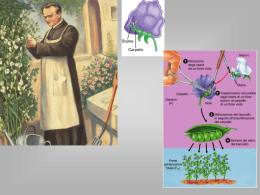
Prima mappa genetica:
sei geni della Drosophila Melanogaster (Sturtevant, 1913)
9
Mappaggio genetico: un esempio
Organismo modello semplice (unico cromosoma)
Numero di geni: 3 (colore di occhi, pelle, capelli)
Ogni gene può essere nello stato
NB: per la stessa posizione di
R: fenotipo rosso
V: fenotipo verde
ricombinazione, l’insieme degli stati
poteva anche essere (p1, m2, m3)
Dati un individuo madre (m1, m2, m3) e un individuo padre (p1, p2, p3),
con mi e pi stati dei geni, un figlio è un individuo con insieme degli
stati fornito da una particolare posizione di ricombinazione i
compresa tra 0 e 3 (ad esempio (m1, p2, p3) per i=1)
Ogni coppia di individui può dare luogo a 8 ricombinazioni diverse
La probabilità di ricombinazione alla posizione i è pari a 1/4
10
Mappaggio genetico: un esempio
Gen1
Gen2
abc
def
abc
aef
abf
abc
def
dbc
dec
def
Dati i fenotipi di un grande numero di figli di un
genitore tutto rosso e uno tutto verde, si vuol
trovare l’ordine dei geni
11
Mappaggio genetico: un esempio
Le diverse possibilità di ricombinazione tra un
individuo (R, R, R) e uno (V, V, V) sono:
per i=0: (V, V, V) o (R, R, R)
per i=1: (R, V, V) o (V, R, R)
per i=2: (R, R, V) o (V, V, R)
NB:per i=3: (R, R, R) o (V, V, Mappe genetiche)
- Probabilità di avere caratteri diversi per i geni in posizione 1 e 2: 1/4
- Probabilità di avere caratteri diversi per i geni in posizione 2 e 3: 1/4
- Probabilità di avere caratteri diversi per i geni in posizione 1 e 3: 1/2
12
Mappaggio genetico: un esempio
Generalizzando si ottiene
Numero di geni: n
Ogni gene può essere nello stato
R: fenotipo rosso
V: fenotipo verde
Dati un individuo madre (m1, m2, …, mn) e un individuo padre (p1, p2, …, pn),
con mi e pi stati dei geni, un figlio è un individuo con insieme degli stati
fornito da una particolare posizione di ricombinazione i compresa tra 0 e
n ((m1, …, mi, pi+1, …, pn) o (p1, …, pi, mi+1, …, mn))
Ogni coppia di individui può dare luogo a 2(n+1) ricombinazioni diverse
La probabilità di ricombinazione alla posizione i (probabilità di avere
diversi i caratteri per i geni nelle posizioni i e i+1) è pari a 1/(n+1)
La probabilità di avere diversi i caratteri per i geni non consecutivi è pari
a d/(n+1) con d distanza tra i caratteri
13
Mappaggio genetico: un esempio
INPUT:
un elevato numero di figli di un individuo tutto rosso (R, R, …, R)
e di uno tutto verde (V, V, …, V)
OUTPUT:
ordine (g1, g2, …, gn) dei geni nell’organismo modello
Misurando la frequenza dei caratteri diversi nella popolazione dei figli, si risale
alla stima delle distanze tra i geni gi e quindi al loro ordine sul cromosoma
14
Mappaggio fisico del DNA
Mappa fisica := localizzazione di marcatori lungo
la sequenza del DNA
Tecnica: RFLP (Restriction Fragments Length
Polymorphism)
Esempio: Siti di restrizione
1970: Hamilton Smith scopre che HindII taglia il DNA in
corrispondenza di GTGCAC o GTTAAC
Il DNA umano è tagliato in circa un milione di
frammenti
Mutazioni interne al sito di restrizione impediscono il
taglio
1973: Danna et al. costruiscono la prima mappa di
restrizione per il DNA del Simian Virus 40
15
Mappaggio fisico del DNA
Il mappaggio fisico del DNA consiste nel
creare alcune copie del DNA da mappare
frammentare con enzimi di restrizione
confrontare i frammenti e le loro sovrapposizioni
Generazione di fingerprints per
analisi dei siti di restrizione
Misura della lunghezza dei frammenti
ibridazione
Ricerca di piccole sequenze che legano i frammenti
16
Analisi dei siti di restrizione
Enzima A
Enzima B
Enzima A+B
3
8
5
4
3
6
1
5
10
11
2
6
7
3
7
17
Problema della doppia digestione (DDP)
INPUT:
tre multinsiemi di numeri interi:
A = {a1, a2, …, an}
B = {b1, b2, …, bm}
O = {o1, o2, …, ok}Il problema DDP è NP-completo
(Goldstein e Waterman, 87)
OUTPUT:
due permutazioni di A e B, pA e pB, tali che, riportando
su una retta gli elementi di A in segmenti consecutivi e
ordinati secondo pA e gli elementi di B in segmenti
consecutivi e ordinati secondo pB, si ottenga una
suddivisione in segmenti corrispondenti agli elementi di
O
18
Problema della doppia digestione (DDP)
Esempio
INPUT:
A = {3, 6, 8, 10}
B = {4, 5, 7, 11}
O = {1, 2, 3, 3, 5, 6, 7}
OUTPUT:
pA
pB
pA U pB
3
4
8
3
1
11
5
4
3
6
11
10
17
7
9
5
20
2
6
3
27
27
7
19
Scaricare