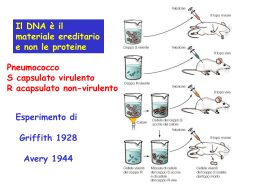
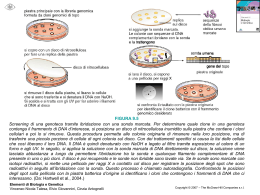
Manipolazione del DNA in genetica umana e medica Corso di Genetica Medica Corsi di Laurea in Fisioterapia, Logopedia, Ortott. Ass. Oft, T.N.P.E. Facoltà di Medicina e Chirurgia Alberto Piazza Biotecnologie per la diagnosi genetica molecolare Analisi a bassa risoluzione – Analisi cromosomica (del cariotipo) – RFLP (restriction fragment length polymorphisms) – Southern blotting - DNA – Northern blotting - RNA – Western blotting - proteine – PCR (polymerase chain reaction) – amplificazione del DNA Analisi ad alta risoluzione – RFLP – DNA Sequencing - determinazione dei nucleotidi – PCR – amplificazione del DNA Sonde (probes) molecolari Reagenti usati per identificare una sequenza specifica di DNA o RNA in un miscuglio complesso in cui è avvenuta una ibridazione (accoppiamento di basi complementari tra il filamento singolo della sonda ed il filamento singolo bersaglio di DNA o RNA). Le sonde sono comunemente marcate da materiale radioattivo o fluorescente. Sonde cDNA- sequenze di DNA complementare a RNA (originariamente prodotte mediante trascrizione inversa di RNA), in grado di identificare sequenze di esoni (espressi) Sonde oligonucleotidiche- sequenze di DNA prodotte sinteticamente lunghe 14- 25 nucletidi Sonde RNA- RNA marcato in grado di identificare sequenze di di DNA o RNA complementari Analisi cariotipica spettrale Ibridazione In Situ Fluorescente (FISH) 24 sonde DNA cromosomaspecifiche, ciascuna marcata con una combinazione differente di 5 fluorocromi IBRIDAZIONE IN SITU CON: A) SONDE CHE VISUALIZZANO SINGOLI CROMOSOMI B) SONDE CHE VISUALIZZANO TUTTI I CROMOSOMI A C) SONDE CHE VISUALIZZANO SPECIFICHE BANDE “CHROMOSOME BARR CODE” C B “Fishing for genes” Endonucleasi di restrizione Enzimi che tagliano il DNA a doppia elica in siti specifici Blunt Ends = estremità prive di estensione Endonucleasi di restrizione EcoRI non taglierà questa sequenza Southern Blotting (DNA) Diagnosi della sindrome da androgeno resistenza per mezzo dell’analisi del Southern blot Il DNA dal gel di agarosio è stato trasferito su nitrocellulosa ed ibridizzato alla sonda cDNA di un recettore androgenetico. Si osservano 3 frammenti genomici nel DNA di entrambi i genitori ma non in quello del figlio. Conclusione: Il gene per il recettore androgenetico (AR) è X-linked. La madre ha solo un gene AR intatto (l’intensità delle bande è simile a quella della padre). Il figlio ha ereditato dalla madre il cromosoma X che ha una delezione nel gene AR. Mutazioni puntiformi Due mutazioni puntiformi che cambiano la sequenza nucleotidica EcoRI non è in grado di tagliare le due sequenze mutate Polimorfismi del DNA polimorfismi del DNA = differenze nella sequenza di DNA –per definizione due o più alleli ad un locus la cui frequenza nella popolazione è >1% Il termine polimorfismo viene esteso ai casi di cambiamento nella sequenza del DNA: negli RFLP per indicare differenze nelle lunghezze dei frammenti di restrizione causate da perdita o guadagno di siti di restrizione nel DNA, nelle delezioni o inserzioni di DNA, nelle ripetizioni di gruppi nucleotidici nei microsatelliti e minisatelliti, nelle ripetizioni di trinucleotidi, nelle mutazioni puntiformi (SNP), etc. Polimorfismi RFLP e Southern Blot Si possono identificare solamente i frammenti di DNA che ibridizzano con la sonda molecolare (probe) Diagnosi molecolare dell’anemia falciforme la sonda non ibridizza con il frammento MstII di 0.2 Kb Analisi di un albero famigliare mediante RFLP e Southern Blot in questa famiglia l’allele 2 è concatenato alla malattia Polimorfismi VNTR e STR I minisatelliti (VNTR, Variable Number of Tandem Repeats) sono lunghi più di 12 basi I microsatelliti (STR, Short Tandem Repeats) sono lunghi da 2 a 4 basi Gli alleli A and B al locus X differiscono per il numero di ripetizioni di minisequenze (tandem repeats) La sonda (probe) 1 – una sonda single-locus - identifica un tratto di DNA esclusivo di un locus La sonda (probe) 2 –una sonda multiple-locus – identifica tratti di DNA che si ripetono in più loci Una famiglia tipizzata per un polimorfismo VNTR “DNA Fingerprinting” per il test di paternità Chi tra F1 e F2 è il padre biologico di C? Sonde a molti loci con molti alleli Dal 5 al 10% dei figli hanno un padre biologico diverso dal padre presunto Il “Northern blot” è una tecnica usata per determinare in quale tessuto o tipo cellulare è espresso un gene, ed anche la lunghezza e la quantità di mRNA. I livelli di mRNA sono spesso correlati ai livelli di proteina presente nel tessuto. AMPLIFICAZIONE IN VITRO DEL DNA “REAZIONE A CATENA DELLA POLIMERASI (PCR)” PCR: Reazione a catena della polimerasi Metodo di amplificazione del DNA usando una polimerasi termostabile quale la Taq DNA polimerasi, uno stampo di DNA, un eccesso di primers e dideossinucleotidi (dNTP) in un buffer. PCR: Reazione a catena della polimerasi PCR: Reazione a catena della polimerasi PCR: amplificazione n.ro cicli n.ro sequenze bersaglio 1 0 3 2 10 256 15 8192 20 262.144 25 8.388.608 30 268.435.456 La sequenza bersaglio è la sequenza di DNA sintetizzata tra i due primers Occorre ~1g di DNA per l’analisi di una sequenza o una digestione con enzimi di restrizione tali da poter essere visibili in elettroforesi. Se vi sono ~5 pg di DNA/cellula umana (5x10-12g) allora ~1 g of DNA potrebbe essere isolato da 200.000 cellule ma avremmo un miscuglio di tutti i geni. In 1 g di DNA genomico, una copia singola di un gene (300 bp) equivarrebbe a ~0.1 pg di DNA. Questo 0.1 pg di DNA potrebbe essere amplificato mediante PCR producendo 0.8 g in 25 cicli e 27 g in 30 cicli. PCR/RFLP per l’analisi dell’anemia falciforme PCR/VNTR/STR in genetica forense Quale delle persone sospette può avere commesso il crimine? La tecnica PCR è utile, spesso necessaria, per amplificare il DNA estratto dai reperti trovati sulla scena del delitto. Si usano sonde multi-locus con molti alleli. IL MODELLO DI WATSON E CRICK LA DOPPIA ELICA VISUALIZZATA CON IL MICROSCOPIO A SCANSIONE A FORZA ATOMICA IMMAGINE AL MICROSCOPIO A FORZA ATOMICA DEL GENE CHE CODIFICA PER LA CONNESSINA UMANA SEQUENZA DEL GENE DELLA BETA GLOBINA UMANA Sequenziamento del DNA (1): strutture dei nucleotidi Sequenziamento del DNA (2): il metodo di Sanger Il contenuto di ciascuna provetta viene caricato in 4 pozzetti separati di un gel di poliacrilammide * Indica il primer (lungo 15 nucleotidi) ** I dNTP sono ad una concentrazione 100 volte superiore di quella dei ddNTP in ciascuna provetta Sequenziamento del DNA (3): il metodo di Sanger pozzetti autoradiogramma Si legga la sequenza man mano sintetizzata dal basso verso l’ alto. Il nucleotide G è il più vicino al primer (* indica il primer (lungo 15 nucleotidi)) e l’estremità 5’ mentre il nucleotide T è all’estremità 3’. 5’GCTCCACGTAACGT3’ Questa sequenza è complementare al filamento stampo. ORGANISMI DI CUI IL GENOMA È COMPLETAMENTE SEQUENZIATO (AGOSTO 2003) Virus Batteri Eucarioti >1000 >100 19 EUCARIOTI Vertebrati Invertebrati Piante Funghi Protozoi 4 3 8 3 1 Il Progetto Genoma Umano Obiettivi 1. Costruire una mappa di marcatori genetici (polimorfismi) polimorfismo – due o più alleli che si manifestano in una popolazione con una frequenza maggiore dell’1% - fino ad oggi sono stati identificati circa 2.165.000 SNP (Single Nucleotide Polymorphisms) e 133.000 STS (Sequence Tagged Sites, coppie di primers PCR), polimorfismi distribuiti nel genoma umano - tali marcatori dovrebbero idealmente essere sufficientemente vicini così che di ogni malattia ereditaria possa essere identificato il gene o i geni responsabili 2. Stabilire una mappa “fisica” della localizzazione dei geni in relazione ad altri geni sui cromosomi 3. Sequenziare l’intero genoma umano aploide di 3 x 109 bp Esempio di mappa fisica e genetica nel Genoma Umano Lungo tutto il genoma sono stati identificati (2002) circa 2.165.000 SNP e 133.000 STS Single Nucleotide Polymorphisms (SNP) Sequence Tagged Sites (STS) (coppie di primers PCR) Restriction Enzyme Sites (NotI) 1 centiMorgan (cM) = 2*106bp STS Notl STS cM Il gene umano medio Uno degli scopi del progetto Genoma Umano è di sequenziare le 3 x109 basi nucleotidiche del genoma il cui numero di geni è stimato intorno a 40.000. Il gene umano medio Lunghezza 17.000 coppie di basi (17 kb) Numero di esoni 7 Lunghezza dell’esone 100-200 nucleotidi Lunghezza dell’introne 200-2.000 nucleotidi
Scaricare