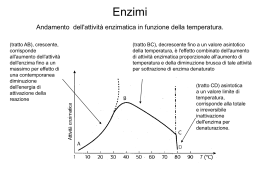
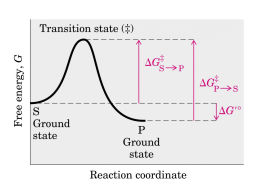
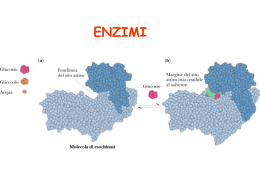
STRUTTURA 3D ENZIMI RNA polimerasi Piruvato carbossilasi Fosfofruttochinasi Glutammina sintetasi DNA polimerasi (E. coli) ATP sintetasi CLASSIFICAZIONE ENZIMI Alcuni tra i principali meccanismi di catalisi enzimatica: •acido base •covalente •elettrostatica e da ione metallico Effetto del mezzo sulla velocità delle reazioni catalizzate da enzimi: temperatura, forza ionica e pH Le proteine, quindi anche gli enzimi, hanno conformazioni 3D complesse strettamente legate alla loro funzionalità: le strutture secondaria e terziaria sono dovute ad interazioni deboli tra atomi coinvolti nel legame peptidico e tra i gruppi –R degli AA costituenti ( idrofobiche, elettrostatiche, legami idrogeno, forze van Der Waals), in relazione al mezzo acquoso e all’ambiente. Vengono studiate le interazioni tra enzimi, proteine, e il mezzo. Queste interazioni stabilizzano o meno la struttura dell’enzima in studio, influenzando la catalisi. Temperatura Forza ionica pH Saggio enzimatico in vitro: concentrazioni di sali e pH del tampone di dosaggio; temperatura di dosaggio – parametri che devono essere standardizzati. TEMPERATURA E VELOCITA’ ENZIMATICA In tutte le reazioni chimiche, cinetica di reazione e temperatura sono strettamente collegate: aumento di T aumento di velocità di reazione. In termini probabilistici, incrementi di T aumentano i moti delle particelle, le collisioni e urti “utili” per abbassare l’E att. e favorire lo stato di transizione della reazione chimica: questo è valido anche per le reazioni catalizzate dagli enzimi. Secondo la reazione di Arrhenius: E*: 1,7-70 Kj M-1 K2 = A2e-Ea2/RT E sat. K = Ae-E*/RT Q10 ≈ 2 In concomitanza: T oltre una certa soglia denaturano la proteina dotata di attività enzimatica – diminuzione delle U enz. A temp. > 40-50°C - drastica riduzione. 50 °C Esistono enzimi più o meno stabili alle alte temperature: isoenzimi termostabili e termolabili; enzimi che sono stabili a 80-100°C (Taq polimerasi); a T <10°C, attività molto rallentata dipende dalla struttura specifica delle proteine enzimatiche SALI E VELOCITA’ ENZIMATICA In generale: effetto simile a quello dato dalla concentrazione di sali sulle macromolecole biologiche e, in particolare, le proteine: la solubilità di una proteina a bassa forza ionica aumenta con l’aumentare della forza ionica (salting in) . Ad alta forza ionica, la proteina precipita, si separa dal mezzo acquoso (salting out). Forza ionica, I = ½ cizi2 -Gli ioni dei sali sciolti in una soluzione acquosa interferiscono con i legami a idrogeno tra H2O (mezzo acquoso) e proteine : modificano le interazioni intramolecolari che stabilizzano le proteine, quindi la loro conformazione e/o solubilità: -Ioni “cosmotropici” o stabilizzanti (Cl-, Br-): aumentano e stabilizzano le interazioni H2O-enzima; aumentano la solubilità nel mezzo acquoso. Ad alte concentrazioni ammonio solfato, salting-out, proteine non denaturate. Effetti relativi che dipendono dal tipo di sale (specie ionica) , dal tipo di proteina- principio base della precipitazione frazionata -Ioni “chaotropici” o denaturanti (ClO4-; SCN-): alterano le interazioni H2O-enzima (proteina) sottraggono H2O all’enzima e quindi ne diminuiscono la solubilità ma ne alterano anche la conformazione 3D (denaturanti). pH E VELOCITA’ ENZIMATICA La [H+] può influire significativamente sulla catalisi enzimatica. Un enzima proteico è suscettibile sia ai cambiamenti conformazionali legati al grado di ionizzazione e carica, a diversi pH, dei gruppi –R ionizzabili degli AA che lo compongono sia al grado di protonazione di gruppi –R ionizzabili presenti sul sito attivo e determinanti per la catalisi enzimatica. A pH estremi le proteine perdono la conformazione 3D , precipitano e quindi molti enzimi si denaturano. Esempio: Se un enzima esplica la sua azione catalitica grazie alla presenza di un’ istidina e di una Lisina nel sito attivo svolgendo un attacco nucleofilo sul substrato in trasformazione oppure se un altro enzima trasforma S in P mediante un’interazione di tipo elettrostatico attraverso un aspartato o una lisina, opportunamente dislocati sul sito attivo, comprendiamo subito che il grado di protonazione dei gruppi in gioco alterano la catalisi: esisteranno solo stretti intervalli di pH per permettere l’interazione ES ottimale. Forma attiva dell’enzima a intervalli di pH che vengono definiti sperimentalmente. Esistono intervalli di pH ottimale. Il pH influenza i parametri cinetici Vmax e Km dell’enzima e può variare anche lo stato di ionizzazione del substrato S e di eventuali coenzimi. Intorno di pH di massima attività enzimatica Da: www.vi.cl Effetto del pH del mezzo sulla velocità di alcuni enzimi pH e velocità enzimatica colinesterasi Velocità iniziale Velocità iniziale chimotripsina pepsina papaina Struttura, meccanismo di catalisi e dipendenza dal pH dell’Adenosina Deaminasi (ADA) La reazione catalizzata dalle adenosina aminoidrolasi è stata trovata sia in organismi procariotici che eucariotici. Nei mammiferi esistono due isoforme principali di ADA, ADA1 e ADA2. ADA1 è legata alla regolazione della risposta immunitaria e alla patogenesi della SCID. Altre patologie sono state associate sia al deficit che alla sovra-espressione di ADA. Struttura: Metalloproteina, con un atomo di Zn come cofattore, legato al sito catalitico. Struttura conservata nell’evoluzione. L’ADA umana è una proteina globulare,con peso molecolare in media circa 40,000 Da, con struttura a/b, detta a barile, tipica anche di altre idrolasi, il sito attivo è una tasca centrale, in corrispondenza del tratto C-terminale dei foglietti b. Catalisi: pH ottimale ADA Nel sito catalitico dell’ADA sono stati individuati alcuni gruppi -R di AA critici e probabilmente coinvolti nella catalisi enzimatica: Asp295, Asp296, Glu217, Hys238, Gly184 (Wilson et al. 1991). Effetto del pH: Esistono varie isoforme e proteine in diversi organismi; l’ADA ha un pH ottimale tra circa 6.8-7.8. Questo è dovuto alla ionizzazione di gruppi -R di AA favorente la catalisi. Agli estremi di pH si ha denaturazione e perdita di attività completa per alterazione dei legami deboli e attrazioni ioniche che stabilizzano la struttura 3D dell’enzima. Riepilogo Esercitazioni Enzimi Saggio enzimatico in condizioni saturanti della LDH in surnatante di omogenato di tessuto muscolare e in siero (confronto): Valutazione della concentrazione di LDH serica e muscolare, UE/ml nei due campioni a confronto. (UE/100 ml) – UI: UE/lt. Substrato: sodio piruvato Coenzima : NADH Tampone fosfato pH 7, temperatura e forza ionica costanti; Enzima a concentrazione costante, campione diluito opportunamente. Lettura 340nm (NADH) per 2 min, calcolo ΔA/min e UE per volume. Att. Specifica: UE/mg proteine totali Determinazione dei parametri cinetici Km e Vmax dell’enzima ADA: Calcolo della V0 a varie concentrazioni di S (adenosina) sia al di sotto che al di sopra della Km attesa. S (adenosina) a concentrazioni crescenti: 5, 10, 20, 40, 60, 80 mM. Tampone fosfato pH 7, temperatura e forza ionica costanti; Enzima alla stessa concentrazione in tutto il dosaggio: costante. Lettura 265nm (adenosina, no inosina). Dipendenza dal pH della V0 dell’ADA: Calcolo della V0 a pH variabile nel mezzo. Substrato a concentrazione costante S ≈ Km, 40 mM; V0= Vmax/2 Tampone fosfato a pH variabile: pH 5, 6, 7, 8, 9, 10. Temperatura e forza ionica costanti; Enzima a concentrazione costante. Lettura 265 nm (adenosina, no inosina).
Scarica