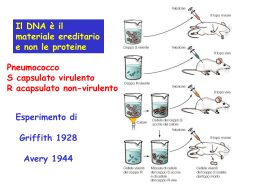
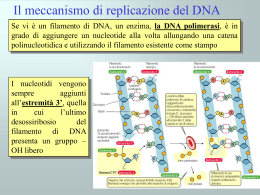
Il materiale ereditario Nel 1930-40 le proteine erano reputate le candidate più probabili a rappresentare il materiale genetico (sono fatte da 20 AA, mentre gli acidi nucleici sono fatti da 4-5 nucleotidi) 1928: Griffith studia i ceppi R e S di Pneumococco ed evidenzia un “Principio Trasformante” in grado di trasformare i batteri uccisi al calore conferendo loro le caratteristiche dei batteri vivi 1944: Avery, Mac Leod e Mc Carty identificano nel DNA il “Principio Trasformante”. Essi lisarono il ceppo S e frazionarono il contenuto cellulare: lipidi, polisaccaridi e proteine non erano in grado di trasformare le cellule batteriche; la trasformazione avveniva solo grazie alla frazione contenente gli acidi nucleici Il DNA è coinvolto nella replicazione virale 1952: Hershey e Chase studiano la replicazione dei batteriofagi, virus che infettano i batteri facendo penetrare solo una parte della loro struttura Marcando alcuni fagi con 35S (Pt) ed altri con 32P (Ac. Nucleici) dimostrarono che il materiale responsabile della replicazione virale è il DNA Gli Acidi Nucleici, DNA ed RNA, sono polimeri dei nucleotidi Struttura del DNA(I) I gruppi fosfato formano legami 3’-5’ fosfodiesterici La catena polinucleotidica ha una polarità con estermità 3’ ed estermità 5’ 1949: Regole di Cargaff: Nel DNA estratto da diversi tessuti N° purine=N° pirimidine Adenine=Timine Citosine=Guanine 1951-1953: Franklin e Wilkins: diffrazione ai raggi X del DNA Struttura del DNA(II) Franklin e Wilkins: dalla analisi cristallografica si evince che il DNA ha struttura elicoidale e che si ripetono le misure di 0.34 nm, 3.4 nm, 2nm Watson e Crick (1953) propongono un modello strutturale del DNA: due catene polinucleotidiche sono avvolte fra di loro a costituire una doppia elica. L’ impalcatura esterna è costituita da zucchero-fosfato, mentre le basi azotate si appaiano all’ interno. Ogni coppia di basi dista dall’ altra 0.34 nm; ciascun passo dell’ elica comprende 10 b.p. (=3.4 nm); la larghezza è 2nm Le due catene hanno disposizione antiparallela Appaiamento specifico delle basi La larghezza di 2 nm si concilia solo con l’ appaiamento fra una purina ed una pirimidina I legami ad H favorevoli si hanno solo fra CG e AT Questo appaiamento specifico delle basi si accorda con i dati di Cargaff La sequenza delle basi nelle due semieliche è complementare Le possibili sequenze nucleotidiche nelle cellule sono infinite e possono immagazzinare un enorme numero di informazioni Replicazione del DNA (I) Nel modello di Watson e Crick “la specificità dell’ appaiamento fra le basi, suggerisce immediatamente un possibile meccanismo di copiatura del materiale genetico” Ciascun filamento di DNA può fungere da stampo per la copia del filamento opposto in una replicazione semiconservativa La prova sperimentale della replicazione semiconservativa, si deve a Meselson e Stahl (1958) La replicazione semiconservativa spiega la trasmissione delle mutazioni Le mutazioni possono insorgere come cambiamento nella sequenza di basi del DNA La replicazione semiconservativa permette alle mutazioni di essere tramandate alle cellule figlie In genere intervengono efficienti sistemi di riparo, ma in media un errore su un miliardo non viene corretto durante la replicazione Replicazione del DNA (II) E’ un processo complesso che richiede l’ intervento di moltissimi enzimi e di energia Innanzitutto le due semieliche del DNA devono essere srotolate ad opera delle DNA elicasi Poi la singola elica deve essere stabilizzata finché non viene copiata, ad opera delle proteine che legano il singolo filamento (SSBP) Nella regione adiacente a quella srotolata, si crea un superavvolgimento, per cui intervengono le Topoisomerasi (I e II) che operano i tagli e poi risaldano i filamenti Replicazione del DNA (III) Le DNA polimerasi aggiungono nucleotidi al 3’ partendo da nucleosidi trifosfati ed usando l’ energia del PP Il nuovo filamento cresce sempre dal 5’ al 3’ Replicazione del DNA (IV) Le DNA polimerasi possono aggiungere nucleotidi al 3’ di una catena polinucleotidica preesistente Nel punto di inizio della replicazione è necessario un innesco, costituito da un RNA primer (5-14 nucleotidi) che viene sintetizzato dalla RNA primasi (che copia RNA su DNA). Poi continua la DNA polimerasi Infine il primer viene degradato da enzimi specifici e sostituito da DNA Replicazione del DNA (V) I filamenti del DNA sono antiparalleli La sintesi può avvenire solo dal 5’ al 3’ Su uno stampo 3’→5’ La replicazione inizia in un punto preciso, l’ origine di replicazione ed entrambi i filamenti vengono copiati nella forca di replicazione (ad Y) La posizione della forca di replicazione cambia continuamente L’ estremità 3’ di uno dei filamenti si allunga in maniera continua verso la forca di replicazione (filamento guida) L’estremità 3’ dell’ altro filamento si allunga in maniera discontinua nella direzione opposta all’ avanzamento della forca (filamento in ritardo) Questi frammenti di Okazaki (100-2000 nucleotidi) vengono riuniti dalla DNA ligasi Le telomerasi I cromosomi eucariotici hanno estremità libere Le DNA polimerasi lavorano in maniera discontinua sul filamento in ritardo e, all’ estremità del cromosoma, lasciano una piccola porzione non replicata Questo DNA telomerico che si perde ad ogni ciclo cellulare, non contiene sequenze codificanti ma sequenze ripetute Il DNA telomerico può essere allungato dalla Telomerasi, particolare DNA polimerasi attiva nelle cellule in intensa replicazione (cellule germinali e neoplastiche) La perdita di DNA telomerico sembra correlata al processo della senescenza e la possibilità di interferire farmacologicamente con le telomerasi apre prospettive nella terapia di malattie degenerative e neoplastiche
Scaricare