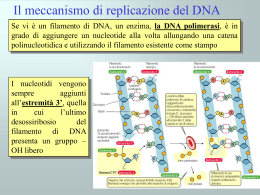
LA BIOLOGIA MOLECOLARE DEL GENE • Un enzima è una molecola proteica che si comporta come catalizzatore biologico • Essi hanno la funzione di velocizzare la maggior parte delle reazioni chimiche che avvengono in un organismo • Si ritrovano immutati al termine della reazione •Perchè una reazione chimica inizi, i reagenti devono assorbire una quantità di energia chiamata energia di attivazione (EA). Enzima Barriera EA Reagenti Figura 5.5A Contenitore 1 Prodotti Contenitore 2 – Un enzima si LEGA AI REAGENTI per abbassare l’energia di attivazione necessaria per avviare una reazione chimica. EA senza enzima EA con enzima Energia Reagenti Differenza netta di energia Prodotti Figura 5.5B Direzione della reazione • Ogni cellula contiene molti enzimi • Ogni reazione cellulare è catalizzata da un enzima specifico • Gli enzimi hanno strutture tridimensionali caratteristiche che determinano le reazioni chimiche che essi sono in grado di catalizzare in una cellula L’azione catalitica di un enzima consiste in un ciclo a quattro fasi che si ripete migliaia di volte in tempi brevissimi 1. Il substato- sito attivo del reagente si combina specificatamentecon la molecola dell’enzima _MECCANISMO CHIAVE-SERRATURA_ 2. la combinazione con l’enzima attiva il substrato rendendo più reattivi alcuni legami più reattivi 3. Il substrato viene trasformato in prodotto 4. I prodotti si dissociano dall’enzima che può nuovamente andare incontro alla fase1 •Esempio di reazione catalizzata da un enzima: 1 Enzima disponibile con il sito attivo vuoto Sito attivo Glucosio Substrato (saccarosio) Il substrato 2 si lega all’enzima che subisce un adattamento indotto Enzima (saccarasi) Fruttosio H2O 4 I prodotti vengono liberati 3 Il substrato si scinde nei prodotti L’AMBIENTE CELLULARE INFLUENZA L’ATTIVITÀ DEGLI ENZIMI – La temperatura, la concentrazione dei sali e il pH influenzano l’attività enzimatica. – Per funzionare, alcuni enzimi richiedono molecole non proteiche chiamate cofattori. – I cofattori possono essere sostanze inorganiche, come gli ioni metallo, o molecole organiche (in questo caso si chiamano coenzimi). – Una sostanza chimica che interferisce con l’attività di un enzima è detta inibitore. – L’azione di un inibitore è irreversibile se si formano legami covalenti tra inibitore ed enzima. È reversibile quando si formano solo legami deboli (come il legame idrogeno). – Gli inibitori competitivi occupano il sito attivo di un substrato. – Gli inibitori non competitivi cambiano la funzione dell’enzima modificando la sua forma. Substrato Sito attivo Enzima Legame normale del substrato Inibitore competitivo Inibitore non competitivo Inibitore enzimatico GENETICA FENOTIPO: l’aspetto fisico e fisiologico di una cellula GENOTIPO : insieme di tutti i geni di una cellula IL FENOTIPO è il risultato dell’espressione del GENOTIPO CONCLUSIONI GENETICA CLASSICA I CROMOSOMI SONO PORTATORI DELL’ EREDITARIETA’ I GENI SI TROVANO SUI CROMOSOMI STRUTTURE PRESENTI NEL NUCLEO DELLE CELLULE EUCARIOTE GENETICA MOLECOLARE INDAGA • COMPOSIZIONE CHIMICA • STRUTTURA • PROPRIETA’ • FUNZIONAMENTO DEI GENI • Ogni cromosoma è composto da una lunga molecola di DNA associata a proteine • Alcuni esperimenti hanno dimostrato che il materiale genetico è formato da DNA • le proteine dei cromosomi hanno funzione strutturale •Il DNA è un acido nucleico costituito da lunghe catene di nucleotidi. Scheletro zucchero-fosfato A C Gruppo fosfato Base azotata Zucchero Nucleotide del DNA A C Base azotata (A, G, C, o T) Gruppo fosfato O H3C C O T T O P O G T CH HC H C CH H Zucchero (deossiribosio) Nucleotide del DNA Polinucleotide del DNA H Timina (T) O O T N CH2 H C N C O O– G C •Il DNA ha quattro tipi di basi azotate: •adenina (A), timina (T), citosina (C) e guanina (G) H O H3C H C C C H H N C H N C N C C C N H O N H H Timina (T) Citosina (C) Pirimidine H N H O N H O C C N C C N H C N N H N C C N H Adenina (A) Guanina (G) Purine H C N H C C N H H •Anche l’RNA è un acido nucleico ma è composto da uno zucchero leggermente differente (il ribosio) e una base azotata chiamata uracile (U) al posto della timina. Base azotata (A, G, C, o U) O Gruppo fosfato H C N C H O Ossigeno C O P Legenda Idrogeno Carbonio Azoto O H CH2 C N O Uracile (U) O– O C H H C H C C H O OH Zucchero (ribosio) Fosforo Nel 1953 James Watson e Francis Crick determinarono la struttura tridimensionale del DNA, basandosi anche sul lavoro di Rosalind Franklin. – La struttura del DNA consiste di due filamenti di polinucleotidi attorcigliati l’uno sull’altro in una doppia elica. – Si può immaginare questa struttura come una scala di corda dotata di rigidi pioli in legno e arrotolata in spire. Torsione – I legami idrogeno tra le basi tengono uniti i filamenti. – Ogni base è appaiata con una base complementare: A con T, e G con C catene complementari G C T A A Coppie di basi appaiate C T C G C G A T T O O P –O O H2C O O P –O O H2C G T O OH P O O H2C –O A O O –O P O H2C A A T A Legame idrogeno OH O O O A T CH2 O O– P O O O CH2 O O– O P O O CH2 O O– P O O O CH2 O O– P HO O C G G C A T T OH G A O C T Modello a nastro Struttura chimica Modello computerizzato CONOSCENDO LA CONFORMAZIONE SPAZIALE DELLA MOLECOLA SI INIZIA AD INTUIRE COME POSSA: IMMAGAZZINARE COPIARE TRASMETTERE L’INFORMAZIONE GENETICA DUPLICAZIONE UTILIZZARE QUESTE INFORMAZIONI PER DIRIGERE LA SINTESI PROTEICA SINTESI PROTEICA – Esempio di divisione Cromosoma cellulare procariotico Membrana plasmatica Parete 1 Il cromosoma si duplica e le due copie si separano 2 La cellula continua ad allungarsi; i cromosomi si spostano 3 La cellula si divide in due cellule figlie – Cellula batterica MODELLO SEMICONSERVATIVO • I due filamenti di DNA di partenza si separano e ciascuno di essi funziona da stampo per formare il filamento complementare. I nucleotidi si allineano uno alla volta lungo il filamento stampo secondo la regola dell’appaiamento delle basi azotate La duplicazione del DNA inizia presso specifici punti di origine della duplicazione sulla doppia elica. Punto di origine della duplicazione Filamento originario Filamento di nuova sintesi Bolla di duplicazione Due molecole figlie di DNA •Ogni filamento di una doppia elica ha un orientamento opposto all’altro. Estremità 5 P Estremità 3 HO 5 4 3 2 2 1 A T 1 5 P P C G P P G C P P T OH Estremità 3 3 4 A P Estremità 5 – La cellula sintetizza un filamento nuovo in maniera continua usando l’enzima DNA-polimerasi. – L’altro filamento è sintetizzato in brevi segmenti consecutivi che sono poi uniti in un unico filamento dall’enzima DNA-ligasi. Molecola di DNA-polimerasi 5 3 DNA originario 3 5 Filamento sintetizzato senza interruzioni 3 5 5 3 DNA-ligasi Direzione complessiva della duplicazione Filamento sintetizzato in segmenti consecutivi PCR • La reazione a catena della polimerasi (in inglese: Polymerase Chain Reaction), comunemente nota con l'acronimo PCR, è una tecnica di biologia molecolare che consente la moltiplicazione (amplificazione) di frammenti di acidi nucleici dei quali si conoscano le sequenze nucleotidiche iniziali e terminali. L'amplificazione mediante PCR consente di ottenere in vitro molto rapidamente la quantità di materiale genetico necessaria per le successive applicazioni Quando il campione di DNA è scarso o impuro, la reazione a catena della polimerasi (Polymerase Chain Reaction, o PCR) è un metodo più appropriato per ottenere un grande quantitativo di un particolare gene. Molecola iniziale di DNA 1 2 4 8 Numero di molecole di DNA • La PCR ricostruisce in vitro uno specifico passaggio della riproduzione cellulare: la ricostituzione (sintesi) di un segmento di DNA "completo" (a doppia elica) a partire da un filamento a singola elica. Il filamento mancante viene ricostruito a partire da una serie di nucleotidi (i "mattoni" elementari che costituiscono gli acidi nucleici) che vengono disposti nella corretta sequenza, complementare a quella del DNA interessato. • Questo processo viene svolto in natura da enzimi chiamati DNApolimerasi, che sono in grado di sintetizzare progressivamente un nuovo filamento di DNA nelle seguenti condizioni: • devono essere disponibili i nucleotidi da polimerizzare, sotto forma di desossiribonucleotidi trifosfati (dNTP); • il DNA deve essere denaturato, ovvero le due eliche che compongono i filamenti devono essere già separate; • il segmento da ricostruire può essere soltanto prolungato, ovvero non è possibile sintetizzare un nuovo filamento a partire da zero; • devono inoltre essere rispettate opportune condizioni di temperatura, pH, ecc. • È possibile quindi ricostruire le condizioni che portano alla formazione dei nuovi segmenti di DNA, ponendo in soluzione: • una quantità, anche minima, del segmento di DNA che si desidera riprodurre; • una quantità opportuna di nucleotidi liberi per costituire i nuovi filamenti; • opportuni "inneschi", detti primer, costituiti da brevi sequenze di DNA (oligonucleotidi) complementari agli estremi 5’ e 3’ del segmento da riprodurre; • altri elementi di supporto (ad es. ioni magnesio), necessari per costituire l'ambiente adatto alla reazione; • una DNA polimerasi (non è necessario che provenga dallo stesso organismo di cui si deve replicare il DNA). • Per avviare la reazione della polimerasi (fase di prolungamento del filamento a partire dal primer 5’) è prima necessario provvedere alla separazione dei filamenti del DNA (fase di denaturazione), quindi alla creazione del legame tra i primer e le regioni loro complementari dei filamenti di DNA denaturati (fase di annealing). Questo processo risulta però incompatibile con la DNA polimerasi umana, che viene distrutta alle temperature necessarie alla denaturazione (9699 °C). • Per ovviare a questo inconveniente si fa ricorso alle polimerasi appartenenti a organismi termofili che non sono inattivate dalle alte temperature, ad esempio la Taq polimerasi proveniente dal batterio termofilo Thermus aquaticus. Ciò consente di realizzare più cicli di PCR in sequenza, in ciascuno dei quali viene duplicato anche il DNA sintetizzato nelle fasi precedenti, ottenendo una reazione a catena che consente una moltiplicazione estremamente rapida del materiale genetico di interesse. • Schema di un ciclo di PCR [La soluzione di DNA da replicare, desossiribonucleotidi trifosfati, ioni magnesio, primer e DNA polimerasi viene portata a una temperatura compresa tra 94 e 99 °C. Ci si trova, di conseguenza, in una situazione in cui la doppia elica del DNA viene completamente scissa ed i due filamenti di cui essa è composta sono liberi (fase di denaturazione). • Successivamente la temperatura viene abbassata fino a 40-55 °C circa al fine di permettere il legame dei primer alle regioni loro complementari dei filamenti di DNA denaturati (fase di annealing). • Infine la temperatura viene alzata fino a 65-72 °C al fine di massimizzare l'azione della DNA polimerasi che determina un allungamento dei primer legati, utilizzando come stampo il filamento singolo di DNA (fase di prolungamento). • Il ciclo descritto viene ripetuto generalmente per circa 30-40 volte. In genere non si superano i 50 cicli in quanto ad un certo punto la quota di DNA ottenuto raggiunge un plateau. Ciò avviene, ad esempio, per carenza degli oligonucleotidi usati come inneschi o per diminuzione dei dNTP. Bisogna inoltre considerare che si potrebbe amplificare in maniera eccessiva anche eventuale materiale genomico contaminante.
Scaricare